Punktmutation
Als Punktmutation wird in der Biologie eine Genmutation bezeichnet, wenn durch die Veränderung nur eine einzelne Nukleinbase betroffen ist.
Formen der Punktmutation
Leseraster bleibt erhalten
Durch die degenerierte Eigenschaft (Redundanz) des genetischen Codes kann eine solche Punktmutation folgenlos bleiben. Degeneriertheit bedeutet hier, dass eine Aminosäure durch mehrere Codons codiert werden kann. Den 20 Aminosäuren stehen 61 codierende Basentripletts zur Verfügung (3 Basentriplett-Kombinationsmöglichkeiten sind nicht-codierend). Falls aus dem ursprünglichen Codon durch die Punktmutation ein Codon wird, das die gleiche Aminosäure codiert, entsteht das gleiche Protein wie mit dem ursprünglichen Codon.
Substitution
Wird bei einer Punktmutation eine Base der DNA gegen eine andere ausgetauscht, so nennt man diesen Vorgang Substitution. Geschieht dieser Austausch in einer codierenden Region, wird eine veränderte mRNA transkribiert und bewirkt den Einbau einer abweichenden Aminosäure, was zu einem veränderten Protein führt, welches ggf. seine Aufgaben – je nach Lage der Mutation und substituierter Aminosäure – nicht, oder nicht mehr vollständig ausführen kann. Ein bekanntes Beispiel für eine Punktmutation beim Menschen ist die Sichelzellenanämie. Eine Verbesserung des Polypeptids ist genauso möglich, wenn auch unwahrscheinlicher. Ein Beispiel dafür wäre eine erhöhte Substratumsetzung eines Enzyms.
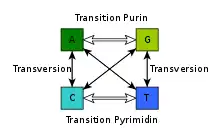
Bei der Punktmutation unterscheidet man zwei Typen der Substitution:
- Transition: Substitution einer Purin- gegen eine andere Purinbase bzw. einer Pyrimidin- gegen eine andere Pyrimidinbase.
- Transversion: Substitution einer Purin- gegen eine Pyrimidinbase oder umgekehrt.
Für codierende Sequenzen kann man die Substitution in folgende Kategorien einteilen:
- Funktional, d. h. sie haben einen Effekt auf die Bildung von Proteinen:
- nonsense-Mutation (sinnentstellende Mutation): codiert für einen Stopp der Translation
- missense-Mutation oder nicht-synonyme Mutation (sinnverändernde Mutation): codiert für eine andere Aminosäure
- readthrough-Mutation: ein Stopcodon wird als Aminosäure codiert
- Nicht-funktional, d. h. durch die Redundanz des genetischen Codes wird weiterhin dieselbe Aminosäure codiert:
- silent-Mutation oder synonyme (stille Mutation): codiert für die gleiche Aminosäure
Es kann sich dabei entweder um eine „missense“-Mutation (sinnverändernde Mutation), also den Austausch einer Aminosäure bei der Translation handeln, oder um eine „nonsense“-Mutation (sinnentstellende Mutation), bei der ein Stopcodon entsteht. Außerdem können „nonsense“-Mutationen in Introns Fehler beim Spleißen bewirken. Dabei führen „Nonsense“-Mutationen nicht zwangsläufig bei einem einzelnen Individuum zu einem auffälligen Phänotyp, können aber über evolutionäre Zeiträume hinweg dennoch nachteilig sein.[1] Als dritte Möglichkeit kann es sich auch um eine stille (stumme) Mutationen oder neutrale Mutationen handeln, bei der eine Base verändert wurde, aber weiterhin die gleiche Aminosäure codiert wird. Das ist durchaus möglich, da es wegen der Degeneration des genetischen Codes für manche Aminosäuren mehrere Codierungsmöglichkeiten gibt. Allerdings haben neuere Forschungen ergeben, dass auch stumme Mutationen Krankheiten auslösen können. Der Grund dafür liegt darin, dass sich die mRNA bei gewissen stummen Mutationen (auf dem Weg vom Zellkern zum Ribosom) anders als gewöhnlich faltet, mit der Folge kaum translatiert zu werden oder gerade zu stark translatiert zu werden.
Verschiebungen des Leserasters
Schlimmer verlaufen kann eine Punktmutation, wenn eine Base komplett wegfällt oder eine neue hinzukommt. Hierbei verliert der mRNA-Strang hinter der Mutation seinen ursprünglichen Sinn, da er nach links oder rechts verschoben wird (Frameshift). Dies hat zur Folge, dass das translatierte Protein später eine völlig andere Struktur aufweist. Seine ursprüngliche Funktion geht dabei meist verloren.
Deletion
Bei einer Deletion handelt es sich um den Verlust einer Base. Die nachfolgenden Basen rücken gegen die Leserichtung auf, was das Leseraster des nachfolgenden Codons in ebendiese Richtung verschiebt.
Insertion
Bei einer Insertion handelt es sich um den Zugewinn einer Base. Die nachfolgenden Basen rücken in Leserichtung auf, was das Leseraster des nachfolgenden Codons in ebendiese Richtung verschiebt.
Beispiel
Die Leserasterverschiebung (Frameshift) lässt sich anhand eines kleinen Beispiels erklären. Wenn man den Satz
ICH MAG NUR EIN EIS
betrachtet, ergibt dieser einen Sinn. Jedes Wort besteht aus drei Buchstaben (Tripletts). Ähnlich liegt die Information auch auf einem Gen vor, hier werden die Basen immer zu so genannten Basentripletts zusammengefasst. Wenn nun ein Buchstabe hinzufügt wird – aber immer noch darauf geachtet wird, dass ein Drei-Buchstaben-Wort-Satz gebildet werden soll – verschwindet diese Information. In diesem Beispiel wird hinter dem Wort „ICH“ der Buchstabe X hinzugefügt. Dies entspricht einer Insertion.
ICH XMA GNU REI NEI S
Wenn nach dem Wort „ICH“ der Buchstabe M gelöscht wird – was einer Deletion gleichkommt – aber die Drei-Buchstaben-Wort-Regel gültig bleibt, ergibt der Satz ebenfalls ab dem Wort ICH keinen Sinn mehr.
ICH AGN URE INE IS
Ähnlich verhält es sich, wenn in einem Gen eine Base hinzugefügt wird. Die genetische Information kann nicht mehr sinnvoll abgelesen werden.
Experimenteller Nutzen
Mit dem Verständnis von Punktmutationen war es möglich, den Ames-Test zu entwickeln, der die Anwesenheit von Mutagenen nachweist.
Siehe auch
- Joseph Merrick (litt durch eine mutmaßliche Punktmutation unter Deformationen)
Weblinks
Einzelnachweise
- Bryndis Yngvadottir, Yali Xue, Steve Searle, Sarah Hunt, Marcos Delgado: A Genome-wide Survey of the Prevalence and Evolutionary Forces Acting on Human Nonsense SNPs. In: The American Journal of Human Genetics. Band 84, Nr. 2, 13. Februar 2009, S. 224–234, doi:10.1016/j.ajhg.2009.01.008 (sciencedirect.com [abgerufen am 26. Oktober 2017]).