Eukaryoten
Eukaryoten oder Eukaryonten (Eukaryota) (von altgriechisch εὖ eu = ‚richtig‘, ‚gut‘ und κάρυον karyon = ‚Nuss‘, ‚Kern‘) sind eine Domäne der Lebewesen, deren Zellen (Eucyten) einen echten Kern und eine reiche Kompartimentierung haben[1] (Tiere, Pflanzen und Pilze). Hierin unterscheiden sie sich von den beiden übrigen Domänen im System der Lebewesen, den prokaryotischen Bakterien und Archaeen (letztere früher auch Urbakterien genannt), beide mit procytischen Zellen.
| Eukaryoten | ||||
|---|---|---|---|---|

Verschiedene Eukaryoten | ||||
| Systematik | ||||
| ||||
| Wissenschaftlicher Name | ||||
| Eukaryota | ||||
| Chatton, 1925 |
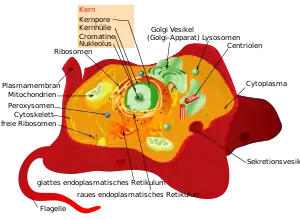
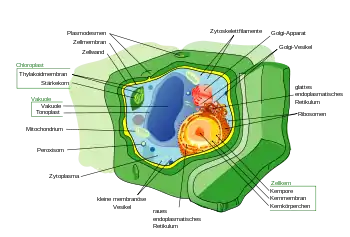
Merkmale
Die Zellen der Eukaryoten haben meistens einen Durchmesser von 10 bis 30 µm.[2] Sie sind in der Regel wesentlich größer als die von Prokaryoten, ihr Volumen beträgt etwa das 100- bis 10.000-Fache. Für ein reibungsloses Funktionieren der zellulären Abläufe über größere Entfernungen innerhalb der Zelle sind ein höherer Organisationsgrad und eine Aufteilung des Zellraums in Kompartimente (abgegrenzte Räume) sowie Transport zwischen diesen Kompartimenten günstig. Aus diesem Grund sind eukaryotische Zellen mittels Zellorganellen strukturiert, welche wie die Organe eines Körpers verschiedene Funktionen ausüben. Das namensgebende Organell ist der Zellkern mit dem Hauptanteil des genetischen Materials der eukaryotischen Zellen. Weitere Gene kommen je nach Art in Mitochondrien (Organellen, die durch chemische Reaktionen Energie zur Verfügung stellen), in Einzelfällen auch in den ihnen verwandten Hydrogenosomen sowie in fast allen Plastiden (unter anderem in den Photosynthese betreibenden Chloroplasten) vor. Dem intrazellulären Transport dienen die Organellen des Endomembransystems.
Struktur und Form wird den eukaryotischen Zellen durch das Cytoskelett verliehen, das auch der Fortbewegung dient. Es ist aus Mikrotubuli, Intermediärfilamenten und Mikrofilamenten aufgebaut. Einige Eukaryoten, beispielsweise Pflanzen und Pilze, haben auch Zellwände, die die Zellen außerhalb der Cytoplasmamembran einschließen und ihre Form bestimmen.
Eine weitere Besonderheit der Eukaryoten liegt in der Proteinbiosynthese: Anders als Prokaryoten sind Eukaryoten in der Lage, mittels derselben DNA-Information durch alternatives Spleißen unterschiedliche Proteine herzustellen.
Mehrzelligkeit
Eukaryoten können Einzeller oder mehrzellige Lebewesen sein. Diese bestehen aus einer größeren Zahl von Zellen mit gemeinsamem Stoffwechsel, wobei spezielle Zelltypen bestimmte Aufgaben übernehmen. Die meisten bekannten Mehrzeller sind Eukaryoten, darunter die Pflanzen, Tiere und mehrzelligen Pilze.
Systematik
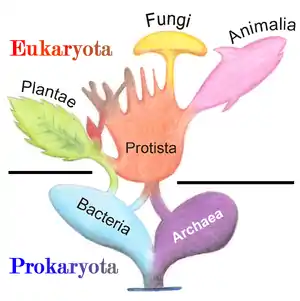

Alastair Simpson, Dalhousie University, 2020
Der Name verweist auf das Vorhandensein eines Zellkerns (Eukaryota/Eukaryonta/Eucarya/Eukarya; zu altgriechisch εὖ eû ‚gut, echt‘ und κάρυον káryon ‚Nuss‘).
In der biologischen Systematik bilden die Eukaryoten eine der drei Domänen, also der höchsten Kategorien zur Klassifizierung von Lebewesen. Die aktuelle Systematik der Eukaryoten wurde von Adl et al. 2012 aufgestellt. Sie gliedert die Eukaryoten wie folgt:[3]
- Amorphea
- super-group Amoebozoa, einzellige Organismen mit im Regelfall amöboider Gestalt, inkl. Schleimpilze und manche Amöben
- super-group Opisthokonta, inkl. Tiere und Pilze
- Diaphoretickes
- super-group Sar, inkl. Stramenopile (auch als Stramenopiles, Chromista oder Heterokonta bezeichnet)
- super-group Archaeplastida, inkl. Pflanzen
- super-group Excavata, einzellige Organismen, die mehrheitlich begeißelt sind
Daneben existieren zahlreiche Taxa mit unsicherer Stellung (incertae sedis), darunter unter anderem
- Cryptophyceae
- Centrohelida
- Haptophyta
- Telonemia
- Kamera lens, eine Geißeltierchenart und der einzige Vertreter der Gattung Kamera
- Picozoa
Systematik nach Cavalier-Smith
Cavalier-Smith gliedert die Eukaryoten wie folgt:[4][5][6][7][8][9][10]
| Eukaryota |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Entwicklungsgeschichte
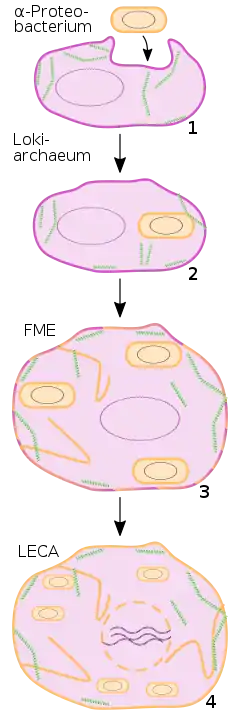
Die ältesten makroskopischen, mehrzelligen, möglicherweise eukaryotischen Fossilien sind 1,5 Milliarden Jahre alt.[13] Es ist nicht bekannt, ob die hypothetischen evolutionären Vorläufer der Eukaryoten – die sogenannten Urkaryoten[14] – bereits Organellen besaßen (diese wären aufgrund des nachteiligen Oberflächen-Volumen-Verhältnisses nötig gewesen) oder sie erst im Laufe ihrer Evolution hin zu den eigentlichen Eukaryoten erwarben. Die bekannteste Theorie zur Entstehung der Organellen ist die Endosymbiontentheorie, die besagt, dass Mitochondrien und Chloroplasten auf Bakterien zurückgehen, die von den zellbiologisch „primitiven“, räuberisch lebenden Urkaryoten ursprünglich als Nahrung aufgenommen und von denen einige durch Zufall nicht verdaut wurden, sondern eine Symbiose mit dem Urkaryoten eingingen.[15][16]
Der letzte gemeinsame Vorfahr aller heutigen Eukaryoten – in der meist englischen Fachliteratur auch als Last Eukaryotic Common Ancestor, abgekürzt LECA, bezeichnet[17] – sollte sowohl einen abgegrenzten Zellkern als auch Mitochondrien besessen haben, da alle bisher untersuchten Eukaryoten Mitochondrien, mitochondrienartige Organellen oder zumindest Kern-DNA von mitochondrialem Ursprung (übertragen durch lateralen Gentransfer) besitzen. Zwar führte Thomas Cavalier-Smith für rezente amitochondriale Einzeller mit Zellkern, die er für evolutionäre Relikte aus der Zeit vor LECA hielt, die Bezeichnung Archezoa ein,[18] jedoch mehrten sich nachfolgend Hinweise darauf, dass es sich bei den Archezoen um sekundär amitochondriale echte Eukaryoten handelt,[19] das heißt um Abkömmlinge von LECA.
Als Alternative zur Urkaryoten-Hypothese wird ein Erwerb von Mitochondrien durch Archaeen diskutiert (Wasserstoff-Hypothese[20]). Dieser Erwerb hätte vor oder gleichzeitig mit dem des Zellkerns stattgefunden haben können und eventuell ebenfalls viralen Ursprungs (unter den NCLDVs) sein können („Viral eukaryogenesis“, ein Teil der „Out-of-Virus-Hypothese“).[21] Als Kandidaten werden DNA-Viren wie die ‚Medusaviridae‘ gehandelt;[22] und auch der ‚Pseudomonas Phage 201phi2-1‘[23][24] aus der Gattung Phikzvirus, Familie Myoviridae, ein Riesen-Bakteriophage, bildet bei der Infektion einer Bakterienzelle eine kernähnliche Struktur, die Proteine abhängig von ihrer Funktion trennt,[25] diese kernartige Struktur und ihre Schlüsseleigenschaften wurden auch in den verwandten Phagen gefunden.[26]
Eine im Herbst 2020 veröffentlichte Studie legt anhand von umfangreichen Genomanalysen tatsächlich nahe, dass – obwohl bisher noch keine primär amitochondrialen Eukaryoten gefunden wurden – die Vorfahren der Eukaryonten zuerst ihre komplexes Genom mit den zugehörigen Strukturen, und danach die Mitochondrien (oder Vorläufer davon) erworben haben.[27]
Eine Zusammenfassung dieser Diskussion findet sich bei Traci Watson (2019);[28] zu den unterschiedlichen Standpunkten siehe Guglielmini et al. (2019)[29][30] und Koonin/Yutin (2018).[31]
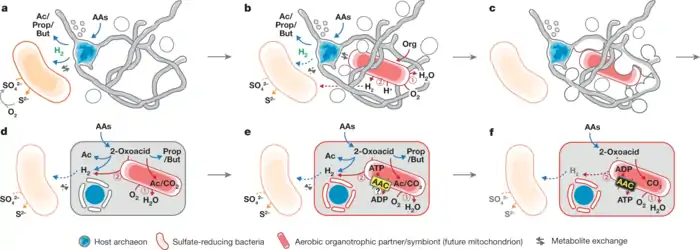
Nach 12 Jahren Forschungsarbeit berichteten Mikrobiologen 2019, erfolgreich ein sich nur sehr langsam vermehrendes Lokiarchaeon aus Tiefseeschlamm isoliert und kultiviert zu haben.[12][32][33] Candidatus Prometheoarchaeum syntrophicum ist ein, nach Vergleich seines Genoms nahe mit den Eukaryoten verwandtes, extrem langsam wachsendes heterotrophes Archaeon, das Energie aus dem Abbau von Peptiden und Aminosäuren gewinnt und dabei Wasserstoff produziert und abgibt. Das Archaeon gedeiht nur in obligater Syntrophie mit Wasserstoff-metabolierenden Organismen, einer davon ein sulfatreduzierendes Delta-Proeobakterium der Desulfovibrionales. Die Autoren nehmen an, dass treibende Kraft der Endosymbiose, die zu den Eukaryoten führte, ein zweites, in ihrem Modellsystem nicht vorhandenes Bakterium, das künftige Mitochondrium, gewesen wäre, dessen Aufgabe zunächst primär die Beseitigung von für das obligat anaerobe Archaeon giftigen Sauerstoffs gewesen sei. Die Rolle des Mitochondriums im Energiestoffwechsel wäre demnach erst sekundär entstanden. Die Autoren halten eine Schlüsselrolle des Wasserstoff-Metabolismus selbst, wie in alternativen Szenarien der Endosymbiontentheorie[34] vorgeschlagen, für unplausibel. Dementsprechend wäre das Wasserstoff-verwertende Bakterium selbst nicht zum Endosymbionten geworden. Die Hypothese setzt einen frühen Ursprung des Mitochondriums in einer noch primitiven, Archaeen-artigen Wirtszelle voraus und unterscheidet sich darin von alternativen Szenarien wie der „Archezoa“-Hypothese von Thomas Cavalier-Smith, nach denen ein fast fertiger Pro-Eukaryot erst spät ein Mitochondrium erworben hätte. Dementsprechend wäre ein „Einfangen“ des künftigen Mitochondriums über Phagocytose unplausibel, da es an den nötigen internen Strukturen wie einem Cytoskelett gefehlt hätte. Interessanterweise verfügt Prometheoarchaeum aber über tentakel-artige Zellfortsätze, mit denen ein ähnlicher Organismus im Archaikum den hypothetischen Endosymbionten alternativ quasi umschlungen haben könnte.
Die Entstehung der Chloroplasten der Photosynthese betreibenden Eukaryoten durch Endosymbiose stellt einen zeitlich späteren Vorgang dar. Zunächst wurden Cyanobakterien von nicht-phototrophen Eukaryoten aufgenommen, was möglicherweise in dieser Form (primäre Endosymbiose) nur einmal geschah. Die anderen Plastiden (Leukoplasten etc.) leiten sich von diesen ab.
Später entstanden komplexe Plastiden (mit mehr als doppelter Membran und ggf. Nukleosom) durch eine sekundäre Endosymbiose, d. h. (weitere) nicht-phototrophe Eukaryoten nahmen (aus der primären Endosymbiose entstandene) phototrophe Eukaryoten durch Endosymbiose auf (so etwa bei den Apicomplexa[35]).
Forschungsgeschichte
Die Einteilung von Lebewesen in Prokaryoten und Eukaryoten wurde erstmals von Edouard Chatton für Protisten deutlich herausgestellt und 1925 veröffentlicht.[36][37]
Diese Einteilung trat zunächst mit der Einführung des Drei-Domänen-Systems durch Carl Woese 1977 in den Hintergrund[38][39], eines Ansatzes, der das zelluläre Leben in die drei Domänen Bakterien, Archaeen und Eukaryoten aufteilt.
Im Jahr 1984 wurde von James A. Lake und Kollegen die Eozyten-Hypothese vorgeschlagen: Man hatte entdeckt, dass bei der Form der Ribosomen in der Archaeen-Gruppe der Crenarchaeota (mit der ursprünglichen Bezeichnung Eozyten) und der Eukaryoten eine erstaunliche Ähnlichkeit besteht, dass aber die Form der Ribosomen sowohl bei den Bakterien als auch bei den Euryarchaeota, einer anderen Archaeen-Gruppe, deutlich davon abweicht. Es wurde daher vermutet, dass die Eukaryoten aus den Crenarchaeota hervorgegangen sind.[40][41]
Trotz weiterer Hinweise in den 1980er Jahren bekam die Eozyten-Hypothese erst in den 2000er Jahren mit dem Fortschritt der Genom-Analyse neuen Auftrieb. In einer ganzen Reihe von Archaeen wurden Gene entdeckt, die man in ähnlicher Form auch in Eukaryoten findet. Entsprechend den Ergebnissen einiger Studien wurde in einer Abwandlung statt der Crenarchaeota die diesen nahestehende Gruppe der Thaumarchaeota als Ursprung der Eukaryoten vorgeschlagen. (Die Crenarchaeota und Thaumarchaeota werden zusammen mit einigen anderen Archaeen-Gruppen in der Supergruppe „TACK“ zusammengefasst).[40][42][43][44][45]
Mit dem Aufkommen der Metagenomanalyse ließen sich seit 2015 in Proben aus der Nähe von Hydrothermalquellen Kandidaten für Archaeen-Gruppen identifizieren, die den Eukaryoten noch viel näher stehen müssen, als alle zuvor betrachteten Gruppen. Die erste Gruppe war die der „Lokiarchaeota“, gefunden bei einem hydrothermalen Vent Namens „Loki's Schloss“ im Arktischen Ozean zwischen Mohns- und Knipovitsch-Kamm.[46] Da diese Erkenntnisse lediglich aus einer Metagenom-Analyse stammen und man die betreffenden Mikroben noch bis auf weiteres nicht kultivieren kann, kommt allen fraglichen Taxa lediglich ein 'Kandidatenstatus' zu, gekennzeichnet durch die Anführungszeichen.
Die „Lokiarchaeota“ werden aufgrund von Ähnlichkeit im Genom mit einigen ebenfalls in diesem Zusammenhang jüngst vorgeschlagenen Gruppen („Thorarchaeota“, „Odinarchaeota“ und „Heimdallarchaeota“) zu einer Kandidaten-Gruppe „Asgard“ zusammengefasst, die unter den Archaeen den Eukaryoten somit am nächsten steht und ein Schwester-Taxon zur Supergruppe „TACK“ darstellt.[47][48][49][50]
Um der engen Verwandtschaft der Archaeen mit den Eukaryoten Rechnung zu tragen, hat Thomas Cavalier-Smith sie bereits 2002 in ein gemeinsames Taxon Neomura gestellt, das damit eine Schwestergruppe zu den Bakterien darstellt.[51]
Diese neue Einteilung wird unterstützt durch neuere Erkenntnisse, wonach die Verwendung des DNA-Genoms als Träger der Erbinformationen bei Bakterien einerseits und Archaeen und Eukaryoten andererseits unterschiedlichen (möglicherweise viralen: anderer Teil der „Out-of-Virus-Hypothese“) Ursprungs zu sein scheint. Der letzte gemeinsame Vorfahr aller heute bekannten Lebewesen wäre dann ein archaischer zellulärer Organismus der RNA-Welt (mit Ribosomen, d. h. Protein-Synthese) gewesen.[21]
Weitere Unterstützung bekommt die Theorie durch Untersuchung des Aufbaus der membranständigen F- und V-/A-Typ-ATPasen. Die F-Typ-ATPasen der Mitochondrien und Chloroplasten sind mit denen der Bakterien homolog (wie nach der Endosymbiosetheorie zu erwarten). Die V-Typ-ATPasen an Zytoplasma-Membranen (Vakuolen) der Eukaryoten sind zu denen der Archaeen homolog, was eine Abstammung der Eukaryoten aus einem Zweig der Archaeen stützt. Vereinzelte Ausnahmen (F-Typ-ATPasen bei einigen Archaeen-Species[52] und V-Typ-ATPasen bei einigen Bakteriengruppen) werden auf horizontalen Gentransfer zurückgeführt.[53] Zentrale Untereinheiten der ATPasen sind über alle Lebewesen homolog, was für einen LUCA mit einer zumindest primitiven Membranhülle spricht.[54][55]
Einzelnachweise
- Eukaryoten. In: Lexikon der Biologie. Spektrum Akademischer Verlag. Heidelberg, 1999, abgerufen am 1. Oktober 2016.
- Gerald Karp, Molekulare Zellbiologie, 2005, S. 25.
- Adl, S. M., Simpson, A. G. B., Lane, C. E., Lukeš, J., Bass, D., Bowser, S. S., Brown, M. W., Burki, F., Dunthorn, M., Hampl, V., Heiss, A., Hoppenrath, M., Lara, E., le Gall, L., Lynn, D. H., McManus, H., Mitchell, E. A. D., Mozley-Stanridge, S. E., Parfrey, L. W., Pawlowski, J., Rueckert, S., Shadwick, L., Schoch, C. L., Smirnov, A. and Spiegel, F. W.: The Revised Classification of Eukaryotes. Journal of Eukaryotic Microbiology, 59: S. 429–514, 2012, doi:10.1111/j.1550-7408.2012.00644.x (PDF)
- Romain Derelle, Guifré Torruella, Vladimír Klimeš, Henner Brinkmann, Eunsoo Kim, Čestmír Vlček, B. Franz Lang, Marek Eliáš: Bacterial proteins pinpoint a single eukaryotic root. In: Proceedings of the National Academy of Sciences. 112, Nr. 7, 17. Februar 2015, ISSN 0027-8424, S. E693–E699. doi:10.1073/pnas.1420657112. PMID 25646484. PMC 4343179 (freier Volltext).
- T. Cavalier-Smith, E. E. Chao, E. A. Snell, C. Berney, A. M. Fiore-Donno, R. Lewis: Multigene eukaryote phylogeny reveals the likely protozoan ancestors of opisthokonts (animals, fungi, choanozoans) and Amoebozoa. In: Molecular Phylogenetics & Evolution. 81, 2014, S. 71–85. doi:10.1016/j.ympev.2014.08.012.
- Thomas Cavalier-Smith: Kingdoms Protozoa and Chromista and the eozoan root of the eukaryotic tree. In: Biology Letters. 6, Nr. 3, 23. Juni 2010, ISSN 1744-9561, S. 342–345. doi:10.1098/rsbl.2009.0948. PMID 20031978. PMC 2880060 (freier Volltext).
- Ding He, Omar Fiz-Palacios, Cheng-Jie Fu, Johanna Fehling, Chun-Chieh Tsai, Sandra L. Baldauf: An Alternative Root for the Eukaryote Tree of Life. In: Current Biology. 24, Nr. 4, Februar, S. 465–470. doi:10.1016/j.cub.2014.01.036.
- Laura A. Hug, Brett J. Baker, Karthik Anantharaman, Christopher T. Brown, Alexander J. Probst, Cindy J. Castelle, Cristina N. Butterfield, Alex W. Hernsdorf, Yuki Amano: A new view of the tree of life. In: Nature Microbiology. 1, Nr. 5, 11. April 2016, ISSN 2058-5276. doi:10.1038/nmicrobiol.2016.48.
- Thomas Cavalier-Smith: Kingdom Chromista and its eight phyla: a new synthesis emphasising periplastid protein targeting, cytoskeletal and periplastid evolution, and ancient divergences. In: Protoplasma. 5. September 2017, ISSN 0033-183X, S. 1–61. doi:10.1007/s00709-017-1147-3.
- Thomas Cavalier-Smith: Euglenoid pellicle morphogenesis and evolution in light of comparative ultrastructure and trypanosomatid biology: semi-conservative microtubule/strip duplication, strip shaping and transformation. In: European Journal of Protistology. Februar. doi:10.1016/j.ejop.2017.09.002.
- Eugene V. Koonin: Energetics and population genetics at the root of eukaryotic cellular and genomic complexity. In: Proceedings of the National Academy of Sciences. 112, Nr. 52, S. 15777–15778. doi:10.1073/pnas.1520869112.
- Hiroyuki Imachi, Masaru K. Nobu, Nozomi Nakahara, Yuki Morono, Miyuki Ogawara, Yoshihiro Takaki, Yoshinori Takano, Katsuyuki Uematsu, Tetsuro Ikuta, Motoo Ito, Yohei Matsui, Masayuki Miyazaki, Kazuyoshi Murata, Yumi Saito, Sanae Sakai, Chihong Song, Eiji Tasumi, Yuko Yamanaka, Takashi Yamaguchi, Yoichi Kamagata, Hideyuki Tamaki & Ken Takai: Isolation of an archaeon at the prokaryote–eukaryote interface. In: Nature. Band 577, 2020, S. 519–525, doi:10.1038/s41586-019-1916-6.
- Eukaryoten: Eine neue Zeittafel der Evolution. Newsroom, Max-Planck-Gesellschaft, 24. Mai 2015.
- C. R. Woese, G. E. Fox: Phylogenetic structure of the prokaryotic domain: the primary kingdoms. In: Proceedings of the National Academy of Sciences. Band 74, Nummer 11, November 1977, S. 5088–5090, PMID 270744, PMC 432104 (freier Volltext).
- Lynn Margulis, Dorion Sagan: Leben: Vom Ursprung zur Vielfalt. Spektrum Akademischer Verlag, Heidelberg, Berlin 1997, ISBN 3-8274-0524-6 (Übersetzung der englischsprachigen Originalausgabe von 1995).
- Lynn Margulis: Die andere Evolution. Spektrum Akademischer Verlag, Heidelberg, Berlin 1999, ISBN 3-8274-0294-8 (Übersetzung der englischsprachigen Originalausgabe von 1998).
- Eugene V. Koonin: The origin and early evolution of eukaryotes in the light of phylogenomics, in: Genome Biology. Band 11, Art.-Nr. 209, 5. Mai 2010, doi:10.1186/gb-2010-11-5-209.
- Tom Cavalier-Smith: Archaebacteria and Archezoa. In: Nature. 339, Nr. 6220, Mai 1989, S. 100–101. doi:10.1038/339100a0. PMID 2497352.
- Patrick J. Keeling: A kingdom’s progress: Archezoa and the origin of eukaryotes. In: BioEssays. Band 20, Nummer 1, Januar 1998, S. 87–95, doi:10.1002/(SICI)1521-1878(199801)20:1<87::AID-BIES12>3.0.CO;2-4 (alternativer Volltextzugriff: SemanticScholar).
- Martin W and Müller M: The hydrogen hypothesis for the first eukaryote. In: Nature. 392, Nr. 6671, 1998, S. 37–41. doi:10.1038/32096. PMID 9510246.
- Patrick Forterre: Evolution - Die wahre Natur der Viren, in: Spektrum August 2017, S. 37-41 (Online-Artikel vom 19. Juli 2017)
- Tokyo University of Science: New giant virus may help scientists better understand the emergence of complex life - Large DNA virus that helps scientists understand the origins of DNA replication and the evolution of complex life. In: EurekAlert!, 30. April 2019. Abgerufen im 2. Juli 2019.
- Pseudomonas phage 201phi2-1, auf: Virus-Host DB, TAX:198110
- Proteomes - Pseudomonas phage 201phi2-1, auf: UniProt
- V Chaikeeratisak, K Nguyen, K Khanna, AF Brilot, ML Erb, JKC Coker, A Vavilina, GL Newton, R Buschauer, K Pogliano, E Villa, DA Agard, J Pogliano: Assembly of a nucleus-like structure during viral replication in bacteria. In: Science. 355, Nr. 6321, 2017, S. 194–197. bibcode:2017Sci...355..194C. doi:10.1126/science.aal2130. PMID 28082593. PMC 6028185 (freier Volltext).
- V Chaikeeratisak, K Nguyen, ME Egan, ML Erb, A Vavilina, J Pogliano: The Phage Nucleus and Tubulin Spindle Are Conserved among Large Pseudomonas Phages. In: Cell Reports. 20, Nr. 7, 2017, S. 1563–1571. doi:10.1016/j.celrep.2017.07.064. PMID 28813669. PMC 6028189 (freier Volltext).
- Julian Vosseberg, Jolien J. E. van Hooff, Marina Marcet-Houben, Anne van Vlimmeren, Leny M. van Wijk, Toni Gabaldón, Berend Snel: Timing the origin of eukaryotic cellular complexity with ancient duplications, in: Nature Ecology & Evolution, 26. Oktober 2020, doi:10.1038/s41559-020-01320-z, dazu:
Timeline Unveiled for One of the Most Important and Puzzling Events in the Evolution of Life, auf: SciTechDaily vom 4. November 2020, Quelle: Institute for Research in Biomedicine (IRB Barcelona) – ‚Bakterien‘ ist hier im Sinn von ‚Prokaryoten‘ zu verstehen. - Traci Watson: The trickster microbes that are shaking up the tree of life, in: Nature vom 14. Mai 2019 (englisch), Trickser-Bakterien schütteln den Stammbaum des Lebens, in: Spektrum.de vom 20. Juni 2019 (deutsch) – die Bezeichnung ‚Bakterien‘ ist nicht ganz korrekt, bei den betrachteten Mikroben handelt es sich um Archaeen oder (nach Ansicht mancher Forscher) jedenfalls um von den Bakterien verschiedene Proto-Eukaryonten.
- Julien Guglielmini, Anthony C. Woo, Mart Krupovic, Patrick Forterre, Morgan Gaia: Diversification of giant and large eukaryotic dsDNnA viruses predated the origin of modern eukaryotes, in: PNAS 116(39), 10./24. September 2019, S. 19585–19592, doi:10.1073/pnas.1912006116, PMID 31506349, Fig. 2
- Julien Guglielmini, Anthony Woo, Mart Krupovic, Patrick Forterre, Morgan Gaia: Diversification of giant and large eukaryotic dsDNA viruses predated the origin of modern eukaryotes, auf: bioRxiv vom 29. Oktober 2018, bioRxiv: 10.1101/455816v1 (Preprint-Volltext), doi:10.1101/455816
- Eugene V. Koonin, Natalya Yutin: Multiple evolutionary origins of giant viruses, in: F1000 Research, 22. November 2018, doi:10.12688/f1000research.16248.1, version 1
- Christa Schleper & Filipa L. Sousa (2020): Meet the relatives of our cellular ancestor. Nature 577: 478-479. doi: 10.1038/d41586-020-00039-y
- Elizabeth Pennisi (2020): Tentacled microbe hints at how simple cells became complex. Science 365 (6454): 631. doi:10.1126/science.365.6454.631
- Purificación López-García & David Moreira (2020): The Syntrophy hypothesis for the origin of eukaryotes revisited. Nature Microbiology5: 655–667. doi:10.1038/s41564-020-0710-4
- Ralph, S.: Evolutionary Pressures on Apicoplast Transit Peptides. In: Molecular Biology and Evolution. 21, Nr. 12, 2004, S. 2183–2191. doi:10.1093/molbev/msh233. PMID 15317876. (Seite nicht mehr abrufbar, Suche in Webarchiven) Info: Der Link wurde automatisch als defekt markiert. Bitte prüfe den Link gemäß Anleitung und entferne dann diesen Hinweis.
- Edouard Chatton: Pansporella perplexa. Réflexions sur la biologie e la phylogénie des protozoaires. In: Annales des Sciences Naturelles: Zoologie Sér. 10, Band 8, 1925, S. 5–84.
- Marie-Odile Soyer-Gobillard: Edouard Chatton (1883–1947) and the dinoflagellate protists: concepts and models. In: International Microbiology. Band 9, 2006, S. 173–177. (pdf)
- Woese C, Fox G: Phylogenetic structure of the bacteria domain: the primary kingdoms. In: Proc Natl Acad Sci USA. 74, Nr. 11, 1977, S. 5088–90. bibcode:1977PNAS...74.5088W. doi:10.1073/pnas.74.11.5088. PMID 270744. PMC 432104 (freier Volltext).
- Woese C, Kandler O, Wheelis M: Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya.. In: Proc Natl Acad Sci USA. 87, Nr. 12, 1990, S. 4576–9. bibcode:1990PNAS...87.4576W. doi:10.1073/pnas.87.12.4576. PMID 2112744. PMC 54159 (freier Volltext). Abgerufen am 11. Februar 2010.
- John M. Archibald: The eocyte hypothesis and the origin of eukaryotic cells. In: PNAS. 105, Nr. 51, 23. Dezember 2008, S. 20049–20050. doi:10.1073/pnas.0811118106. Abgerufen am 5. Oktober 2012.
- James A. Lake, Eric Henderson, Melanie Oakes, Michael W. Clark: Eocytes: A new ribosome structure indicates a kingdom with a close relationship to eukaryotes. In: PNAS. 81, Nr. 12, Juni 1984, S. 3786–3790. doi:10.1073/pnas.81.12.3786. PMID 6587394. PMC 345305 (freier Volltext). Abgerufen am 5. Oktober 2012.
- S. Kelly, B. Wickstead, K. Gull: Archaeal phylogenomics provides evidence in support of a methanogenic origin of the Archaea and a thaumarchaeal origin for the eukaryotes. In: Proceedings of the Royal Society B. 278, 2011, S. 1009–1018. doi:10.1098/rspb.2010.1427. PMID 20880885. PMC 3049024 (freier Volltext). Abgerufen am 5. Oktober 2012.
- Anthony M. Poole, Nadja Neumann: Reconciling an archaeal origin of eukaryotes with engulfment: a biologically plausible update of the Eocyte hypothesis. In: Research in Microbiology. 162, 2011, S. 71–76. doi:10.1016/j.resmic.2010.10.002. Abgerufen am 5. Oktober 2012.
- Lionel Guy, Thijs J. G. Ettema: The archaeal 'TACK' superphylum and the origin of eukaryotes. In: Trends in Microbiology. 19, Nr. 12, Dezember 2011, S. 580–587. doi:10.1016/j.tim.2011.09.002. PMID 22018741. Abgerufen am 5. Oktober 2012.
- Cymon J. Cox, Peter G. Foster, Robert P. Hirt, Simon R. Harris, T. Martin Embley: The archaebacterial origin of eukaryotes. In: PNAS. 105, 23. Dezember 2008, S. 20356–20361. doi:10.1073/pnas.0810647105. PMID 19073919. PMC 2629343 (freier Volltext). Abgerufen am 5. Oktober 2012.
- Anja Spang, Jimmy H. Saw, Steffen L. Jørgensen, Katarzyna Zaremba-Niedzwiedzka, Joran Martijn, Anders E. Lind, Roel van Eijk, Christa Schleper, Lionel Guy, Thijs J. G. Ettema: Complex archaea that bridge the gap between prokaryotes and eukaryotes. In: Nature. 521, 2015, ISSN 0028-0836, S. 173–179. doi:10.1038/nature14447. PMID 25945739. PMC 4444528 (freier Volltext).
- Laura Eme, Anja Spang, Jonathan Lombard, Courtney W. Stairs, Thijs J. G. Ettema: Archaea and the origin of eukaryotes. In: Nature Reviews Microbiology. 15, Nr. 12, 10. November 2017, ISSN 1740-1534, S. 711–723. doi:10.1038/nrmicro.2017.133.
- Katarzyna Zaremba-Niedzwiedzka et al.: Asgard archaea illuminate the origin of eukaryotic cellular complexity, in: Nature 541, S. 353–358 vom 19. Januar 2017, doi:10.1038/nature21031.
- NCBI taxonomy page on Archaea.
- Bram Henneman: Genomic diversity, lifestyles and evolutionary origins of DPANN archaea: Chapter 1: Introduction, Leiden University Repository, 5. Dezember 2019. Siehe Fig. 1.1
- Cavalier-Smith T.: The phagotrophic origin of eukaryotes and phylogenetic classification of Protozoa. In: Int. J. Syst. Evol. Microbiol.. 52, Nr. Pt 2, März 2002, S. 297–354. doi:10.1099/00207713-52-2-297. PMID 11931142.
- Regina Saum et al.: The F1FO ATP synthase genes in Methanosarcina acetivorans are dispensable for growth and ATP synthesis, in: FEMS Microbiology Letters Vol. 300 Issue 2 vom November 2009, S. 230–236, doi:10.1111/j.1574-6968.2009.01785.x
- Elena Hilario, Johann Peter Gogarten: Horizontal transfer of ATPase genes - the tree of life becomes a net of life. In: BioSystems. 31, Nr. 2–3, 1993, S. 111–119. doi:10.1016/0303-2647(93)90038-E. PMID 8155843 ScienceDirect Dieser Artikel bei University of Connecticut (PDF).
- Armen Y. Mulkidjanian, Michael Y. Galperin, Kira S. Makarova, Yuri I. Wolf, Eugene V. Koonin: Evolutionary primacy of sodium bioenergetics. In: Biology Direct. 3, Nr. 13, 2008. doi:10.1186/1745-6150-3-13.
- Armen Y. Mulkidjanian, Kira S. Makarova, Michael Y. Galperin, Eugene V. Koonin: Inventing the dynamo machine: the evolution of the F-type and V-type ATPases. In: Nature Reviews Microbiology. 5, Nr. 11, 2007, S. 892–899. doi:10.1038/nrmicro1767. Dieser Artikel bei Uni Osnabrück: Perspectives (PDF) (Seite nicht mehr abrufbar, Suche in Webarchiven) Info: Der Link wurde automatisch als defekt markiert. Bitte prüfe den Link gemäß Anleitung und entferne dann diesen Hinweis.
Weblinks
- Eukaryonten Zellaufbau
- Eukaryoten (Tree of Life Web Project) (englisch)
- Botanik online
- Wilfried Probst: Frühe Evolution und Symbiose, Europa-Universität Flensburg, Institut für Biologie und Sachunterricht und ihre Didaktik: §LECA und Mitochondrien, abgerufen am 19. April 2019
- Masaharu Takemura: Medusavirus Ancestor in a Proto-Eukaryotic Cell: Updating the Hypothesis for the Viral Origin of the Nucleus, in: Front. Microbiol. 11:571831, 3. September 2020, doi:10.3389/fmicb.2020.571831, dazu:
Tessa Koumoundouros: Giant Viruses Could Explain The Mysterious Evolution of a Key Part of Our Cells, auf: scinecealert vom 10. September 2020
In Ancient Giant Viruses Lies the Truth: Medusavirus Key to Deciphering Evolutionary Mystery, auf: SciTechDaily vom 11. September 2020 - Ancestors of Legionella Bacteria – Which Causes Legionnaires’ Disease – Infected Cells Two Billion Years Ago. Auf SciTechDaily vom 15. Februar 2022. Quelle: Universität Uppsala.