FokI
FokI ist ein Restriktionsenzym aus dem Bakterium Flavobacterium okeanokoites.
| Type-2 restriction enzyme FokI | ||
|---|---|---|
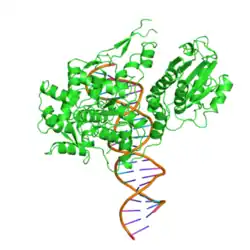 | ||
| FokI an DNA gebunden, nach PDB 1FOK | ||
| Andere Namen |
Endonuclease FokI, Type II restriction enzyme FokI, Type IIS restriction enzyme FokI | |
| Masse/Länge Primärstruktur | 583 Aminosäuren, 66.219 Da | |
| Sekundär- bis Quartärstruktur | Homodimer | |
| Bezeichner | ||
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 3.1.21.4 | |
Eigenschaften
FokI ist eine Endonuklease (Typ II, Subtyp S), die dsDNA nach der Erkennungssequenz 5'-GGATG-3' schneidet, je nach Strang 9 bzw. 13 Nukleotide hinter der Erkennungssequenz.[1] Durch den Schnitt der doppelsträngigen DNA durch FokI entsteht ein sticky end (Ende mit 4 Nukleotiden Überhang) und je einer Phosphatgruppe an beiden 5'-Enden der doppelsträngigen DNA-Produkte. FokI wird durch eine DCM-Methylierung und durch eine CpG-Methylierung gehemmt, nicht aber durch eine DAM-Methylierung. Nach einer Restriktion von DNA in vitro kann FokI durch 20-minütiges Erhitzen auf 65 °C denaturiert und somit inaktiviert werden. Meistens wird FokI als rekombinantes Protein in E.coli hergestellt. FokI schneidet dsDNA mit zwei Erkennungssequenzen in beliebiger Orientierung besser als mit einer, wobei sie mehrere hundert Nukleotide auseinander liegen können.[2]
Anwendungen
FokI wird für Restriktionsverdaue im Rahmen von Klonierungen oder Restriktionsanalysen (vor allem von Polymorphismen des Gens des Vitamin-D-Rezeptors zur Bestimmung der Anfälligkeit für Tuberkulose[3] und Nierenversagen[4]) verwendet. Der enzymatische Anteil von FokI wird im Zuge eines Proteindesigns mit den Erkennungsdomänen anderer Restriktionsenzyme als Fusionsprotein verwendet, z. B. zur Erzeugung von Zinkfingernukleasen[2] oder in Kombination mit einem inaktivierten Cas9.[5][6][7]
Einzelnachweise
- D. A. Wah, J. Bitinaite, I. Schildkraut, A. K. Aggarwal: Structure of FokI has implications for DNA cleavage. In: Proceedings of the National Academy of Sciences. Band 95, Nummer 18, September 1998, S. 10564–10569, PMID 9724743, PMC 27934 (freier Volltext).
- S. E. Halford, L. E. Catto, C. Pernstich, D. A. Rusling, K. L. Sanders: The reaction mechanism of FokI excludes the possibility of targeting zinc finger nucleases to unique DNA sites. In: Biochemical Society transactions. Band 39, Nummer 2, April 2011, S. 584–588, doi:10.1042/BST0390584, PMID 21428944.
- Y. Cao, X. Wang, Z. Cao, X. Cheng: Vitamin D receptor gene FokI polymorphisms and tuberculosis susceptibility: a meta-analysis. In: Archives of Medical Science. Band 12, Nummer 5, Oktober 2016, S. 1118–1134, doi:10.5114/aoms.2016.60092, PMID 27695504, PMC 5016579 (freier Volltext).
- L. Yang, L. Wu, Y. Fan, J. Ma: Associations among four polymorphisms (BsmI, FokI, TaqI and ApaI) of vitamin D receptor gene and end-stage renal disease: a meta-analysis. In: Archives of medical research. Band 46, Nummer 1, Januar 2015, S. 1–7, doi:10.1016/j.arcmed.2014.11.017, PMID 25434518.
- Tsai, S. Q. et al. (2014). Dimeric CRISPR RNA-guided FokI nucleases for highly specific genome editing. Nature Biotechnol. 32, 569–576 doi:10.1038/nbt.2908
- Guilinger, J. P., Thompson, D. B. & Liu, D. R. (2014). Fusion of catalytically inactive Cas9 to FokI nuclease improves the specificity of genome modification. Nature Biotechnol. 32, 577–582 doi:10.1038/nbt.2909
- Wyvekens, N., Topkar, V. V., Khayter, C., Joung, J. K. & Tsai, S. Q. (2015). Dimeric CRISPR RNA-guided FokI-dCas9 nucleases directed by truncated gRNAs for highly specific genome editing. Hum. Gene Ther. 26, 425–431 doi:10.1089/hum.2015.084