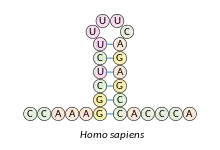
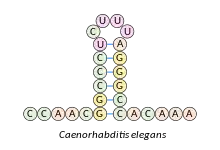
Haarnadelstruktur
Intramolekulare Basenpaarungen, die eine Haarnadelstruktur bilden, kommen in einsträngiger DNA und RNA vor. Hierbei handelt es sich um sekundäre Stamm-Schleife-Strukturen (englisch stem-loop) mit doppelsträngig ausgebildetem Stamm und kurzer einzelsträngiger Schleife, auch Haarnadel (hairpin) bzw. Haarnadelschleife (hairpin loop) genannt. Diese treten auf, wenn zwei Abschnitte des gleichen Moleküls – oft mit palindromischer Nukleotid-Sequenz – durch komplementäre Basenpaare eine doppelsträngige Region bilden und den ungepaarten Zwischenabschnitt als Schleife einschließen. Die entstehende „Lollipop-Struktur“ ist ein Schlüsselelement im Aufbau vieler RNA-Sekundärstrukturen.
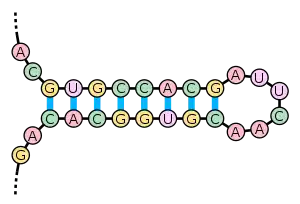
Bildung und Stabilität
Die Ausbildung einer Haarnadelstruktur hängt von der Stabilität der resultierenden Helix- und Schleifenregionen ab. Die erste Voraussetzung ist eine Sequenz, die sich auf sich selber zurückfalten kann, um eine gepaarte Doppelhelix zu bilden. Für die Stabilität dieser Helix entscheidend sind ihre Länge, die Anzahl der Basenpaarungen sowie etwaige Versetzungen bzw. Wölbungen im gepaarten Bereich (durch die insbesondere kurze Helices instabil werden). Paarungen von Guanin mit Cytosin haben drei Wasserstoffbrückenbindungen und sind damit stabiler als die Adenin-Uracil-Paare, die nur zwei Wasserstoffbrücken ausbilden. Daneben sind in RNA auch Guanin-Uracil-Paarungen mit zwei Wasserstoffbrücken üblich und günstig. Die Ausbildung einer Helix wird auch begünstigt durch Interaktionen der gestapelten Basen, die die π-Bindungen der aromatischen Ringe in eine günstige Ausrichtung bringen.
Die Stabilität der Schleife beeinflusst ebenfalls die Bildung einer Haarnadelstruktur. Es existieren keine Schleifen mit weniger als drei Basen. Große Schleifen sind ebenfalls instabil, wenn sie keine weiteren sekundären Strukturen tragen (wie etwa Pseudoknoten-Paarungen). Die optimale Schleifenlänge scheint zwischen 4 und 8 Basen zu liegen. Die Schleife mit der Sequenz UUCG wird Tetraloop genannt und ist aufgrund der Interaktionen ihrer Nukleotide besonders stabil.
Vorkommen in der RNA
Haarnadelstrukturen treten zum Beispiel in prä-microRNAs und tRNAs auf. tRNAs bestehen aus einer kleeblattförmigen Anordnung von drei echten Haarnadelstrukturen und einem beide Enden zusammenfassenden Stammbereich. Das Anticodon, mit dem während der Translation ein Codon erkannt wird, sitzt auf einer der ungepaarten Schleifen der tRNA. Neben ineinander verschachtelten Schleifenbildungen können auch miteinander verschränkte Haarnadelstrukturen auftreten, so in Pseudoknoten.
Viele Ribozyme beinhalten ebenfalls Haarnadelstrukturen, wie z. B. das Hairpin-Ribozym, welches sogar nach diesem charakteristischen Strukturmerkmal benannt wurde. Im Hammerhead-Ribozym finden sich ebenfalls Haarnadelstrukturen, die in einem zentralen ungepaarten Abschnitt zusammenhängen, wo die Schneideregion liegt. Die grundlegende Sekundärstruktur des Hammerhead-Ribozyms ist notwendig für die Schneidefunktion.
Auch bei der Termination der Transkription in Prokaryoten spielen Haarnadelstrukturen eine wichtige Rolle. Sie formen sich in einem mRNA-Strang während der Transkription und sorgen dafür, dass die RNA-Polymerase von dem DNA-Strang gelöst wird. Dieser Prozess stellt eine Rho-unabhängige oder intrinsische Termination dar, die beteiligten Sequenzen heißen Terminator-Sequenzen. Sie bilden die Grundlage für eine Form der Genregulation, die als Attenuation bekannt ist.
Beispiel
Ein Beispiel stellt die Histon-3′-UTR-Stammschleife dar. Im Tierreich wird ein Großteil der Histone (basische Proteine, die im Zellkern die enge spiralförmige Packung der DNA ermöglichen) von replikationsabhängigen Histon-Genen kodiert, deren mRNA am 3‘-Ende nicht polyadenyliert endet (Poly(A)-Schwanz), sondern in einer evolutionär konservierten Sequenz aus 25 bis 26 Nukleotiden. Diese Sequenz umfasst die Histon-3′-UTR-Stammschleife mit 6 Nukleotidpaaren im Stamm und 4 Nukleotiden in der Schleife, flankiert von 5 vorangehenden und 4 bis 5 nachfolgenden Nukleotiden. Die Struktur interagiert mit dem Stem-loop binding protein (SLBP, Stamm-Schleifen-Bindungsprotein), das an mehreren Schritten des Histon-mRNA-Metabolismus beteiligt ist, wie etwa der Steuerung der Prozessierung von prä-mRNA in die reife Form, der Translation und dem mRNA-Abbau.[1]
Siehe auch
Literatur
- J. D. Watson, T. A. Baker, S. P. Bell, A. Gann, M. Levine, R. Losick: Molecular Biology of the Gene. 5. Auflage. CSHL Press Pearson Benjamin Cummings, 2004, ISBN 0-8053-4635-X, Kapitel 6.
Einzelnachweise
- W.F. Marzluff, E.J. Wagner, R.J. Duronio: Metabolism and regulation of canonical histone mRNAs: life without a poly(A) tail. In: Nature Reviews Genetics. Band 9, 2008, doi:10.1038/nrg2438.