CRISPRi
CRISPRi (von CRISPR interference) ist eine biochemische Methode zur Hemmung einer bestimmten Transkription, die eine Genexpression des blockierten Gens hemmt (Gen-Knockdown).[1]
Eigenschaften
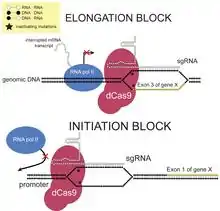
CRISPRi basiert – wie auch die CRISPR/Cas-Methode und CRISPRa – auf einem antiviralen Abwehrmechanismus in Bakterien, dem CRISPR.[2] CRISPRi verwendet einen RNA-Proteinkomplex, der an eine DNA-Sequenz binden kann und dort eine Genexpression gewünschter Gene einleiten kann. Dabei wird die Eigenschaft von Cas9 mit einer sgRNA genutzt, an eine gewünschte DNA-Sequenz zu binden.[3] Da Cas9 mit sgRNA natürlicherweise auch die gebundene DNA schneidet, wird eine Mutante von Cas9 namens dCas9 (von dead Cas9) verwendet, die keine DNA mehr schneiden kann, weil die Endonukleasefunktion für beide DNA-Stränge durch bestimmte Punktmutationen (D10A und H840A) deaktiviert ist.[4] In eukaryotischen Zellen wird dCas9 mit einem Kernlokalisierungssignal verwendet, damit dCas9 in den Zellkern importiert wird.[5] In Bakterien kann CRISPRi sowohl in Gram-negativen E. coli[6][7] als auch Gram-positiven B. subtilis verwendet werden.[8] Durch bakterielle Konjugation kann CRISPRi im Sinne eines horizontalen Gentransfers an andere Bakterien und Bakterienarten weitergegeben werden.[9] Aufgrund der einfachen modularen Gestaltung der sgRNA eignet sich CRISPRi zum Hochdurchsatz-Screening.[10] Durch Verwendung mehrerer sgRNA können parallel mehrere Gene gehemmt werden.
Zur Verstärkung der Hemmwirkung kann ein Fusionsprotein von dCas9 mit einem Repressor verwendet werden, z. B. mit der Proteindomäne Krüppel associated box (KRAB), wodurch die Transkription des gebundenen Gens in menschlichen Zellen zu 99 % unterdrückt wird.[11] Weitere Faktoren, welche die Effizienz von CRISPRi beeinflussen, sind die Bindung der korrekten Sequenz des Startpunktes der Transkription, das Design der sgRNA und zugängliche Bereiche im Chromatin.[12] Durch eine suboptimale Bindung der sgRNA an die DNA-Zielsequenz können auch geringere Hemmungsquoten erzielt werden.[10]
Die Notwendigkeit eines Vorhandenseins eines Protospacer Adjacent Motif (PAM) in der zu bindenden DNA begrenzt die Anzahl möglicher Zielsequenzen.[10] Chromatinfaltungen und DNA-Modifikationen könnten die Bindung des dCas9-sgRNA-Komplexes stören.[10] CRISPRi kann auch andere Gene in unmittelbarer Umgebung zur Zielsequenz hemmen, die beispielsweise gespleißt werden oder auf dem gegenüberliegenden DNA-Strang liegen. Ebenso kann auch ein bidirektionaler Promotor in beiden Richtungen gehemmt werden.[13]
Eine alternative Methode zur Hemmung einer Genexpression verwendet die RNA-Interferenz. Während CRISPRi die Transkription eines bestimmten Gens hemmt, führt die RNA-Interferenz zu einem Abbau bestimmter RNA-Sequenzen.
Einzelnachweise
- L. S. Qi, M. H. Larson, L. A. Gilbert, J. A. Doudna, J. S. Weissman, A. P. Arkin, W. A. Lim: Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. In: Cell. Band 152, Nummer 5, Februar 2013, S. 1173–1183, doi:10.1016/j.cell.2013.02.022, PMID 23452860, PMC 3664290 (freier Volltext).
- R. Barrangou, C. Fremaux, H. Deveau, M. Richards, P. Boyaval, S. Moineau, D. A. Romero, P. Horvath: CRISPR provides acquired resistance against viruses in prokaryotes. In: Science. Band 315, Nummer 5819, März 2007, S. 1709–1712, doi:10.1126/science.1138140, PMID 17379808.
- W. Jiang, D. Bikard, D. Cox, F. Zhang, L. A. Marraffini: RNA-guided editing of bacterial genomes using CRISPR-Cas systems. In: Nature Biotechnology. Band 31, Nummer 3, März 2013, S. 233–239, doi:10.1038/nbt.2508, PMID 23360965, PMC 3748948 (freier Volltext).
- D. J. Brocken, M. Tark-Dame, R. T. Dame: dCas9: A Versatile Tool for Epigenome Editing. In: Current issues in molecular biology. Band 26, 2018, S. 15–32, doi:10.21775/cimb.026.015, PMID 28879853.
- L. S. Qi, M. H. Larson, L. A. Gilbert, J. A. Doudna, J. S. Weissman, A. P. Arkin, W. A. Lim: Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. In: Cell. Band 152, Nummer 5, Februar 2013, S. 1173–1183, doi:10.1016/j.cell.2013.02.022, PMID 23452860, PMC 3664290 (freier Volltext).
- W. Jiang, D. Bikard, D. Cox, F. Zhang, L. A. Marraffini: RNA-guided editing of bacterial genomes using CRISPR-Cas systems. In: Nature Biotechnology. Band 31, Nummer 3, März 2013, S. 233–239, doi:10.1038/nbt.2508, PMID 23360965, PMC 3748948 (freier Volltext).
- X. T. Li, Y. Jun, M. J. Erickstad, S. D. Brown, A. Parks, D. L. Court, S. Jun: tCRISPRi: tunable and reversible, one-step control of gene expression. In: Scientific Reports. Band 6, 12 2016, S. 39076, doi:10.1038/srep39076, PMID 27996021, PMC 5171832 (freier Volltext).
- J. M. Peters, A. Colavin, H. Shi, T. L. Czarny, M. H. Larson, S. Wong, J. S. Hawkins, C. H. Lu, B. M. Koo, E. Marta, A. L. Shiver, E. H. Whitehead, J. S. Weissman, E. D. Brown, L. S. Qi, K. C. Huang, C. A. Gross: A Comprehensive, CRISPR-based Functional Analysis of Essential Genes in Bacteria. In: Cell. Band 165, Nummer 6, Juni 2016, S. 1493–1506, doi:10.1016/j.cell.2016.05.003, PMID 27238023, PMC 4894308 (freier Volltext).
- W. Ji, D. Lee, E. Wong, P. Dadlani, D. Dinh, V. Huang, K. Kearns, S. Teng, S. Chen, J. Haliburton, G. Heimberg, B. Heineike, A. Ramasubramanian, T. Stevens, K. J. Helmke, V. Zepeda, L. S. Qi, W. A. Lim: Specific gene repression by CRISPRi system transferred through bacterial conjugation. In: ACS synthetic biology. Band 3, Nummer 12, Dezember 2014, S. 929–931, doi:10.1021/sb500036q, PMID 25409531, PMC 4277763 (freier Volltext).
- M. H. Larson, L. A. Gilbert, X. Wang, W. A. Lim, J. S. Weissman, L. S. Qi: CRISPR interference (CRISPRi) for sequence-specific control of gene expression. In: Nature protocols. Band 8, Nummer 11, November 2013, S. 2180–2196, doi:10.1038/nprot.2013.132, PMID 24136345, PMC 3922765 (freier Volltext).
- L. A. Gilbert, M. H. Larson, L. Morsut, Z. Liu, G. A. Brar, S. E. Torres, N. Stern-Ginossar, O. Brandman, E. H. Whitehead, J. A. Doudna, W. A. Lim, J. S. Weissman, L. S. Qi: CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. In: Cell. Band 154, Nummer 2, Juli 2013, S. 442–451, doi:10.1016/j.cell.2013.06.044, PMID 23849981, PMC 3770145 (freier Volltext).
- A. Radzisheuskaya, D. Shlyueva, I. Müller, K. Helin: Optimizing sgRNA position markedly improves the efficiency of CRISPR/dCas9-mediated transcriptional repression. In: Nucleic acids research. Band 44, Nummer 18, Oktober 2016, S. e141, doi:10.1093/nar/gkw583, PMID 27353328, PMC 5062975 (freier Volltext).
- Ashish Goyal, Ksenia Myacheva, Matthias Groß, Marcel Klingenberg, Berta Duran Arqué, Sven Diederichs: Challenges of CRISPR/Cas9 applications for long non-coding RNA genes. In: Nucleic Acids Research. , S. E12, doi:10.1093/nar/gkw883. PMID 27694625. PMC 5388423 (freier Volltext).