CRISPRa
CRISPRa (von CRISPR activation) ist eine biochemische Methode zur Erzeugung von Transkriptionsfaktoren mit bestimmbarer DNA-Zielsequenz.[1]
Eigenschaften
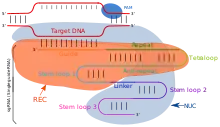
CRISPRa basiert – wie auch die CRISPR/Cas-Methode und CRISPRi – auf einem antiviralen Abwehrmechanismus in Bakterien, dem CRISPR.[2] CRISPRa verwendet einen RNA-Proteinkomplex, der an eine DNA-Sequenz binden kann und dort eine Genexpression gewünschter Gene einleiten kann. Dabei wird die Eigenschaft von Cas9 mit einer sgRNA genutzt, an eine gewünschte DNA-Sequenz zu binden.[3] Da Cas9 mit sgRNA natürlicherweise auch die gebundene DNA schneidet, wird eine Mutante von Cas9 namens dCas9 (von dead Cas9) verwendet, die keine DNA mehr schneiden kann, weil die Endonukleasefunktion für beide DNA-Stränge durch bestimmte Punktmutationen (D10A und H840A) deaktiviert ist.[4] In eukaryotischen Zellen wird dCas9 mit einem Kernlokalisierungssignal verwendet, damit dCas9 in den Zellkern importiert wird. dCas9 wird für CRISPRa mit einem Transkriptionsfaktor kombiniert, dabei entsteht ein Fusionsprotein aus dCas9 und Transkriptionsfaktor - entweder N- oder C-terminal. Der Anteil des Transkriptionsfaktors bindet weitere Proteine, die für eine Genexpression notwendig sind.
Die Zielsequenz, die beim CRISPRa gebunden wird, benötigt ein Protospacer adjacent Motif, damit dCas9 bindet. Daneben muss die sgRNA eine zur Zielsequenz revers-komplementäre Sequenz aufweisen, damit eine Bindung durch Basenpaarung zwischen sgRNA und Zielsequenz erfolgen kann. Weiterhin sollte sichergestellt sein, dass die sgRNA nur an die gewünschte Zielsequenz bindet, und nicht an anderen identischen oder sehr ähnlichen Zielsequenzen im verwendeten Organismus.[5] Die Zielsequenz wird oftmals in einem Promotor-Bereich gelegt, um den Transkriptionsfaktor in die Nähe eines Gens zu bringen. Gelegentlich werden gleichzeitig verschiedene Zielsequenzen und Transkriptionsfaktoren verwendet, um die Genexpression zu verstärken.[6][7]
CRISPRa wird für das gezielte Aktivieren von Genen,[8] wie beispielsweise zur Änderung der Zelldifferenzierung[9][10] (einschließlich der Erzeugung von induzierten pluripotenten Stammzellen),[11][12] für Hochdurchsatz-Screening von Genen[13] und für die Überexpression von Proteinen verwendet.[14] Dabei wird die sgRNA und das Fusionsprotein (aus dCas9 und Transkriptionsfaktor) durch ein Plasmid oder einen viralen Vektor codiert und in Zellen eingebracht. Durch Verwendung dieser beiden Varianten in Kombination mit einem induzierbaren Promotor kann die Genexpression zeitlich gesteuert werden.[15]
Typen
In Eukaryoten
In Eukaryoten, genauer in Zellkulturen der Fruchtfliege, der Hausmaus und des Menschen werden als Transkriptionsfaktoren P64-p65-Rta, SAM (Synergistic activation mediator) und SunTag verwendet.[16] Die Zielsequenz sollte 50 bis 500 Nukleotide vor dem zu aktivierenden Gen liegen.[5]
VP64-p65-Rta
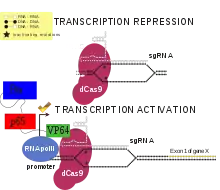
Das VP64-p65-Rta (auch VPR) verwendet dCas9 und sgRNA im Zusammenhang mit den drei Transkriptionsfaktoren Vp64, p65 und Rta in Serie am C-Terminus von dCas9. Durch die Verwendung von drei Transkriptionsfaktoren wird die Genexpression im Vergleich zu nur einem verstärkt. Durch die Verwendung von verschiedenen sgRNA können mehrere Zielsequenzen und somit auch verschiedene Gene innerhalb einer Zelle aktiviert werden.[17]
Synergistic activation mediator
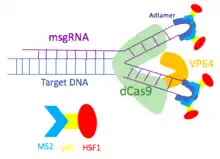
Der Synergistic activation mediator (SAM) verwendet die drei Transkriptionsfaktoren MS2, p65 und HSF1. In der dabei verwendeten sgRNA befinden sich in der tetra loop und der stem loop 2 Aptamere zur Bindung von MS2, sodass MS2-Dimere gebildet werden. Das MS2 ist ein Fusionsprotein mit p65 und HSF1.[18][19]
SunTag
.jpg.webp)
Das SunTag ist ein repetitives Epitop (10-fach), das an ein modifiziertes dCas9 fusioniert wurde und mehrere scFv-Fragment mit dem Transkriptionsfaktor VP-64 bindet.[20][21] Damit die scFv-Fragmente in den Zellkern transportiert werden, werden sie mit einem Kernlokalisierungssignal modifiziert.
In Prokaryoten
In Prokaryoten werden CRISPRa-Varianten verwendet, die an σ70- oder σ54-abhängigen Promotoren die Genexpression einleiten.[22][15][1] Bei der Verwendung von σ54-abhängigen Promotoren als Zielsequenz wurde eine bis zu 1000-fache Verstärkung der Genexpression und einen 70-fachen Unterschied von korrekter Bindung im Vergleich zu einer Basenfehlpaarung der Bindung an die Zielsequenz beschrieben.[1]
Literatur
- M. N. Hsu, Y. H. Chang, V. A. Truong, P. L. Lai, T. K. Nguyen, Y. C. Hu: CRISPR technologies for stem cell engineering and regenerative medicine. In: Biotechnology Advances. [elektronische Veröffentlichung vor dem Druck] September 2019, doi:10.1016/j.biotechadv.2019.107447, PMID 31513841.
Einzelnachweise
- Y. Liu, X. Wan, B. Wang: Engineered CRISPRa enables programmable eukaryote-like gene activation in bacteria. In: Nature Communications. Band 10, Nummer 1, August 2019, S. 3693, doi:10.1038/s41467-019-11479-0, PMID 31451697, PMC 6710252 (freier Volltext).
- R. Barrangou, C. Fremaux, H. Deveau, M. Richards, P. Boyaval, S. Moineau, D. A. Romero, P. Horvath: CRISPR provides acquired resistance against viruses in prokaryotes. In: Science. Band 315, Nummer 5819, März 2007, S. 1709–1712, doi:10.1126/science.1138140, PMID 17379808.
- W. Jiang, D. Bikard, D. Cox, F. Zhang, L. A. Marraffini: RNA-guided editing of bacterial genomes using CRISPR-Cas systems. In: Nature Biotechnology. Band 31, Nummer 3, März 2013, S. 233–239, doi:10.1038/nbt.2508, PMID 23360965, PMC 3748948 (freier Volltext).
- D. J. Brocken, M. Tark-Dame, R. T. Dame: dCas9: A Versatile Tool for Epigenome Editing. In: Current issues in molecular biology. Band 26, 2018, S. 15–32, doi:10.21775/cimb.026.015, PMID 28879853.
- S. E. Mohr, Y. Hu, B. Ewen-Campen, B. E. Housden, R. Viswanatha, N. Perrimon: CRISPR guide RNA design for research applications. In: The FEBS journal. Band 283, Nummer 17, 09 2016, S. 3232–3238, doi:10.1111/febs.13777, PMID 27276584, PMC 5014588 (freier Volltext).
- Perez-Pinera P, Kocak DD, Vockley CM, Adler AF, Kabadi AM, Polstein LR, Thakore PI, Glass KA, Ousterout DG, Leong KW, Guilak F, Crawford GE, Reddy TE, Gersbach CA: RNA-guided gene activation by CRISPR-Cas9-based transcription factors. In: Nature Methods. 10, Nr. 10, Oktober 2013, S. 973–6. doi:10.1038/nmeth.2600. PMID 23892895. PMC 3911785 (freier Volltext).
- Maeder ML, Linder SJ, Cascio VM, Fu Y, Ho QH, Joung JK: CRISPR RNA-guided activation of endogenous human genes. In: Nature Methods. 10, Nr. 10, Oktober 2013, S. 977–9. doi:10.1038/nmeth.2598. PMID 23892898. PMC 3794058 (freier Volltext).
- M. Gebre, J. L. Nomburg, B. E. Gewurz: CRISPR-Cas9 Genetic Analysis of Virus-Host Interactions. In: Viruses. Band 10, Nummer 2, 01 2018, S. , doi:10.3390/v10020055, PMID 29385696, PMC 5850362 (freier Volltext).
- A. A. Dominguez, W. A. Lim, L. S. Qi: Beyond editing: repurposing CRISPR-Cas9 for precision genome regulation and interrogation. In: Nature reviews. Molecular cell biology. Band 17, Nummer 1, Januar 2016, S. 5–15, doi:10.1038/nrm.2015.2, PMID 26670017, PMC 4922510 (freier Volltext).
- K. Takahashi, S. Yamanaka: Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors. In: Cell. Band 126, Nummer 4, August 2006, S. 663–676, doi:10.1016/j.cell.2006.07.024, PMID 16904174.
- N. A. Kearns, R. M. Genga, M. S. Enuameh, M. Garber, S. A. Wolfe, R. Maehr: Cas9 effector-mediated regulation of transcription and differentiation in human pluripotent stem cells. In: Development. Band 141, Nummer 1, Januar 2014, S. 219–223, doi:10.1242/dev.103341, PMID 24346702, PMC 3865759 (freier Volltext).
- J. Hu, Y. Lei, W. K. Wong, S. Liu, K. C. Lee, X. He, W. You, R. Zhou, J. T. Guo, X. Chen, X. Peng, H. Sun, H. Huang, H. Zhao, B. Feng: Direct activation of human and mouse Oct4 genes using engineered TALE and Cas9 transcription factors. In: Nucleic acids research. Band 42, Nummer 7, April 2014, S. 4375–4390, doi:10.1093/nar/gku109, PMID 24500196, PMC 3985678 (freier Volltext).
- M. Kampmann: CRISPRi and CRISPRa Screens in Mammalian Cells for Precision Biology and Medicine. In: ACS chemical biology. Band 13, Nummer 2, 02 2018, S. 406–416, doi:10.1021/acschembio.7b00657, PMID 29035510, PMC 5886776 (freier Volltext).
- M. F. La Russa, L. S. Qi: The New State of the Art: Cas9 for Gene Activation and Repression. In: Molecular and Cellular Biology. Band 35, Nummer 22, November 2015, S. 3800–3809, doi:10.1128/MCB.00512-15, PMID 26370509, PMC 4609748 (freier Volltext).
- C. Dong, J. Fontana, A. Patel, J. M. Carothers, J. G. Zalatan: Synthetic CRISPR-Cas gene activators for transcriptional reprogramming in bacteria. In: Nature Communications. Band 9, Nummer 1, 06 2018, S. 2489, doi:10.1038/s41467-018-04901-6, PMID 29950558, PMC 6021436 (freier Volltext).
- Chavez A, Tuttle M, Pruitt BW, Ewen-Campen B, Chari R, Ter-Ovanesyan D, Haque SJ, Cecchi RJ, Kowal EJ, Buchthal J, Housden BE, Perrimon N, Collins JJ, Church G: Comparison of Cas9 activators in multiple species. In: Nature Methods. 13, Nr. 7, Juli 2016, S. 563–567. doi:10.1038/nmeth.3871. PMID 27214048. PMC 4927356 (freier Volltext).
- Chavez A, Scheiman J, Vora S, Pruitt BW, Tuttle M, EP, Lin S, Kiani S, Guzman CD, Wiegand DJ, Ter-Ovanesyan D, Braff JL, Davidsohn N, Housden BE, Perrimon N, Weiss R, Aach J, Collins JJ, Church GM: Highly efficient Cas9-mediated transcriptional programming. In: Nature Methods. 12, Nr. 4, April 2015, S. 326–8. doi:10.1038/nmeth.3312. PMID 25730490. PMC 4393883 (freier Volltext).
- Zhang Y, Yin C, Zhang T, Li F, Yang W, Kaminski R, Fagan PR, Putatunda R, Young WB, Khalili K, Hu W: CRISPR/gRNA-directed synergistic activation mediator (SAM) induces specific, persistent and robust reactivation of the HIV-1 latent reservoirs. In: Scientific Reports. 5, Nr. 1, November 2015, S. 16277. bibcode:2015NatSR...516277Z. doi:10.1038/srep16277. PMID 26538064. PMC 4633726 (freier Volltext).
- Konermann S, Brigham MD, Trevino AE, Joung J, Abudayyeh OO, Barcena C, Hsu PD, Habib N, Gootenberg JS, Nishimasu H, Nureki O, Zhang F: Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. In: Nature. 517, Nr. 7536, Januar 2015, S. 583–8. bibcode:2015Natur.517..583K. doi:10.1038/nature14136. PMID 25494202. PMC 4420636 (freier Volltext).
- L. A. Gilbert, M. A. Horlbeck, B. Adamson, J. E. Villalta, Y. Chen, E. H. Whitehead, C. Guimaraes, B. Panning, H. L. Ploegh, M. C. Bassik, L. S. Qi, M. Kampmann, J. S. Weissman: Genome-Scale CRISPR-Mediated Control of Gene Repression and Activation. In: Cell. Band 159, Nummer 3, Oktober 2014, S. 647–661, doi:10.1016/j.cell.2014.09.029, PMID 25307932, PMC 4253859 (freier Volltext).
- Tanenbaum ME, Gilbert LA, Qi LS, Weissman JS, Vale RD: A protein-tagging system for signal amplification in gene expression and fluorescence imaging. In: Cell. 159, Nr. 3, Oktober 2014, S. 635–46. doi:10.1016/j.cell.2014.09.039. PMID 25307933. PMC 4252608 (freier Volltext).
- D. Bikard, W. Jiang, P. Samai, A. Hochschild, F. Zhang, L. A. Marraffini: Programmable repression and activation of bacterial gene expression using an engineered CRISPR-Cas system. In: Nucleic acids research. Band 41, Nummer 15, August 2013, S. 7429–7437, doi:10.1093/nar/gkt520, PMID 23761437, PMC 3753641 (freier Volltext).