RNA-Interferenz
Die RNA-Interferenz (kurz RNAi oder auch RNA-Silencing) ist ein natürlicher Mechanismus in den Zellen von Lebewesen mit einem Zellkern (Eukaryoten), welcher der zielgerichteten Abschaltung von Genen dient. Sie ist ein Spezialfall der Gen-Stilllegung. Die RNA-Interferenz beruht auf einer Wechselwirkung kurzer Stücke von Ribonukleinsäure (RNA) mit der Erbinformation-übertragenden mRNA unter Beteiligung mehrerer Enzymkomplexe. Als Folge wird die mRNA in mehrere Bruchstücke gespalten und die zu übertragende Information wird zerstört oder eine Translation in ein Protein verhindert.
In den Biowissenschaften hat sich RNA-Interferenz als eine neue Möglichkeit zur Stilllegung von Genen („Gen-Knockdown“) etabliert. Seit 2018 sind bereits erste RNAi-basierte Therapeutika zugelassen, zahlreiche weitere werden klinisch entwickelt. Für die Entdeckung des Mechanismus der RNA-Interferenz erhielten die beiden US-Wissenschaftler Andrew Z. Fire und Craig C. Mello 2006 den Nobelpreis für Physiologie oder Medizin.
Vorkommen

Das Phänomen der RNA-Interferenz kann in allen Reichen eukaryotischer Lebewesen, einschließlich Pilzen, Pflanzen und Tieren, beobachtet werden. Daher wird angenommen, dass die RNA-Interferenz ein entwicklungsgeschichtlich sehr alter Mechanismus ist.[1] Besonders gut sind die Mechanismen der RNA-Interferenz in den biologischen Modellorganismen Arabidopsis thaliana (Ackerschmalwand), Caenorhabditis elegans und Drosophila melanogaster untersucht. Insbesondere der Fadenwurm C. elegans dient als Modellorganismus zur Erforschung der RNA-Interferenz, da in diesen relativ einfach gebauten Organismus besonders leicht interferierende RNA über die als Nahrung dienenden genetisch veränderten E.-coli-Bakterien eingebracht werden kann und bei ihm robuste Interferenzeffekte zu beobachten sind.[2]
Einige Bestandteile der RNA-Interferenz-Maschinerie, wie beispielsweise die für Funktion der RNA-Interferenz essenziellen Argonautenproteine, kommen auch in Prokaryoten vor.[1] Ein der RNA-Interferenz der Eukaryoten weitgehend ähnelnder Prozess ist der CRISPR-Mechanismus der Prokaryoten zur Verteidigung gegen das Eindringen fremden Erbguts durch Viren.
Funktion
In der Natur kommen verschiedene Typen interferierender RNA vor. Wenngleich diese verschiedenen Typen interferierender RNA gemeinsame oder verwandte Mechanismen nutzen, so ist ihre Funktion zum Teil sehr verschiedenartig.
Virusabwehr
Die RNA-Interferenz spielt insbesondere bei Pflanzen bei der Verteidigung gegen fremde RNA eine wichtige Rolle. Sie stellt somit einen zentralen Bestandteil des pflanzlichen Abwehrsystems gegen RNA-Viren dar. An dieser Verteidigungsfunktion ist die siRNA (small interfering RNA), die in pflanzlichen Zellen als eine Folge der Infektion mit einem RNA-Virus bei der Vervielfältigung (Replikation) der Virus-RNA gebildet wird und zugleich der Zelle zur Erkennung und Zerstörung dieser fremden RNA dient, beteiligt. Zahlreiche Viren versuchen ihrerseits über eine Hemmung der an der RNA-Interferenz beteiligten Proteine, diesem Abwehrmechanismus zu entgehen.[3]
Ähnliche Mechanismen der RNA-Interferenz zur Infektionsabwehr konnten auch bei Pilzen, Fadenwürmern und Insekten gefunden werden. Bei Säugetieren ist das Vorkommen eines körpereigenen siRNA-basierten Abwehrmechanismus nicht gesichert. Diese Aufgabe kann bei Säugetieren von spezieller zelleigener miRNA (micro RNA) mit einer direkten hemmenden Wirkung auf die Replikation von Viren wahrgenommen werden.[3]
Regulation der Genexpression
Die RNA-Interferenz spielt unter Beteiligung der zelleigenen miRNA bei vielen mehrzelligen Lebewesen bei der Regulation der Genexpression eine wichtige Rolle. Die im menschlichen Organismus auf etwa 1000 geschätzten verschiedenen miRNAs kontrollieren die Aktivität von etwa 30 % der menschlichen Gene.[4] Hiervon sind unter anderem zahlreiche Funktionen des Immunsystems betroffen. Damit wird der Regulation der Genaktivität durch miRNA eine ähnlich große Bedeutung beigemessen wie der Regulation durch Transkriptionsfaktoren.
Auch eine zelleigene Variante der siRNA, die sogenannte esiRNA (endogenous siRNA), ist an der Regulation der Genexpression beteiligt. Diese sowohl bei Pflanzen, Pilzen als auch Tieren vorkommende interferierende RNA dient im Gegensatz zur exogenen siRNA nicht der Virusabwehr.[5]
Kontrolle der Transposons
Eine weitere Aufgabe, die Kontrolle sogenannter springender Gene (Transposons), wird insbesondere von einem weiteren Typ interferierender RNA-Moleküle wahrgenommen, der piRNA (PIWI interacting RNA). Diese Funktion der RNA-Interferenz spielt beim Menschen eine besondere Rolle bei der Spermatogenese und der Entwicklung des Embryos.
Mechanismus
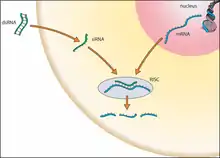
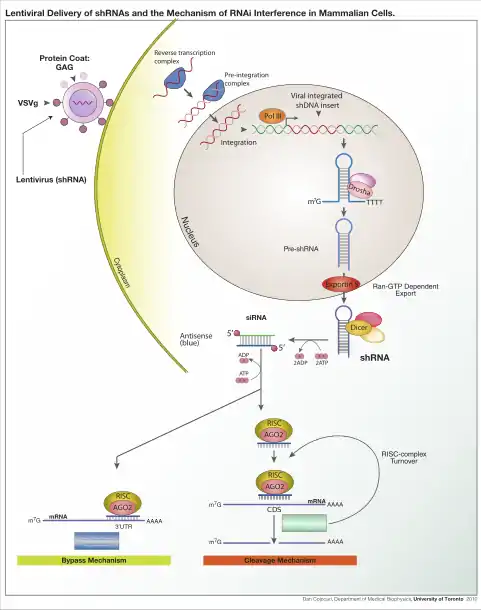
Es sind mehrere einander verwandte Mechanismen der RNA-Interferenz bekannt. An diesen Mechanismen sind als Komponenten kleine spezialisierte zumeist doppelsträngige RNA-Moleküle, ein als RNA-induced silencing complex (RISC) bezeichneter Enzymkomplex und die Ziel-RNA beteiligt. Die RNA-Interferenz kann im Allgemeinen in drei Phasen unterteilt werden. Im ersten Schritt werden in der Zelle größere doppelsträngige RNA-Moleküle mit Hilfe von Ribonuklease-Enzymen, wie Dicer und Drosha, in kurze doppelsträngige RNA-Fragmente geschnitten. Im zweiten Schritt werden diese Fragmente in Einzelstränge gespalten, und ein Strang, der sogenannte ‚Leitstrang‘ (nicht zu verwechseln mit dem Leitstrang der DNA-Replikation), wird in den RISC-Enzymkomplex aufgenommen. Letztlich kann der aufgenommene Leitstrang den Enzymkomplex aktivieren, eine mRNA zu spalten, die in ihrer Basensequenz zu der Sequenz des Leitstrangs komplementär ist. Auf diese Weise bestimmt der Leitstrang, welche mRNA in welcher Weise gespalten wird. Alternativ dazu kann der Enzymkomplex die Funktion einer zum Leitstrang komplementären mRNA als Informationsüberträger ohne Spaltung blockieren. Beide Wege können zu einer Unterdrückung der Umsetzung einer genetischen Information über ein Protein in ein Merkmal führen.[6]
Bildung interferierender RNA
Ein erster zentraler Schritt der RNA-Interferenz ist die Bildung doppelsträngiger RNA-Moleküle (dsRNA) mit einer Länge von etwa 20 bis 30 Basenpaaren. Abhängig von ihrer Herkunft kann zwischen verschiedenen Typen kurzer doppelsträngiger RNA-Moleküle unterschieden werden.
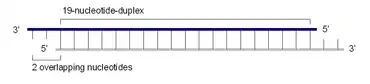
Die siRNA, eine etwa 19 bis 23 Basenpaare umfassende doppelsträngige kleine RNA mit jeweils zwei 3'-endständig überstehenden Nukleotiden, wird durch eine Spaltung eines großen doppelsträngigen RNA-Moleküls gebildet. Diese Vorläufer-RNA kann mehrere Hunderte bis Tausende Basenpaare lang sein und fällt beispielsweise bei der Vervielfältigung viraler RNA an. dsRNA kann nicht nur exogenen, sondern auch endogenen Ursprungs sein.[7] An der Spaltung ist insbesondere das Enzym Dicer, eine sogenannte RNase III, beteiligt. Dieser Mechanismus wird insbesondere zur Verteidigung gegen RNA-Viren genutzt.

Dem gegenüber ist die miRNA durch die genomische DNA codiert. In einem mehrstufigen Prozess wird aus nichtproteincodierenden Bereichen der DNA die miRNA gebildet. Primär wird pri-miRNA (primary miRNA) mit Hilfe der RNA-Polymerase II durch Transkription als RNA-Einzelstrang erzeugt, der sich aufgrund beträchtlicher palindromischer Bereiche zu einer charakteristischen Sekundärstruktur faltet. In tierischen Zellen wird die pri-miRNA noch im Zellkern mit Hilfe eines Enzymkomplexes, dem Microprocessor complex, unter Beteiligung der RNase Drosha in die haarnadelförmige pre-miRNA (precursor miRNA) mit einer Länge von 65 bis 70 Nukleotiden gespalten. Die gebildete pre-miRNA wird schließlich im Zytosol mit Hilfe von Dicer in einen jeweils etwa 21 bis 25 Basenpaare umfassenden miRNA-Doppelstrang geschnitten. In pflanzlichen Zellen hingegen erfolgt die Bildung eines miRNA-Doppelstrangs direkt aus der pri-miRNA unter Beteiligung der im Zellkern lokalisierten RNase DCL1.
Eine in tierischen Geschlechtszellen vorkommende Variante der RNA-Interferenz nutzt einzelsträngige piRNA (PIWI-interacting RNA). Die Bildung dieser etwa 24 bis 31 Nukleotide umfassenden interferierenden RNA-Moleküle unterscheidet sich wesentlich von der der siRNA und miRNA. An der noch nicht vollständig aufgeklärten Biogenese der piRNA sind weder doppelsträngige RNA-Vorläufermoleküle noch RNAsen vom Typ III beteiligt. Es wird unter anderem angenommen, dass die piRNA nach der Bildung eines Primärtranskripts durch Transkription eines piRNA-Clusters in einem als Ping-Pong-Mechanismus bezeichneten Kreislaufprozess unter Beteiligung von PIWI-Proteinen gebildet wird.[8]
Neben diesen interferierenden RNAs sind weitere zur RNA-Interferenz befähigte RNA-Typen, wie beispielsweise 21U-RNA[9] und die esiRNA[5], bekannt.
Bildung des RNA-induced silencing complex
Das zentrale Glied der RNA-Interferenz ist ein als RNA-induced silencing complex (RISC) bezeichneter Enzymkomplex. Die von Dicer gebildeten doppelsträngigen siRNA- oder miRNA-Moleküle werden an die Argonautenproteine des RNA-induced silencing complex übergeben. Dieser mit doppelsträngiger RNA beladene Komplex wird auch als Prä-RISC bezeichnet. Es werden innerhalb des Prä-RISC die Doppelstränge der gebundenen siRNA oder miRNA gespalten. Der als Leitstrang bezeichnete RNA-Einzelstrang verbleibt im RNA-induced silencing complex, der in diesem Zustand Holo-RISC genannt wird, während der andere Strang den Komplex verlässt und abgebaut wird. Schließlich wird eine zum Leitstrang komplementäre mRNA in den RNA-induced silencing complex eingebaut.
Spaltung der Ziel-RNA
Die am besten erforschte Konsequenz aus der Aktivierung des RNA-induced silencing complex ist die Spaltung einer im Komplex gebundenen und zum Leitstrang komplementären mRNA. Dieser Mechanismus, der insbesondere bei siRNA beobachtet werden kann, setzt ein Argonautenprotein mit Endonuklease-Aktivität und eine möglichst perfekte Komplementarität zwischen Leitstrang und Ziel-mRNA voraus.[10] Von den vier Argonautenproteinen des Menschen, die an der siRNA- oder miRNA-vermittelten RNA-Interferenz beteiligt sind, besitzt lediglich AGO2 eine Endonukleaseaktivität.[11] Eine Spaltung der Ziel-RNA kann darüber hinaus als eine Folge der piRNA-vermittelten RNA-Interferenz beobachtet werden. Diese Spaltung ist auf die Endonukleaseaktivität der im Ping-Pong-Zyklus beteiligten PIWI-Proteine zurückzuführen. Die gespaltene Ziel-mRNA kann in den P-Bodys weiter abgebaut werden.
Inhibition der Translation
Die wichtigste Funktion des RNA-induced silencing complex (insbesondere bei der miRNA-vermittelten RNA-Interferenz) ist die Hemmung der Übersetzung der Information einer mRNA in ein Protein an den Ribosomen (Translation). Für diese Funktion ist weder eine Endonukleaseaktivität noch eine hochgradige Komplementarität zwischen dem Leitstrang und der Ziel-mRNA Voraussetzung. Lediglich die Nukleotide 2 bis 7 des Leitstrangs müssen zu denen der Ziel-mRNA komplementär sein.[12] Die zugrundeliegenden molekularen Mechanismen der Translationshemmung sind weniger gut erforscht. Mindestens zwei mögliche Mechanismen, die in den Zellen der Taufliege Drosophila melanogaster bereits beobachtet werden konnten, werden für die Hemmung der Umsetzung der mRNA-Information in ein Protein verantwortlich gemacht. Zum einen kann der RNA-induced silencing complex über Protein-Protein-Wechselwirkungen mit Translationsinitialisierungsfaktoren blockieren. Zum anderen kann ein Abbau des Polyadenylierungssignals der mRNA durch den aktivierten RNA-induced silencing complex beobachtet werden. Beide Mechanismen haben eine Hemmung der Translation zur Folge.[13]
Inhibition der Transkription
Neben der RNA-Interferenz können kleine doppelsträngige RNA-Moleküle zu einer Hemmung der Transkription führen. An diesem im Zellkern stattfindenden sogenannten transkriptionellen Gen-Silencing ist eine als RNA-induced transcriptional silencing complex (RITS) bezeichnete Variante des RNA-induced silencing complex beteiligt. Der aktivierte RNA-induced transcriptional silencing complex führt über Histonmodifikationen zur Bildung von Heterochromatin-Bereichen des Erbguts. Diese Bereiche sind nicht mehr für die Enzyme der Transkription zugänglich.
RNA-Aktivierung
Ein weiteres, auf kleine RNA-Moleküle zurückzuführendes Phänomen neben der RNA-Interferenz ist die sogenannte RNA-Aktivierung (RNAa). Als zugrundeliegende Mechanismen werden eine Aktivierung der Transkription durch Promotor-spezifische interferierende RNA, eine Aktivierung der Translation unter Beteiligung von Argonautenproteinen, eine Wechselwirkung mit zelleigener miRNA und Antisense-Mechanismen diskutiert.[14][15][16]
Anwendung
Grundlagenforschung
Die RNA-Interferenz wird in der Grundlagenforschung zur Aufklärung der noch unbekannten Funktion eines zu untersuchenden bekannten Gens und dessen codierten Proteins genutzt. Die RNA-Interferenz erlaubt die gezielte Ausschaltung jedes beliebigen Gens. Aus dieser Ausschaltung kann die Funktion des von Gen codierten Proteins abgeleitet werden. Es muss einzig die Nukleotidsequenz des zu untersuchenden Gens bekannt sein, um potenziell interferierende RNA-Moleküle zu entwickeln. Damit ist die auch als „Gen-Knockdown“ bezeichnete Anwendung der RNA-Interferenz zur Untersuchung der Funktion eines Gens und dessen codierten Proteins deutlich weniger aufwendig als das konventionelle Gen-Knockout. Zudem ist die Anwendung der RNA-Interferenz zeit- und erfolgversprechender als die Entwicklung eines Liganden als Inhibitor der Proteinfunktion. Dies ist insbesondere der Fall, wenn eine pharmakologische Unterscheidung nahe verwandter Proteine praktisch unmöglich ist.[4]
Auch für die umgekehrte Fragestellung, die Suche nach den für eine bekannte Funktion oder ein bestimmtes Merkmal verantwortlichen Genen oder Proteinen, eignet sich die experimentelle Nutzung der RNA-Interferenz. Hierfür werden RNA-Interferenzbibliotheken mit interferierenden RNAs gegen jedes einzelne Gen eingesetzt, die mit Hilfe des Hochdurchsatz-Screenings genutzt werden. Diese sogenannten genomweiten RNA-Interferenzscreenings finden auch in der Pharmaforschung auf der Suche nach neuen Zielmolekülen für neue Wirkstoffe (Target discovery) Anwendung.
Für beide Anwendungsgebiete der RNA-Interferenz in der Grundlagenforschung werden insbesondere synthetische siRNA- oder shRNA-Moleküle (short hairpin RNA) eingesetzt. Sie werden entweder direkt mit Hilfe diverser Transfektionstechniken in die Zellen eingeschleust oder indirekt in den Zellen nach Einbringung eines Vektors, wie beispielsweise eines shRNA-codierenden Plasmids oder eines Virus, unter Ausnutzung der zellulären Transkription gebildet. Zur Vermeidung einer Fehlinterpretation durch die in der Praxis immer wieder auftretenden unspezifischen Effekte werden geeignete Kontrollexperimente durchgeführt oder die Spezifität wird mit Hilfe mehrerer interferierender RNAs gegen ein und dasselbe Gen bestätigt.[4]
Therapie
Obgleich die RNA-Interferenz ein erst kürzlich entdeckter biologischer Mechanismus ist, sind die ersten RNA-Interferenz-basierten Therapeutika bereits in den USA und auch der EU zugelassen. 2018 wurde mit Patisiran das erste RNAi-basierte Medikament zugelassen.[17][18] Patisiran zielt auf die TTR-mRNA mit dem Ziel einer Reduktion des Proteins TTR, das in der Leber gebildet wird. Bei Patienten mit hereditärer Transthyrethin-Amyloidose (hATTR) liegt eine Mutation im TTR-Gen vor. Infolgedessen werden fehlerhafte TTR-Proteine gebildet, die sich nicht korrekt in einer Tetramer-Struktur falten. Durch die fehlerhafte Faltung verklumpen die TTR-Proteine und lagern sich in Organen und Gewebe ab (Amyloid), was zu Funktionsstörungen im Organismus führt. Durch das Herunterfahren der TTR-Produktion kann die Progression der Erkrankung gestoppt werden. 2019 und 2020 folgten mit den Zulassungen von Givosiran zur Behandlung der Akuten intermittierenden Porphyrie[19][20] und Lumasiran zur Behandlung der Primären Hyperoxalurie Typ 1[21][22] die nächsten RNAi-Therapeutika zur Behandlung seltener, genetisch bedingter Erkrankungen. Mit Inclisiran wurde Ende 2020 zudem erstmals ein siRNA-basierter Wirkstoff zur Behandlung der familiären Hypercholesterinämie zugelassen. Am weitesten fortgeschritten in der Entwicklung von RNAi-basierten Therapeutika gilt das Unternehmen Alnylam Pharmaceuticals aus den USA. Alle bislang zugelassenen RNAi-Therapeutika stammen aus der Entwicklung von Alnylam. Inclisiran wurde auslizensiert an The Medicines Company, die 2020 von Novartis gekauft wurden.
Diese rasante Entwicklung kann zum einen auf das Potenzial der RNA-Interferenz als therapeutische Methode und andererseits auf die jahrelange Erfahrung mit Antisense-Oligonukleotiden und Ribozymen in der klinischen Entwicklung zurückgeführt werden.[4] Am weitesten fortgeschritten war die Entwicklung von Bevasiranib, einer gegen den Vascular Endothelial Growth Factor (VEGF) gerichteten siRNA, die zur Behandlung der altersbedingten Makuladegeneration eingesetzt werden sollte, jedoch in einer klinischen Studie der Phase III scheiterte. Kurz nach diesem Rückschlag stellten mehrere große pharmazeutische Unternehmen, darunter Hoffmann-La Roche, ihre auf siRNA basierenden Entwicklungsprogramme ein.[23] Ungeachtet dessen befinden sich weitere RNA-Interferenz-basierte Therapeutika, wie die gegen den VEGF-Rezeptor-1 gerichtete Sirna-027 und die gegen das Gen RTP801 gerichtete PF-655 zur Behandlung der altersbedingten Makuladegeneration sowie die gegen ein virales Nukleokapsid gerichtete ALN-RSV01 zur Behandlung von Respiratory-Syncytial-Virus-Infektionen, derzeit mindestens in Phase II der klinischen Erprobung.[24]
Auch zelleigene interferierende RNAs stellen potenzielle Ziele für die pharmazeutische Entwicklung dar. Sogenannte Antagomire, welche zelleigene miRNAs über Antisense-Mechanismen blockieren, befinden sich in der präklinischen Entwicklung.[25]
Geschichte der Entdeckung

Um das Jahr 1990 versuchte die Forschergruppe um Joseph Mol und Richard Jorgensen, die Blütenfärbung von Petunien zu verstärken.[26] Sie beabsichtigten, zusätzliche Kopien des Gens Dihydroflavonolreduktase in die Pflanzen einzubringen, und hofften dadurch die Produktion der Blütenfarbstoffe (aus der Gruppe der Flavonoide) anregen zu können. Das Gegenteil war jedoch der Fall. Zur Überraschung aller waren die meisten der genetisch veränderten Pflanzen weniger stark gefärbt als unbehandelte Pflanzen, einige sogar schneeweiß. Für dieses Phänomen wurde zunächst der Begriff „Cosuppression“ geprägt, da nicht nur die in die Pflanzen eingebrachten Gene, sondern auch das korrespondierende, natürlich in den Pflanzen vorkommende Gen der Dihydroflavonolreduktase kein bzw. nur wenig funktionelles Protein lieferte.
Weitere Arbeiten zeigten einige Jahre später, dass die Gene nicht nur auf der Ebene der Transkription abgeschaltet wurden, sondern dass zusätzlich die von ihnen produzierte mRNA in den Zellen schnell abgebaut wurde – ein Vorgang, der Post-Transcriptional Gene Silencing (PTGS) getauft wurde. Etwa zur gleichen Zeit wurden ähnliche Phänomene aus Pilzen (Neurospora crassa) unter dem Namen Quelling, aus Algen und aus dem Fadenwurm C. elegans beschrieben.

1998 schließlich war nach einer Reihe von Untersuchungen klar, dass die mRNA selbst maßgeblich an dem Phänomen des PTGS beteiligt ist. Fast gleichzeitig mit der vollständigen Sequenzierung des Genoms von C. elegans beschrieben Andrew Fire und Craig Mello 1998 die Technik der RNA-Interferenz (RNAi), bei der doppelsträngige RNA in C. elegans zu einem effizienten und spezifischen Gen-Knockdown führt.[27] Hierfür wurde ihnen der Nobelpreis für Physiologie oder Medizin des Jahres 2006 zuerkannt. Wie genau das Einbringen von doppelsträngiger RNA in den Organismus jedoch zum Abbau der Ziel-RNA führt, wurde erst klar, als 1999 Andrew J. Hamilton und David C. Baulcombe kurze RNA-Moleküle mit einer Länge von etwa 25 Nukleotiden isolieren konnten, die in direktem Zusammenhang zu der regulierten RNA steht: die siRNA, die der RNAi ihre Spezifität verleiht, indem sie die Ziel-RNA über Basenpaarung binden.[28]
Bei dem Versuch, die von Craig Mello und Andrew Fire verwendete Strategie auf Wirbeltiere zu übertragen, traten jedoch in der Folgezeit erhebliche Probleme auf: Die verwendeten Zellen schienen die langen doppelsträngigen RNAs nicht zu tolerieren, und es kam zum programmierten Zelltod (Apoptose). Erst 2001 veröffentlichten Sayda Elbashir und Thomas Tuschl einen Weg, wie dieses Problem umgangen werden kann. Sie verwendeten kurze doppelsträngige RNA mit je 21 Nukleotiden, die nicht zu einer Apoptose führen, aber funktionell für ein Gen-Silencing ausreichend sind.[29]
Es wurde postuliert, dass interferierende RNA bei der Abwehr von RNA-Viren und bei der Regulation einiger mobiler genetischer Elemente (Transposone) eine Rolle spielen könnten. Daher versuchte man – motiviert durch die Tatsache, dass die RNA-Interferenz einen in Eukaryoten wohl universellen Prozess darstellt – interferierende RNA aus unbehandelten Zellen zu isolieren. So stieß man auf eine weitere Gruppe von kleinen RNA-Molekülen, die miRNAs. Zwei dieser miRNAs, lin-4 und let-7, waren bereits zuvor in C. elegans entdeckt und zunächst als stRNAs (small temporal RNAs) bezeichnet worden.
Animationsvideos
- Gene zum Schweigen gebracht – der faszinierende Mechanismus der RNA-Interferenz Ein Animations-Film auf dem Video-Kanal des Verlages Spektrum der Wissenschaft, Veröffentlicht am 22. Juni 2012, Abgerufen am 27. Oktober 2016
- Weitere Animationsvideos zum Thema bei YouTube.
Literatur
- Jochen Graw: Genetik. Springer, Berlin; 5., vollständig überarbeitete Auflage 2010; ISBN 978-3-642-04998-9; S. 313ff.
- Jocelyn E. Krebs, Elliott S. Goldstein und Stephen T. Kilpatrick: Lewin's Genes X. Jones & Bartlett Pub (Ma); 10. Auflage 2010; ISBN 978-0-7637-6632-0; S. 861ff.
- Siomi H, Siomi MC: On the road to reading the RNA-interference code. In: Nature. 457, Nr. 7228, Januar 2009, S. 396–404. doi:10.1038/nature07754. PMID 19158785.
- Jinek M, Doudna JA: A three-dimensional view of the molecular machinery of RNA interference. In: Nature. 457, Nr. 7228, Januar 2009, S. 405–412. doi:10.1038/nature07755. PMID 19158786.
Weblinks
Einzelnachweise
- Cerutti H, Casas-Mollano JA: On the origin and functions of RNA-mediated silencing: from protists to man. In: Curr. Genet.. 50, Nr. 2, August 2006, S. 81–99. doi:10.1007/s00294-006-0078-x. PMID 16691418. PMC 2583075 (freier Volltext).
- Kamath RS, Ahringer J: Genome-wide RNAi screening in Caenorhabditis elegans. In: Methods. 30, Nr. 4, August 2003, S. 313–321. PMID 12828945.
- Haasnoot J, Westerhout EM, Berkhout B: RNA interference against viruses: strike and counterstrike. In: Nat. Biotechnol.. 25, Nr. 12, Dezember 2007, S. 1435–1443. doi:10.1038/nbt1369. PMID 18066040.
- Kurreck J: RNA interference: from basic research to therapeutic applications. In: Angew. Chem. Int. Ed. Engl.. 48, Nr. 8, 2009, S. 1378–1398. doi:10.1002/anie.200802092. PMID 19153977.
- Sontheimer EJ, Carthew RW: Silence from within: endogenous siRNAs and miRNAs. In: Cell. 122, Nr. 1, Juli 2005, S. 9–12. doi:10.1016/j.cell.2005.06.030. PMID 16009127.
- Siomi H, Siomi MC: On the road to reading the RNA-interference code. In: Nature. 457, Nr. 7228, Januar 2009, S. 396–404. doi:10.1038/nature07754. PMID 19158785.
- Jochen Graw: Genetik. Springer, Berlin; 5., vollständig überarbeitete Auflage 2010; ISBN 978-3-642-04998-9; S. 317.
- Aravin AA, Hannon GJ, Brennecke J: The Piwi-piRNA pathway provides an adaptive defense in the transposon arms race. In: Science. 318, Nr. 5851, November 2007, S. 761–764. doi:10.1126/science.1146484. PMID 17975059.
- Ruby JG, Jan C, Player C, et al.: Large-scale sequencing reveals 21U-RNAs and additional microRNAs and endogenous siRNAs in C. elegans. In: Cell. 127, Nr. 6, Dezember 2006, S. 1193–1207. doi:10.1016/j.cell.2006.10.040. PMID 17174894.
- Liu J, Carmell MA, Rivas FV, et al.: Argonaute2 is the catalytic engine of mammalian RNAi. In: Science. 305, Nr. 5689, September 2004, S. 1437–1441. doi:10.1126/science.1102513. PMID 15284456.
- Pratt AJ, MacRae IJ: The RNA-induced silencing complex: a versatile gene-silencing machine. In: J. Biol. Chem.. 284, Nr. 27, Juli 2009, S. 17897–178901. doi:10.1074/jbc.R900012200. PMID 19342379. PMC 2709356 (freier Volltext).
- Lewis BP, Burge CB, Bartel DP: Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microRNA targets. In: Cell. 120, Nr. 1, Januar 2005, S. 15–20. doi:10.1016/j.cell.2004.12.035. PMID 15652477.
- Iwasaki S, Kawamata T, Tomari Y: Drosophila argonaute1 and argonaute2 employ distinct mechanisms for translational repression. In: Mol. Cell. 34, Nr. 1, April 2009, S. 58–67. doi:10.1016/j.molcel.2009.02.010. PMID 19268617.
- Li LC, Okino ST, Zhao H, et al.: Small dsRNAs induce transcriptional activation in human cells. In: Proc. Natl. Acad. Sci. U.S.A.. 103, Nr. 46, November 2006, S. 17337–42. doi:10.1073/pnas.0607015103. PMID 17085592. PMC 1859931 (freier Volltext).
- Vasudevan S, Tong Y, Steitz JA: Switching from repression to activation: microRNAs can up-regulate translation. In: Science. 318, Nr. 5858, Dezember 2007, S. 1931–4. doi:10.1126/science.1149460. PMID 18048652.
- Krützfeldt J, Rajewsky N, Braich R, et al.: Silencing of microRNAs in vivo with 'antagomirs'. In: Nature. 438, Nr. 7068, Dezember 2005, S. 685–689. doi:10.1038/nature04303.
- FDA approves first-of-its kind targeted RNA-based therapy to treat a rare disease, PM FDA vom 10. August 2018, abgerufen am 30. März 2021
- EMA: Onpattro. European Medicines Agency, abgerufen am 30. März 2021 (englisch).
- FDA approves first treatment for inherited rare disease. In: FDA. 20. November 2019, abgerufen am 30. März 2021.
- K. Graefe: Erstes Medikament für seltene Erbkrankheit, Pharmazeutische Zeitung, vom 10. März 2020, abgerufen am 30. März 2021
- FDA Approves First Drug to Treat Rare Metabolic Disorder, PM FDA vom 23. November 2020, abgerufen am 31. März 2021
- Oxlumo, Website der EMA, abgerufen am 31. März 2021
- Ledford H: Drug giants turn their backs on RNA interference. In: Nature. 468, Nr. 7323, November 2010, S. 487. doi:10.1038/468487a. PMID 21107398.
- Tiemann K, Rossi JJ: RNAi-based therapeutics-current status, challenges and prospects. In: EMBO Mol Med. 1, Nr. 3, Juni 2009, S. 142–151. doi:10.1002/emmm.200900023. PMID 20049714.
- Alderton GK: Therapy: A big step for targeting RNAs. In: Nature Reviews Cancer. 10, 2010, S. 313. doi:10.1038/nrc2848.
- Napoli C, Lemieux C, Jorgensen R: Introduction of a chalcone synthase gene into Petunia results in reversible co-suppression of homologous genes in trans. In: Plant Cell. 2, Nr. 4, 1990, S. 279–289. PMID 12354959. PMC 159885 (freier Volltext).
- Fire A., Xu S., Montgomery M.K., Kostas S.A., Driver S.E., Mello C.C.: Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. In: Nature. 391, 1998, S. 806–811. PMID 9486653.
- Hamilton A.J., Baulcombe D.C.: A species of small antisense RNA in posttranscriptional gene silencing in plants. In: Science. 286, Nr. 5441, 1999, S. 950–952. PMID 10542148.
- Elbashir S.M., Harborth J., Lendeckel W., Yalcin A., Weber K., Tuschl T.: Duplexes of 21-nucleotide RNAs mediate RNA interference in cultured mammalian cells. In: Nature. 411, 2001, S. 494–498. PMID 11373684.