Cas9
Cas9 (von englisch CRISPR-associated, veraltet auch Cas5, Csn1 oder Csx12) ist eine Endonuklease und ein Ribonukleoprotein aus Bakterien.
| Cas9 | ||
|---|---|---|
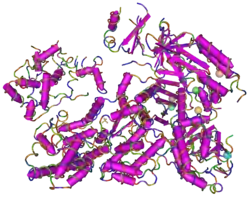 | ||
| CRISPR-associated protein Cas9 von Streptococcus pyogenes | ||
| Andere Namen |
SpyCas9, Cas5, Csn1, Csx12 | |
| Masse/Länge Primärstruktur | 1.368 Aminosäuren, 158.441 Da | |
| Bezeichner | ||
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 3.1.-.- | |
Eigenschaften
Das CRISPR/Cas-System entstammt einem adaptiven antiviralen Abwehrmechanismus aus Bakterien, dem CRISPR.[1][2] Die Endonuklease Cas9 kann eine bestimmte RNA-Sequenz (crRNA repeat, Sequenz GUUUUAGAGCU(A/G)UG(C/U)UGUUUUG)[3] binden und DNA schneiden. Diese crRNA repeat-Sequenz bildet eine RNA-Sekundärstruktur, bindet eine tracrRNA und wird dann von Cas9 gebunden.[4] An der crRNA repeat-Sequenz befindet sich anschließend eine an die Ziel-DNA bindende Sequenz (crRNA spacer), beide Sequenzen werden zusammen als crRNA bezeichnet. Als zweiter Teil dient die crRNA spacer-Sequenz in der Funktion eines variablen Adapters, welche komplementär zur Ziel-DNA ist und an die Ziel-DNA bindet. Dadurch wird die DNA nahe der Bindungsstelle geschnitten. Zur Hemmung von Cas-Proteinen werden von Bakteriophagen Anti-CRISPR-Proteine gebildet.
Struktur
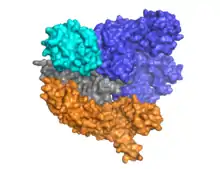
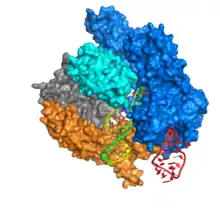
Cas9 besteht aus zwei Bereichen, mit der RNA eingebettet zwischen dem α-helikalen Bereich (blau) und dem Bereich der Endonuklease (cyan, orange, grau). Die beiden Bereiche sind über eine Helix verbunden. Der Nukleasebereich enthält zwei Endonuklease-Proteindomänen, RuvC (grau) und die HNH-Nuclease (cyan). Die HNH-Nuklease schneidet den an die crRNA gebundenen DNA-Strang, während die RuvC den gegenüberliegenden Strang schneidet. Die zu schneidende DNA muss das Protospacer adjacent Motif (PAM) enthalten, das aus den drei Nukleotiden NGG besteht, und von der PAM-interacting domain am C-Terminus von Cas9 (PI-Domäne, orange) gebunden wird. Cas9 besitzt drei verschiedene Konformationen, ungebunden, an RNA gebunden und an RNA und DNA gebunden.
Cas9 bindet die stem-loop-Sekundärstruktur der crRNA.[5] Der Komplex aus Cas9, crRNA und tracrRNA bindet an die Ziel-DNA und schneidet beide DNA-Stränge.[6] Die beiden RNA können auch in einem einzigen, abschnittsweise selbsthybridisierenden Strang untergebracht werden (single-guide RNA, sgRNA).[7] Die DNA-Bindungsdomäne (recognition domain, REC) die Endonukleasedomäne (NUC) sind räumlich getrennt. Da die HNH-Domäne flexibel mit dem restlichen Protein verbunden ist, ist sie im Cas9-Proteinkristall nicht erkennbar. Cas9 verwendet Manganionen als Cofaktoren.[8]
Typen
Es existieren mehr als 40 verschiedene Cas-Proteinfamilien.[9] Die Familien können in drei Typen eingeteilt werden.[10] Typ I, II und IIIa binden und schneiden doppelsträngige DNA, während Typ IIIb einzelsträngige RNA bindet und schneidet.[11][10] Bei allen Typen erfolgt die Bildung des spacers in Bakterien durch Cas1 und Cas2.[11] Bei den Typen I-A und I-E erfolgt der DNA-Schnitt durch Cas3, während bei Typ II Cas9, bei Typ III-A Csm6 und bei Typ III-B Cmr4 den Schnitt bewirkt.[11] Die Typen I und III sind strukturell verwandt, was einen gemeinsamen Ursprung nahelegt.[10] Das helikale Protein Cas des Typs III besitzt mehrere β-hairpins, die in Abständen von sechs Nukleotiden die Doppelhelix der crRNA und der Ziel-RNA für den Schnitt auseinanderdrücken.[10]
Anwendungen
Cas9 wird als Bestandteil des CRISPR/Cas-Systems zum Genome Editing verwendet.
Einzelnachweise
- R. Sorek, V. Kunin, P. Hugenholtz: CRISPR–a widespread system that provides acquired resistance against phages in bacteria and archaea. In: Nature reviews. Microbiology. Band 6, Nummer 3, März 2008, S. 181–186, ISSN 1740-1534. doi:10.1038/nrmicro1793. PMID 18157154.
- D. Rath, L. Amlinger, A. Rath, M. Lundgren: The CRISPR-Cas immune system: biology, mechanisms and applications. In: Biochimie. Band 117, Oktober 2015, S. 119–128, doi:10.1016/j.biochi.2015.03.025, PMID 25868999 (Review).
- G. Gasiunas, R. Barrangou, P. Horvath, V. Siksnys: Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. In: Proceedings of the National Academy of Sciences. Band 109, Nummer 39, September 2012, S. E2579–E2586, ISSN 1091-6490. doi:10.1073/pnas.1208507109. PMID 22949671. PMC 3465414 (freier Volltext).
- V. Kunin, R. Sorek, P. Hugenholtz: Evolutionary conservation of sequence and secondary structures in CRISPR repeats. In: Genome biology. Band 8, Nummer 4, 2007, S. R61, ISSN 1465-6914. doi:10.1186/gb-2007-8-4-r61. PMID 17442114. PMC 1896005 (freier Volltext).
- Wiedenheft B, Sternberg SH, Doudna JA: RNA-guided genetic silencing systems in bacteria and archaea. In: Nature. 482, Nr. 7385, Feb 2012, S. 331–8. bibcode:2012Natur.482..331W. doi:10.1038/nature10886. PMID 22337052.
- Ran FA, Hsu PD, Wright J, Agarwala V, Scott DA, Zhang F: Genome engineering using the CRISPR-Cas9 system. In: Nat Protoc. 8, Nr. 11, 2013, S. 2281–308. doi:10.1038/nprot.2013.143. PMID 24157548. PMC 3969860 (freier Volltext).
- Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E: A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. In: Science. 337, Nr. 6096, 2012, S. 816–21. bibcode:2012Sci...337..816J. doi:10.1126/science.1225829. PMID 22745249.
- M. Jinek, F. Jiang, D. W. Taylor, S. H. Sternberg, E. Kaya, E. Ma, C. Anders, M. Hauer, K. Zhou, S. Lin, M. Kaplan, A. T. Iavarone, E. Charpentier, E. Nogales, J. A. Doudna: Structures of Cas9 endonucleases reveal RNA-mediated conformational activation. In: Science. Band 343, Nummer 6176, März 2014, S. 1247997, doi:10.1126/science.1247997, PMID 24505130, PMC 4184034 (freier Volltext).
- D. H. Haft, J. Selengut, E. F. Mongodin, K. E. Nelson: A guild of 45 CRISPR-associated (Cas) protein families and multiple CRISPR/Cas subtypes exist in prokaryotic genomes. In: PLoS computational biology. Band 1, Nummer 6, November 2005, S. e60, ISSN 1553-7358. doi:10.1371/journal.pcbi.0010060. PMID 16292354. PMC 1282333 (freier Volltext).
- D. W. Taylor, Y. Zhu, R. H. J. Staals, J. E. Kornfeld, A. Shinkai, J. van der Oost, E. Nogales, J. A. Doudna: Structures of the CRISPR-Cmr complex reveal mode of RNA target positioning. In: Science. 348, 2015, S. 581, doi:10.1126/science.aaa4535.
- J. van der Oost, E. R. Westra, R. N. Jackson, B. Wiedenheft: Unravelling the structural and mechanistic basis of CRISPR-Cas systems. In: Nature reviews. Microbiology. Band 12, Nummer 7, Juli 2014, S. 479–492, doi:10.1038/nrmicro3279, PMID 24909109, PMC 4225775 (freier Volltext) (Review).