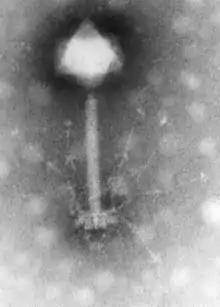
Cyanophagen
Cyanophagen sind eine nicht-taxonomische Gruppe von Viren, die Cyanobakterien (auch Blaugrünbakterien, veraltet Cyanophyten oder Blaualgen) infizieren. Sie werden daher als Bakteriophagen klassifiziert.
Cyanobakterien sind ein Phylum von Bakterien, die ihre Energie durch den Prozess der Photosynthese gewinnen.[1][2] Obwohl Cyanobakterien wie eukaryotische Pflanzen einen photoautotrophen Stoffwechsel haben, besitzen sie als echte Bakterien eine prokaryotische Zellstruktur. Cyanophagen kommen sowohl im Süß- als auch im Salzwasser (Meerwasser) vor.[3]
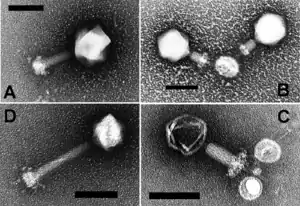
Alle bekannten Cyanophagen gehören zur Virusklasse der Caudoviricetes (Viren mit Kopf-Schwanz-Aufbau). Sie haben einen ikosaedrischen Kopf (Kapsid), der das virale Genom in Form doppelsträngiger DNA (dsDNA) enthält, und an dem der Schwanz über Verbindungsproteine befestigt ist. Die Größe des Kopfes und des Schwanzes variiert zwischen den verschiedenen Arten (Spezies) von Cyanophagen.[4]
Die herkömmliche Klassifizierung teilt die Viren mit Kopf-Schwanz-Aufbau (die heutige Klasse Caudoviricetes) ein nach morphologischen Kriterien (Schwanzlänge, Kontraktibilität) in Myoviren, Podoviren und Siphoviren, herkömmlich als Familien definiert. Entsprechend dem Vorschlag, dieses Schema für de Cyanophagen zu übernehmen, wurden die informellen Gattungen „Cyanomyovirus“, „Cyanopodovirus“ respektive „Cyanostylovirus“ (für die Siphoviren unter den Cyanophagen). Die heutige Virus-Taxonomie gründet sich aber immer mehr auf Genom-Sequenzierung statt auf morphologische Kriterien, weil diese die Verwandtschaftsbeziehungen besser wiedergeben und andernfalls Ergebnisse der Metagenomik gar nicht eingeordnet werden können. In der Konsequenz hat das International Committee on Taxonomy of Viruses (ICTV) bereits mehrere Familien von den obigen drei klassischen morphologie-begründeten Familien abgespalten. Im März 2021 wurde vorgeschlagen, diesen Prozess so weit fortzusetzen, dass die drei klassischen Familien der Schwanzviren als Taxa völlig aufgelöst werden und nur noch als nicht-taxonomischen morphologische Klassifizierungen verbleiben.[5]
Forschungsgeschichte
Cyanophagen wurden erstmals von Safferman und Morris im Jahr 1963 beschrieben.[6][1][7][8] Der von Safferman und Morris damals isolierte Cyanophage stammte aus einem Abwasserteich (en. waste stabilization pond) der Indiana University (USA), und war in der Lage, drei Gattungen von Cyanobakterien zu infizieren: Lyngbya, Plectonema und Phormidium. Der Phage wurde daher nach deren Anfangsbuchstaben mit dem Akronym LPP-1 bezeichnet.[9] In der Folge wurden weitere Serotypen von Cyanophagen mit diesem Wirtsspektrum gefunden, LPP-2, LPP-3, LPP-4 und LPP-5.[10][7][8] LPP-1 infiziert dabei speziell die Spezies Plectonema boryanum.[11][12][13]
Die Morphologie der Phagen der LPP-Gruppe zeigte weitgehende Übereinstimmung mit den bekannten Phagen T3 und T7. T3 und T7 waren ursprünglich in der Familie der Podoviren (d. h. in der informellen Gattung „Cyanopodovirus“) angesiedelt. Nach Abspaltung der neuen Familie Autographiviridae wurden diese beiden Vertreter der T-Phagen mit ungerader Nummer (als nicht-taxonomische Gruppe auch T-odd/T-uneven phages genannt) in deren Unterfamilie Studiervirinae (Gattungen Teetrevirus respektive Teseptimavirus) verschoben. Viele weitere Cyanopodovirus-Spezies sind inzwischen vom ICTV in die Familie Autographiviridae (und dort meist in die Gattung Teseptimavirus) verschoben worden; aufgrund ihrer Ähnlichkeit mit T3 und T7 sind die LPP-Mitglieder daher ebenfalls in der Unterfamilie Studiervirinae zu vermuten. Insgesamt dürfte die informelle Gattung „Cyanopodovirus“ heute eher in dieser Familie angesiedelt sein als noch bei den Podoviridae. Weitere T7 und dem in den USA gefundenen LPP-1 morphologisch ähnliche Cyanophagen sind GIII (gefunden in Israel) und D-1 (gefunden in Schottland).[14][15]
Mit und nach den Funden der Cyanopodoviren (bzw. aus der LPP-Gruppe) wurden weitere Cyanophagen mit abweichender Morphologie gefunden.[9] „Cyanophage AS-1“[16] und „Cyanobacteria phage N1“ (alias „Anabaena phage N-1“)[17] sind vorgeschlagene Mitglieder der Myoviridae (also der informellen Gattung „Cyanomyovirus“). Die N-1-Cyanophagen ähneln den Phagen T2 und T4 (beide Spezies Escherichia-Virus T4, Unterfamilie Tevenvirinae dieser Familie).[7]
Weitere Cyanophagen wie „Cyanophage S-1“[10][18] und „Cyanophage S-2L“ (alias „Cyanobacteria phage S-2L“ – Wirte aus den Gattungen Synechococcus[19] und Synechocystis,[20] siehe auch 2,6-Diaminopurin)[21][22][23][24][25] (Zusammenfassungen auf sciencealert und ScienceNews).[26][27] werden den Siphoviridae (und damit der informellen Gattung „Cyanostylovirus“) zugeordnet. Auch hier gibt es einen Vorschlag zur Abtrennung und Verschiebung in eine eigene Familie „Cyanostyloviridae“ oder „Styloviridae“.[28][1][29][30][31]
Während die Cyanophagen der Klasse Caudoviricetes (mit Kopf-Schwanz-Aufbau) relativ gut erforscht sind, gilt das nicht für schwanzlose Cyanophagen, zum Beispiel filamentöse (als mögliche Ordnung käme etwa Tubulavirales in Frage). Über die ersten filamentösen Cyanophagen berichteten Deng und Hayes 2008 mit „Filamentous phage A-CF1“, „Filamentous phage M-CF1“, „Filamentous phage (P-CF1)“. mit Wirten unter den Gattungen Anabaena, Microcystis und Planktothrix (Fundort Cotswold Water Park, UK; siehe Liste der Schutzgebiete in South West England).[32][33][Anm. 1]
Es folgten E-Bin Gao und Kollegen 2009 mit dem schwanzlosen Cyanophagen „Planktothrix-Phage PaV-LD“ (PaV-LD: „Planktothrix agardhii Virus isolated from Lake Donghu“) mit ikosaedrischer, schwanzloser Struktur und Wirt Planktothrix,[34][33] nach NCBI aber dennoch zu den Siphoviridae gestellt.[35]
Cyanophagen infizieren eine Vielzahl von Cyanobakterien und sind wichtige Regulatoren der Cyanobakterienpopulationen in aquatischen Umgebungen. Sie können bei der Prävention und Bekämpfung von Cyanobakterienblüten („Blaualgenblüten“) in Süßwasser- und Meeresökosystemen helfen. Diese Blüten können eine Gefahr für Mensch und Tier darstellen, insbesondere in eutrophierten Süßwasserseen. Infektionen mit diesen Viren ist in Cyanobakterienspezies der Gattung Synechococcus in marinen Umgebungen weit verbreitet. Man hat festgestellt, dass bis zu 5 % der Zellen mariner Cyanobakterien reife Cyanophagenpartikel enthalten.[36]
Nomenklatur
Ursprünglich wurden die folgenden drei Familien mit Cyanophagen-Mitgliedern vom International Committee on Taxonomy of Viruses (ICTV) anerkannt: Myoviridae, Podoviridae und Siphoviridae.[37] Ursprünglich wurden Cyanophagen nach ihren Wirten benannt. Wegen der Fähigkeit von Cyanophagen, mehrere Wirte zu infizieren, kann das Fehlen eines universellen Benennungssystems zu Schwierigkeiten bei ihrer taxonomischen Klassifizierung führen.[30] Viele andere Klassifizierungssysteme verwendeten serologische, morphologische oder physiologische Eigenschaften.[38][39] Zur Lösung wurde das folgende Verfahren zur Benennung von Stämmen vorgeschlagen: Cyanophage Xx-YYZaa, wobei Xx die ersten beiden Buchstaben des Gattungs- und Artnamens des Wirts sind, YY die Herkunft des Exemplars kodiert, Z ist der Anfangsbuchstabe der (herkömmlichen) Virusfamilie — der Morphologie-Grundtyp also: M, P oder S, und aa ist die Referenznummer des Virus.[3] Diese Bezeichnungen haben solange Vorschlagscharakter, bis eine endgültige Bestätigung und taxonomische Einordnung durch das ICTV erfolgt.
Morphologie

von links nach rechts.
Wie alle anderen schwanzförmigen Bakteriophagen der Klasse Caudoviricetes haben Cyanophagen einen Schwanz und ein Proteinkapsid, das das genetische Material umgibt. Diese doppelsträngige DNA ist ca. 45 kbp (Kilobasenpaare) lang und kodiert bei einigen Cyanophagen für Photosynthesegene, eine Integrase, oder Gene, die mit dem Phosphatstoffwechsel zu tun haben (en. phosphate-inducible).[40] Über den Schwanz nehmen die Viruspartikel (Virionen) Kontakt mit der Wirtszelle auf und durch ihn wird bei der Infektion die Virus-DNA in die Wirtszelle injiziert. Basierend auf morphologischen Merkmalen werden Cyanophagen in Myoviren, Podoviren und Siphoviren (ursprünglich Virusfamilien) klassifiziert. Obwohl vom Internationalen Komitee für Taxonomie der Viren (ICTV) nicht formell anerkannt, wurden Cyanophagen dem entsprechend provisorisch in die informellen Gattungen „Cyanomyovirus“, „Cyanopodovirus“ bzw. „Cyanostylovirus“ klassifiziert, je nachdem, welchem der drei morphologischen Grundtypen sie angehören.[30]
„Cyanomyovirus“
Die Typusart der informellen Gattung „Cyanomyovirus“ (Cyanophagen mit myovirenartiger Morphologie, d. h. Mitglieder der Familie Myoviridae und ihrer Abspaltungen) ist der Cyanophage AS-1,[16] der aus einem Abwasserteich (en. waste stabilization pond) isoliert wurde,[41] und auch die erste offiziell anerkannte Spezies war.[42] Ihr Kopf ist ikosaedrisch (im Allgemeinen ideal-isometrische, nicht gestreckt) mit einem Durchmessern von 55 bis 90 nm, der Schwanz kann kontraktil oder nicht kontraktil sein mit einer Länge von 20 bis 244 nm und Breite von 15 bis 23 nm (üblicherweise also etwa 110 × 10 nm); sowie ggf. einem Schrumpfungsbereich von 93 nm.[7][9][3] Es gibt jedoch eine große morphologische Variation in dieser Gruppe, was möglicherweise damit zusammenhängt, dass die verschiedenen Vertreter ein großes Wirtsspektrum abdecken.[43] An der Verbindungsstelle zwischen dem langen Schwanz und dem Kopf befindet sich eine Grundplatte, an der kurze Stifte befestigt sind, und im Kopfteil einen inneren Kern, ähnlich wie bei anderen Myoviren.[41]
Einige weitere Cyanomyoviren sind:
- Spezies Synechococcus-Virus SPM2 (en. Synechococcus virus SPM2, Typus), mit Synechococcus-Phage S-PM2 alias Bacteriophage S-PM, Typusstamm[44][45]
- Spezies Synechococcus-Virus Syn5[46] mit Synechococcus-Phage syn5 alias Cyanophage Syn5 – wurde vom ICTV bestätigt und der offiziellen Gattung Voetvirus (Autographiviridae) zugewiesen.
- Spezies „Synechococcus-Phage Syn9“ (alias Bacteriophage Syn9, Cyanophage Syn9)[47][48]
- Spezies Synechococcus-Virus SSM1[49] mit Synechococcus-Phage S-SM1 alias Cyanophage S-SM1 – wurde vom ICTV bestätigt und der offiziellen Gattung Thetisvirus (Myoviridae) zugewiesen.
- Species Prochlorococcus-Virus PSSM2[50] mit Prochlorococcus-Phage P-SSM2 – wurde vom ICTV bestätigt und der offiziellen Gattung Salacisavirus (Myoviridae) zugewiesen.
- Spezies „Prochlorococcus-Phage P-SSM4“ (mit Cyanophage P-SSM4)[51][45]
- Spezies „Synechococcus-Phage S-RSM2“ (mit Bacteriophage S-RSM2)[52][45]
- Spezies „Synechococcus-Phage S-BM4“ (mit Cyanophage S-BM4 alias Bacteriophage S-BM4)[53][45]
- Spezies „Synechococcus-Phage S-WHM1“ (mit Cyanophage S-WHM1)[54][45]
- Spezies „Synechococcus-Phage S-RSM88“ (mit Cyanophage S-RSM88 alias Bacteriophage S-RSM88)[55][45]
- Spezies „Prochlorococcus-Virus 4f“, nach NCBI der Gattung Teseptimavirus (Autographiviridae) zugeordnet.[56]
- Spezies „Prochlorococcus-Virus 5e“, dito
- Spezies „Prochlorococcus-Virus 5f“, dito
- Spezies „Prochlorococcus-Virus 6b“, dito
- Spezies „Prochlorococcus-Virus 6ed6p“, dito
- Spezies „Prochlorococcus-Virus 7g“, dito
- Spezies „Prochlorococcus-Virus d67f2“, dito
- Spezies „Synechococcus-Virus 11bc6“, dito
- Spezies „Synechococcus-Virus 11ec6“, dito
- Spezies „Synechococcus-Virus 2fc6“, dito
- Spezies „Synechococcus-Virus 4dc“, dito
- Spezies „Synechococcus-Virus 5gcp“, dito
- Spezies „Synechococcus-Virus 6bc6“, dito
- Spezies „Synechococcus-Virus 7dc6“, dito
- Spezies „Synechococcus-Virus 9ec6“, dito
- Spezies „Synechococcus-Virus 9ecp“, dito
- Spezies „Synechococcus-Virus c7e4“, dito
- Spezies „Synechococcus-Phage S-SBP1“ (alias „Cyanopodovirus S-SBP1“, Wirt Synechococcus-Stamm WH7803)[57][58]
- Spezies „Synechococcus-Phage S-RIP2“ (alias „Cyanophage S-RIP2“, veraltet „Cyanopodovirus S-RIP2“)[57][59]
- Spezies „Synechococcus podovirus MPP-A“[60]
- Spezies „Synechococcus podovirus MPP-B“[60]
- Spezies Prochlorococcus-Virus PSSP7 (alias Prochlorococcus-Phage P-SSP7, Cyanopodovirus P-SSP7)[61][57] – wurde vom ICTV bestätigt und der offiziellen Gattung Tiamatvirus (Autographiviridae) zugewiesen.
- Spezies Synechococcus-Virus P60 (alias Synechococcus-Phage P60)[62] – wurde vom ICTV bestätigt und der offiziellen Gattung Tiilvirus (Autographiviridae) zugewiesen.
„Cyanopodovirus“
Die informelle Gattung „Cyanopodovirus“ (Cyanophagen mit podovirenartiger Morphologie, d. h. Mitglieder der Familie Podoviridae und ihrer Abspaltungen wie der Autographiviridae), kommen sowohl im Süß- als auch im Meerwasser vor.[63] Die Typusspezies ist der „Cyanophage LPP-1“, der Cyanobakterien der Gattungen Lyngbya, Plectonema und Phormidium infiziert.[64] Auch hier ist der Kopf (Kapsid) ein isometrisches Ikosaeder (was in der zweidimensionalen Projektion hexagonal, d. h. sechseckig erscheint) mit einem Durchmesser von 58 nm. Der Schwanz ist hohl mit sechsfach-radialer Symmetrie, und besteht aus Ringen von sechs Untereinheiten, ist aber im Vergleich zu den Cyanomyoviren kurz (20 × 15 nm).[9][3][7]
Einige weitere Cyanopodoviren sind:
„Cyanostylovirus“
Die Typusspezies der informellen Gattung „Cyanostylovirus“ (Cyanophagen mit siphovirenartiger Morphologie, d. h. Mitglieder der Familie Siphoviridae bzw. ihrer Abspaltungen – etwa der vorgeschlagenen „Cyanostyloviridae“/„Styloviridae“) ist der „Cyanophage S-1“ ist, von dem bekannt ist, dass er die Gattung Synechococcus infiziert.[3] Der Kopf ist mit einem Durchmesser von 50 nm kleiner als bei den anderen beiden Gruppen, der Schwanz ist jedoch mit 140 nm länger.[69] Einige Vertreter dieser Gruppe haben Schwänze, die zwischen 200 und 300 nm lang sind.[43]
Einige Cyanostyloviren sind:
Wirte
Das Wirtsspektrum der Cyanophagen ist sehr komplex. Es wird angenommen, dass sie eine wichtige Rolle bei der Kontrolle von Cyanobakterien-Populationen spielen.[1] Es wurde berichtet, dass Süßwasser-Cyanophagen Wirte verschiedener Gattungen infizieren. Dies könnte aber auch Probleme bei der taxonomischen Einordnung ihrer Wirte widerspiegeln, da Bakteriophagen der Klasse Caudoviricetes gewöhnlich sehr wirtsspezifisch sind. Nichtsdestotrotz wurden sie basierend auf der Taxonomie ihrer Wirtsorganismen in folgende drei Hauptgruppen klassifiziert:[1][3]
LPP-Gruppe
Die LPP-Gruppe ist die erste, die zu den Cyanopodoviren gehört.[1] Zu dieser Gruppe von Viren gehört das erste Cyanophagen-Isolat überhaupt, das Cyanobakterien, damals „Blaualgen“ (en. blue-green algae) genannt, infiziert.[42][3] Die Cyanophagen dieser Gruppe sind leicht aus der Umwelt zu isolieren.[3] Als Podoviren tragen sie kurze, nicht kontraktile Schwänze und verursachen die Lyse mehrerer Spezies innerhalb dreier Gattungen von Cyanobakterien (Lyngbya, Plectonema und Phormidium).[3] Aus den Anfangsbuchstaben dieser drei Gattungen wurde der Name LPP abgeleitet.[73] Die verschiedenen Mitglieder dieser Gruppe von Cyanophagen haben das gleiche Wirtsspektrum, zeigen jedoch serologische Unterschiede.[73]
AS/SM-Gruppe
Die AS/SM-Gruppe stellt die dritte Gruppe von Cyanophagen dar, die nach dem Wirtsspektrum klassifiziert wurden.[1] Diese damals neue Gruppe von Cyanophagen („Blaualgenviren“) infiziert einzellige Formen von Cyanobakterien.[3][74][41] „Cyanophage AS-1“ (alias „Myovirus AS-1“)[16] infiziert Anacystis nidulans,[75] Synechococcus cedrorum, Synechococcus elongatus und die Netzblaualge Microcystis aeruginosa[76][77] „Cyanophage S-SM1“[78] (alias „Podovirus SM-1“) infiziert ebenfalls die einzelligen Cyanobakterien Synechococcus elongatus und Microcystis aeruginosa.[3][79][9] Ein weiterer Vertreter dieser Gruppe, „Cyanophage S-SM2“[80] (alias „Podovirus SM-2“), infiziert und lysiert neben Synechococcus elongatus ebenfalls Microcystis aeruginosa.[79]
A/AN/N/NP-Gruppe
Dieses ist die zweite Gruppe von Cyanophagen dar, die nach ihrem Wirtsspektrum klassifiziert wurden.[69][1][81][82] Die Mitglieder dieser Gruppe spielen eine wichtige Rolle bei der Infektion und Lyse von Spezies der Gattungen Nostoc, Anabaena und Plectonema.[1] Die Untergruppe A verursacht Lyse und infiziert Anabaena-Spezies.[3] Der Wirtsbereich der AN-Untergruppe umfasst sowohl Anabaena- als auch Nostoc-Arten, während die N-Untergruppe nur Nostoc-Arten infiziert. Zu dieser letzten Untergruppe gehört „Cyanobacteria phage N1“ (alias „Cyanophage N-1“).[3] N-1 ist eng mit dem „Cyanophagen A-1“ verwandt,[83] und NCBI fasst beide in einer gemeinsamen Spezies „Anabaena phage N-1“ zusammen.[17] „Cyanophage N-1“ ist insofern bemerkenswert, als er für ein funktionelles CRISPR-Array kodiert, das dem Wirt möglicherweise Immunität gegen die Infektion durch konkurrierende Cyanophagen verleiht.[83] Schließlich werden Cyanobakterien-Isolate von Nostoc- und Plectonema-Arten von der NP-Untergruppe infiziert.[3] Die Vertreter dieser Gruppe haben alle einen breiten Wirtsbereich. Auffällig sind auch die vielen Mutationen bei diesen Viren.[3]
Replikation
Die Replikation von Cyanophagen hat zwei dominante Pfade: den lytischen Zyklus und den lysogenen Zyklus. Die Replikation der viralen Nukleinsäure (Virus-DNA) und die unmittelbare Synthese des viruskodierten Proteins ist Teil des lytischen Zyklus. Im Gegensatz den lytischen Phagen, die nur in diesen lytischen Zyklus eintreten können, können temperierte („gemäßigte“) Phagen entweder in den lytischen Zyklus eintreten oder sich stabil in das Wirtsgenom integrieren und damit in den lysogenen Zyklus eintreten.[84] Um den metabolischen Bedarf der Replikation zu decken, setzen Viren eine Vielzahl von Strategien ein, um ihrem Wirt Nährstoffe zu entziehen. Eine dieser Techniken besteht bei Cyanophagen darin, ihre Wirtszelle auszuhungern. Dies geschieht durch Hemmung der CO2-Fixierung der Wirtszelle, was es dn Phagen ermöglicht, photosynthetisch gebildetes ATP aus der Wirtszelle zu rekrutieren, um seine Nukleotid- und Stoffwechselanforderungen zu erfüllen.[85] Viele Cyanophagen enthalten Hilfsgene, die als auxiliary metabolic genes (AMGs) bezeichnet werden und die für kritische, Schritte des Wirtsorganismus (Flaschenhälse) kodieren.[85]
AMGs kodieren Gene für den Pentosephosphatweg, die Phosphatakquisition,[86] den Schwefelstoffwechsel und die DNA/RNA-Verarbeitung; all diese Gene greifen in den Stoffwechsel der Wirtszelle ein. Metagenomanalysen stützen die Annahme, dass diese Gene die virale Replikation durch den Abbau von Wirts-DNA und -RNA sowie einer Verlagerung des Wirtszellstoffwechsels auf die Nukleotidbiosynthese fördern.[85] Cyanophagen nutzen diese Gene auch, um die Photosynthese des Wirts während der Infektion aufrechtzuerhalten, indem sie die Energie von der Kohlenstofffixierung auf den Anabolismus verlagern, was das Virus ausnutzt.[87] AMGs kodieren auch für Proteine, die bei der Reparatur des Wirts-Photosystems helfen, das anfällig für Photodegradation ist.[87] Ein solches Beispiel sind virale D1-Proteine, die das D1-Protein der Wirtszelle ersetzen, wenn es beschädigt wird.[87] Da das Virus die Photosynthese hochreguliert, kommt es zu einer erhöhten Rate des D1-Proteinabbaus und die Wirtszelle allein kann diese Proteine nicht mehr in ausreichendem Maß ersetzen. Daher hilft der Cyanophage aus, so dass die Wirtszelle weiterhin Energie für den Cyanophagen-Replikationszyklus bereitstellen kann.[87]
Naturgegeben hängt die Replikation von Cyanophagen stark vom Tageszyklus ab. Der erste Schritt im Infektionszyklus besteht darin, dass der Cyanophage Kontakt zu den Cyanobakterien aufnimmt und an sie bindet; schon dieser Adsorptionsprozess ist stark von der Lichtintensität abhängig. Feldstudien zeigen auch, dass die Infektion und Replikation von Cyanophagen direkt oder indirekt mit dem Hell-Dunkel-Zyklus der Wirtszellen synchronisiert ist.[88]
Infektionsmechanismus
Cyanophagen nutzen wie andere Bakteriophagen die Brownsche Bewegung, um mit Bakterien zu kollidieren, und verwenden dann Rezeptorbindungsproteine, um bestimmte Proteine an der Zelloberfläche der Wirtszelle (Virusrezeptoren) zu erkennen, was zur Adhärenz (Anheftung) des Virusteilchens an die Wirtszelle führt. Die Viruspartikel der Caudoviricetes mit ihrem Kopf-Schwanz-Aufbau, darunter die mit kontraktilen Schwänzen, besitzen solche Rezeptorbindungsproteine an ihren Schwänzen, mit denen sie hochkonservierte Proteine auf der Oberfläche der Wirtszelle zu erkennen.[89] Cyanophagen haben auch mehrere Oberflächenproteine mit immunglobulin-ähnlichen Domänen (en. Ig-like domains, vgl. Muskelspezifische Rezeptortyrosinkinase#Proteinstruktur, Myelin-Oligodendrozyten-Glykoprotein#Physiologie), die für die Adhärenz benutzt werden.[89] Einige Cyanophagen (wie etwa die Spezies Synechococcus-Virus Syn5 alias Synechococcus-Phage syn5, Cyanophage Syn5)[90] produzieren auch eine hornartige Struktur, die vom Scheitelpunkt Kapsids gegenüber dem Schwanz („oben“) absteht. Es wird angenommen, dass die hornartige Struktur bei der Anhaftung an Zellen in der natürlichen Umgebung hilft, dies wurde jedoch nicht bestätigt.[91]
Lytischer Zyklus
Cyanophagen können sowohl den lytischen als auch den lysogenen Zyklus durchlaufen, abhängig von den Viren und ihrer Umgebung.[92][93] In einer Studie über Cyanomyoviren, die marine Synechococcus-Arten infizieren, wurde gezeigt, dass die lytische Phase etwa 17 Stunden dauert, wobei die durchschnittliche Anzahl der Virionen, die pro lysierter Zelle produziert werden (burst size ‚Burst-Größe‘), zwischen 328 bei starkem Licht und 151 bei schwachem Licht liegt.[13] Offenbar gibt es eine Korrelation zwischen Lichtintensität und Burst-Größe.[88] Weitere Studien zeigen, dass die Replikation von Cyanophagen durch Energie aus dem photosynthetischen Stoffwechsel der Wirtszelle angetrieben wird.[88] Die Lyse (Biologie) der Wirtszelle erfolgt tendenziell nach Abschluss der Replikation der Wirts-DNA und unmittelbar vor dessen Zellteilung, da dann mehr Ressourcen für die Replikation der Viruspartikel zur Verfügung stehen.
Ökologische Bedeutung
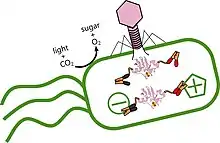
Ökosystem
Marine Cyanobakterien der Gattung Prochlorococcus sind die kleinsten und häufigsten Primärproduzenten der Welt. Cyanophagen mit lytischem Zyklus bringen diese zum Platzen.[95][40] Andere marine Cyanophagen der Familie Myoviridae oder deren Abspaltungen (d. h. marine Phagen der informellen Gattung „Cyanomyovirus“) helfen bei der Regulierung der Primärproduktion hauptsächlich durch die Infektion von Synechococcus-Spezies.[3] Cyanophagen der anderen beiden herkömmlichen Phagenfamilien, Podoviridae und Siphoviridae (d. h. der informellen Gattungen „Cyanopodovirus“ und „Cyanostylovirus“), kommen dagegen normalerweise in Süßwasser-Ökosystemen vor. In Küstenbereich der Ozeane kann die Anzahl der Viruspartikel, die Synechococcus-Spezies infizieren, 106 ml−1 (pro Milliliter) und in Sedimenten 105 g−1 (pro Gramm) überschreiten.[3] Schätzungsweise 3 % der Synechococcus-Population werden täglich durch Cyanophagen entfernt. Cyanophagen sind sowohl in der Wassersäule (vertikal) als auch geografisch (horizontal) weit verbreitet.[3][95][96] Cyanophagen-Populationen wurden durch Metagenomanalysen in mikrobiellen Matten in der Arktis und in hypersalinen Lagunen nachgewiesen.[96][4] Sie können Temperaturen von 12–30 °C und einen Salzgehalte von 18–70 ppt aushalten.[4] Die DNA von Cyanophagen ist anfällig für UV-Abbau, kann aber in Wirtszellen durch einen „Photoreaktivierung“ genannten Prozess wiederhergestellt werden.[97] Die Virionen der Cyanophagen können sich wie bei allen Viren nicht unabhängig bewegen und sind für ihren Transport auf Strömungen, Vermischung und ihre Wirtszellen angewiesen. Sie können ihre Wirte nicht aktiv ansteuern und müssen warten, bis sie auf diese treffen. Die höhere Wahrscheinlichkeit einer Kollision könnte erklären, warum Cyanophagen der Myoviren vor allem eine der häufigsten Cyanobakterien-Gattungen, Synechoccocus, infizieren.[3] Hinweise auf eine gemeinsames saisonales Auftreten zwischen den Phagen und Wirten (seasonal co-variation) sowie eine Zunahme der Cyanophagen oberhalb eines Schwellenwerts von 103 bis 104 Synechococcus pro Milliliter könnten auf eine Dynamik nach dem Prinzip „Kill the Winner“ (KTW) hindeuten.[3]
Biologische und physikalische Auswirkungen
Mitglieder der Gattung Synechococcus tragen ca. 25 % zur photosynthetischen Primärproduktivität im Ozean bei und haben einen signifikanten Bottom-up-Effekt auf höhere trophische Ebenen.[98] Die gelöste organische Materie (en. dissolved organic matter, DOM), die durch die virale Lyse von Cyanophagen freigesetzt wird, kann in den mikrobiellen Kreislauf geschleust werden, wo sie recycelt oder von heterotrophen Bakterien abgestoßen wird, um als nicht verwertbares Material (en. recalcitrant matter) schließlich im Sediment begraben zu werden.[98][99] Dies ist ein wichtiger Schritt in der atmosphärischen Kohlenstoffbindung, die als biologische Pumpe (en. biological pump) bezeichnet wird, sowie in der Aufrechterhaltung anderer biogeochemischer Kreisläufe.[98]
Cyanobakterien betreiben sauerstoffhaltige Photosynthese, von der man annimmt, dass sie der Ursprung des atmosphärischen Sauerstoffs vor etwa 2,5 Milliarden Jahren (Ga) ist.[100] Die Population und damit die Rate der Sauerstoffentwicklung kann durch Cyanophagen reguliert werden. Bei bestimmten Arten von Cyanobakterien, die Stickstofffixierung betreiben (wie z. B. Trichodesmium), sind Cyanophagen zudem in der Lage, die Zufuhrrate von bioverfügbarem organischem Stickstoff per Lyse zu erhöhen.[101][102]
Cyanophagen infizieren auch Cyanobakterien, die (bakterielle) Algenblüten verursachen, und die durch die Produktion von Microcystinen giftig für die Gesundheit von Menschen und anderen Tieren sein können. Sie können Eutrophierung verursachen, was zu Zonen minimaler Sauerstoffkonzentration führt. Algenblüten verursachen ökologische und wirtschaftliche Probleme und beeinträchtigen in Süßwassersystemen die Qualität des Trinkwassers.[103] Folgende cyanobakterieleen Verursacher von Algenblüten und können durch Cyanophagen infiziert werden:[84]
- Lyngbya birgei
- Anabaena circinalis, Anabaena flosaquae und
- Microcystis aeruginosa
Unter normalen Bedingungen sind die Cyanophagen in der Lage schädliche Algenblüten verhindern.[103] Spitzen in Cyanobakterienpopulationen werden in der Regel durch einen Nährstoffanstieg verursacht, der durch den Abfluss von Düngemitteln, Staub und Abwasser verursacht wird.[104] Indem lytische Cyanophagen die Wirte abtöten, können sie dazu beitragen, das natürliche Gleichgewicht von Ökosystemen wiederherzustellen.
Zusätzlich zur Regulierung der Populationsgröße beeinflussen Cyanophagen wahrscheinlich auch die genetische Zusammensetzung der Gewässer, indem sie anderes Phytoplankton, das normalerweise von Cyanobakterien gehemmt wird, das Wachstum ermöglichen.[104] Die Spezifität, mit der Cyanophagen auf verschiedene Wirte abzielen, beeinflusst auch die Struktur der ökologischen Gemeinschaft. Aufgrund der lysogenen Phase ihres Replikationszyklus können Cyanophagen als mobile genetische Elemente für die genetische Diversifizierung ihrer Wirte durch horizontalen Gentransfer (HGT) fördern.[105][85] Ob die lytische oder die lysogene Phase in einem bestimmten Gebiet dominiert, hängt, so die Hypothese, von eutrophen bzw. oligotrophen Bedingungen ab.[99] Die Zunahme der Anzahl der Begegnungen zwischen Phage und Wirt steht in direktem Zusammenhang mit einer Zunahme der Infektionsrate, was den Selektionsdruck erhöht und etwa Synechococcus an der Küste resistenter gegen virale Infektionen macht als seine Offshore-Pendants.[3]
Anmerkungen
- Weitere in der Studie vorgeschlagene Cyanophagen sind:
- Wirt Anabaena: „Siphoviridae-A-CS1“ und „Myoviridae A-CM1“
- Wirt Microcystis: „Podoviridae M-CP1“
- Wirt Planktothrix: „Siphoviridae PZ10“
Weblinks
- M. R. Clokie, N. H. Mann: Marine cyanophages and light. In: Environ. Microbiol.. 8, Nr. 12, Dezember 2006, S. 2074–2082. doi:10.1111/j.1462-2920.2006.01171.x. PMID 17107549.
- N. H. Mann: Phages of the marine cyanobacterial picophytoplankton. In: FEMS Microbiol. Rev.. 27, Nr. 1, April 2003, S. 17–34. doi:10.1016/S0168-6445(03)00016-0. PMID 12697340.
- J. H. Paul, M. B. Sullivan: Marine phage genomics: what have we learned?. In: Current Opinion in Biotechnology. 16, Nr. 3, Juni 2005, S. 299–307. doi:10.1016/j.copbio.2005.03.007. PMID 15961031.
- C. A. Suttle; B. A. Whitton, M. Potts (Hrsg.): Chapter 20: Cyanophages and their role in the ecology of cyanobacteria. In: The Ecology of Cyanobacteria: Their Diversity in Time and Space. Kluwer Academic Publishers, 2000, ISBN 978-0-7923-4755-2, S. 563–589.
- Dona A. C. T. Thuduhenage, Anjana Chamilka Thuduhena: Harmful Cyanobacterial Blooms And Developed Cyanophages As A Biological Solution, in: SviCell Bacterial Empire Band 2, Nr. 1, S. 6–9, Januar 2019, doi:10.13140/RG.2.2.16241.94568, ResearchGate, insbes. Fig. 3. In Tabelle 1 ist LPP-1 fehlerhaft den Siphoviridae zugeordnet, seine Morphologie ist jedoch Podovirus-artig (Fig. 3a)
- Virus Ecology Group (VEG) (via WebArchiv vom 3. April 2017)
- Bacteriophage Ecology Group (BEG) (via WebArchiv vom 3. Juni 2013)
- The Sullivan Lab, Ohio State University (OSU)
Einzelnachweise
- Han Xia, Tianxian Li, Fei Deng, Zhihong Hu: Freshwater cyanophages. In: Virologica Sinica. 28, Nr. 5, 1. Oktober 2013, ISSN 1674-0769, S. 253–259. doi:10.1007/s12250-013-3370-1. PMID 24132756. ResearchGate.
- Brian A. Whitton, Malcolm Potts: The Ecology of Cyanobacteria: Their Diversity in Time and Space. Kluwer Academic, Boston 2000, ISBN 978-0-7923-4735-4, S. 563–589.
- Curtis A. Suttle: Cyanophages and Their Role in the Ecology of Cyanobacteria. In: Brian A. Whitton (Hrsg.): The Ecology of Cyanobacteria (en). Springer Netherlands, 1. Januar 2000, ISBN 978-0-7923-4735-4, S. 563–589, doi:10.1007/0-306-46855-7_20.
- Curtis A. Suttle, Amy M. Chan: Marine cyanophages infecting oceanic and coastal strains of Synechococcus: abundance, morphology, cross-infectivity and growth characteristics. In: Marine Ecology Progress Series. 92, 26. Januar 1993, S. 99–109. bibcode:1993MEPS...92...99S. doi:10.3354/meps092099.
- Dann Turner, Andrew M. Kropinski, Evelien M. Adriaenssens: A Roadmap for Genome-Based Phage Taxonomy, in: MDPI Viruses Band 13, Nr. 3, Section Bacterial Viruses, 18. März 2021, 506, doi:10.3390/v13030506
- Robert S. Safferman, M. E. Morris: Algal virus: Isolation, in: Science Band 140, Nr. 3567, 10. Mai 1963, S. 679–680, doi:10.1126/science.140.3567.679
- Marisha Garg: Cyanophages: Discovery, Morphology and Replication| Microbiology, auf: Biology Discussion
- P. Ashwathi: Cyanophages: Morphology and Growth Cycle | Microbiology, auf: Biology Discussion
- Etana Padan, Moshe Shilo: Cyanophages- viruses attacking blue-green algae. In: Bacteriological Reviews. 37, Nr. 3, September 1973, S. 343–370. doi:10.1128/MMBR.37.3.343-370.1973. PMID 4202147. PMC 413822 (freier Volltext). Mit Photos und Schemazeichnung.
- T. A. Sarma: Cyanophages, in: Handbook of Cyanobacteria, CRC Press, 2012, ISBN 1-4665-5941-1
- Louis A. Sherman, Robert Haselkorn: LPP-1 infection of the blue-green alga Plectonema boryanum. I. Electron microscopy. J Virol Band 6, Nr. 6, Dezember 1970, S. 820–833, doi:10.1128/JVI.6.6.820-833.1970, PMC 376200 (freier Volltext), PMID 4992998
- Louis A. Sherman, Robert Haselkorn: LPP-1 infection of the blue-green alga Plectonema boryanum. II. Viral Deoxyribonucleic Acid Synthesis and Host Deoxyribonucleic Acid Breakdown. J Virol Band 6, Nr. 6, Dezember 1970, S. 820–833, doi:10.1128/JVI.6.6.834-840.1970, PMC 376201 (freier Volltext), PMID 4992999
- Sean Malcolm Brigden: Dynamics of Cyanophage Replication, Dissertation (Msc) am Department of Botany, University of British Columbia (UBC), Mai 2003, doi:10.14288/1.0091069
- Ronald Luftig, Robert Haselkorn: Comparison of blue-green algae virus LPP-1 and the morphologically related viruses GIII and coliphage T7, in: Virology, Band 34, Nr. 4, April 1968, S. 675–678, doi:10.1016/0042-6822(68)90088-3, PMID 5650701, ResearchGate
- M. J. Daft, J. Begg, W. D. P. Stewart: A Virus of Blue-Green Algae From Freshwater Habitats in Scotland, in: New Phytologist, Band 69, Nr. 4, Oktober 1970, S. 1029–1038, doi:10.1111/j.1469-8137.1970.tb02483.x
- NCBI: Cyanobacteria phage AS-1 (spezies)
- NCBI: Anabaena phage N-1 (spezies)
- Markus G. Weinbaue: Ecology of prokaryotic viruses. (PDF; 658 kB) In: FEMS Microbiology Reviews, Band 28, Nr. 2, 1. Mai 2004, Epub 27. Oktober 2003, S. 127–181. doi:10.1016/j.femsre.2003.08.001. Offenbar Gleichsetzung von „Cyanophage AS-1“ und „S-1“, in der Folge „Cyanostylovirus“ zu Myoviridae.
- Ivan Y. Khudyakov, M. D. Kirnos, N. I. Alexandrushkina, B. F. Vanyushin: Cyanophage S-2L contains DNA with 2,6-diaminopurine substituted for adenine. In: Virology. 88, Nr. 1, 1978, S. 8–18. PMID 676082.
- Yan-Jiun Lee, Nan Dai, Shannon E. Walsh, Stephanie Müller, Morgan E. Fraser, Kathryn M. Kauffman, Chudi Guan, Ivan R. Corrêa Jr., Peter R. Weigele: Identification and biosynthesis of thymidine hypermodifications in the genomic DNA of widespread bacterial viruses. (PDF) In: PNAS, 16. März 2018; doi:10.1073/pnas.1714812115
- NCBI: Cyanophage S-2L (species)
- Andrew M. Kropinski et al.: The Sequence of Two Bacteriophages with Hypermodified Bases Reveals Novel Phage-Host Interactions. In: Viruses, Band 10, Nr. 5, Mai 2018, S. 217, doi:10.3390/v10050217, PMC 5977210 (freier Volltext), PMID 29695085.
- Yan Zhou, Xuexia Xu, Yifeng Wei, Yu Cheng, Yu Guo, Ivan Y. Khudyakov, Fuli Liu, Ping He, Zhangyue Song, Zhi Li, Yan Gao, Ee Lui Ang, Huimin Zhao, Yan Zhang, Suwen Zhao: A widespread pathway for substitution of adenine by diaminopurine in phage genomes. In: Science. 372, Nr. 6541, 30. April 2021. doi:10.1126/science.abe4882.
- Dona Sleiman, Pierre Simon Garcia, Marion Lagune, Jerome Loc’h, Ahmed Haouz, Najwa Taib, Pascal Röthlisberger, Simonetta Gribaldo, Philippe Marlière, Pierre Alexandre Kaminski: A third purine biosynthetic pathway encoded by aminoadenine-based viral DNA genomes. In: Science. 372, Nr. 6541, 30. April 2021. doi:10.1126/science.abe6494.
- Valerie Pezo, Faten Jaziri, Pierre-Yves Bourguignon, Dominique Louis, Deborah Jacobs-Sera, Jef Rozenski, Sylvie Pochet, Piet Herdewijn, Graham F. Hatfull, Pierre-Alexandre Kaminski, Philippe Marliere: Noncanonical DNA polymerization by aminoadenine-based siphoviruses. In: Science. 372, Nr. 6541, 30. April 2021. doi:10.1126/science.abe6542.
- Jacinta Bowler: Some Viruses Have a Completely Different Genome to The Rest of Life on Earth, auf: sciencealert vom 4. Mai 2021
- Tina Hesman Saey: Some viruses thwart bacterial defenses with a unique genetic alphabet, auf: ScienceNews vom 5. Mai 2021
- US20060270005A1, Genomic library of cyanophage s-2l and functional analysis
- M. H. V. van Regenmortel et al.: ICTV 7th Report (PDF; 857 kB) 2000
- Robert S. Safferman, R. E. Cannon, P. R. Desjardins, B. V. Gromov, R. Haselkorn, L. A. Sherman, M. Shilo: Classification and Nomenclature of Viruses of Cyanobacteria. In: Intervirology. 19, Nr. 2, 1983, S. 61–66. doi:10.1159/000149339. PMID 6408019.
- Roger Hull, Fred Brown, Chris Payne (Hrsg.): Virology: A Directory and Dictionary of Animal, Bacterial and Plant Viruses. Macmillan Reference Books, ISBN 978-1-349-07947-6; doi:10.1007/978-1-349-07945-2 books.google.de
- Li Deng, Paul K. Hayes: Evidence for cyanophages active against bloom-forming freshwater cyanobacteria, in: Freshwater Biology Band 53, Nr. 6, Juni 2008, S. 1240–1252, doi:10.1111/j.1365-2427.2007.01947.x, ResearchGate
- Mélanie Gerphagnon, Deborah J. Macarthur, Delphine Latour, Claire M. M. Gachon, Floris Van Ogtrop, Frank H. Gleason, Télesphore Sime-Ngando: Microbial players involved in the decline of filamentous and colonial cyanobacterial blooms with a focus on fungal parasitism, in: sfam Environmental Microbiology, Band 17, Nr. 8, S. 2573–2587, doi:10.1111/1462-2920.12860, PDF; insbes. Supplement (PDF) (PDF) Table 2
- E-Bin Gao, Xiu-Ping Yuan, Ren-hui Li, Qi-Ya Zhang: Isolation of a novel cyanophage infectious to the filamentous cyanobacterium Planktothrix agardhii (Cyanophyceae) from Lake Donghu, China; in: Aquat Microb Ecol (AME), Band 54, Nr. 1, Februar 2009, S.&nnbsp;163–170; doi:10.3354/ame01266, int-res.com (PDF; 650 kB)
- NCBI: Planktothrix phage PaV-LD (species)
- Lita M. Proctor, Jed A. Fuhrman: Viral mortality of marine bacteria and cyanobacteria. In: Nature. 343, Nr. 6253, 1990, S. 60–62. bibcode:1990Natur.343...60P. doi:10.1038/343060a0.
- A. M. Q. King, E. Lefkowitz, M. J. Adams, E. B. Carstens: Virus Taxonomy Classification and Nomenclature of Viruses: Ninth Report of the International Committee on Taxonomy of Viruses. Elsevier, 2012, ISBN 978-0-12-384684-6.
- Adrian J. Gibbs: Molecular Bases of Virus Evolution. Cambridge University Press, 2005, ISBN 978-0-521-02289-7.
- R. Y. Stanier: Purification and properties of unicellular blue-green algae (order Chroocococcales). In: Bacteriological Reviews. 35, Nr. 2, 1971, S. 171–205. doi:10.1128/MMBR.35.2.171-205.1971. PMID 4998365. PMC 378380 (freier Volltext).
- Matthew B. Sullivan, Maureen L. Coleman, Peter Weigele, Forest Rohwer, Sallie W. Chisholm: Three Prochlorococcus Cyanophage Genomes: Signature Features and Ecological Interpretations. In: PLOS Biology. 3, Nr. 5, 19. April 2005, ISSN 1545-7885, S. e144. doi:10.1371/journal.pbio.0030144. PMID 15828858. PMC 1079782 (freier Volltext).
- Robert S. Safferman, Theodor O. Diener, Paul R. Desjardins, M. E. Morris: Isolation and characterization of AS-1, a phycovirus infecting the blue-green algae, Anacystis nidulans and Synechococcus cedrorum. In: Virology. 47, Nr. 1, 1972, S. 105–113. doi:10.1016/0042-6822(72)90243-7. PMID 4110125.
- Robert S. Safferman, Mary-Ellen Morris: Growth Characteristics of the Blue-Green Algal Virus LPP-1. In: J. Bacteriol.. 88, Nr. 3, 1964, S. 771–775. doi:10.1128/JB.88.3.771-775.1964. PMID 14208517. PMC 277376 (freier Volltext).
- B.V. Gromov: Cyanophages. In: Annales de l’Institut Pasteur / Microbiologie. 134, Nr. 1, 1983, S. 43–59. doi:10.1016/s0769-2609(83)80096-9. PMID 6416127.
- NCBI: Synechococcus phage S-PM2 (species)
- John H. Paul, Matthew B. Sullivan: Marine phage genomics: what have we learned?, Curr. Op. in Biotechnology Band 16, Nr. 3, Juni 2005, S. 299–307; doi:10.1016/j.copbio.2005.03.007, PMID 15961031, PMC 15961031 (freier Volltext), siehe Fig. 3 (Genomkarte)
- NCBI: Synechococcus virus Syn5 (species)
- NCBI: Synechococcus phage syn9 (species)
- NCBI: Cyanophage Syn9 (species)
- NCBI: Synechococcus virus S-SM1 (species)
- NCBI: Prochlorococcus virus PSSM2 (species)
- NCBI: Prochlorococcus phage P-SSM4 (species)
- NCBI: Synechococcus phage S-RSM2 (species)
- NCBI:Cyanophage S-BM4 (species)
- NCBI:Synechococcus phage S-WHM1 (specis)
- NCBI:Cyanophage S-RSM88 (species)
- NCBI: unclassified Teseptimavirus (list)
- Sijun Huang et al.: Temporal transcriptomes of a marine cyanopodovirus and its Synechococcus host during infection, in: MicrobiologyOpen, 30. Dezember 2020, doi:10.1002/mbo3.1150
- NCBI: Synechococcus phage S-SBP1 (species)
- NCBI: Synechococcus phage S-RIP2 (species)
- Sijun Huang, Si Zhang, Nianzhi Jiao, Feng Chen: Comparative Genomic and Phylogenomic Analyses Reveal a Conserved Core Genome Shared by Estuarine and Oceanic Cyanopodoviruses. In: PLOS ONE, 16. November 2015, doi:10.1371/journal.pone.0142962
- NCBI: Prochlorococcus virus PSSP7 (species)
- NCBI: Synechococcus virus P60 (species)
- Nien-Tai Hu, Teresa Thiel, Thomas H. Giddings, C.Peter Wolk: New Anabaena and Nostoc cyanophages from sewage settling ponds. In: Virology. 114, Nr. 1, 1981, S. 236–246. doi:10.1016/0042-6822(81)90269-5. PMID 6269286.
- I. R. Schneider, Theodor O. Diener, Robert S. Safferman: Blue-Green Algal Virus LPP-1: Purification and Partial Characterization. In: Science. 144, Nr. 3622, 29. Mai 1964, ISSN 0036-8075, S. 1127–1130. bibcode:1964Sci...144.1127S. doi:10.1126/science.144.3622.1127. PMID 14148431.
- Itai Sharon et al.: Viral photosynthetic reaction center genes and transcripts in the marine environment, in: The ISME Journal Band 1, S. 492–501, Oktober 2007, doi:10.1038/ismej.2007.67
- Ruth-Anne Sandaa et al.: Photosynthetic genes in viral populations with a large genomic size range from Norwegian coastal waters, in: FEMS Microbiology Ecology, Band 63, Nr. 1, 1. Januar 2008, S. 2–11, doi:10.1111/j.1574-6941.2007.00400.x
- Stephen Tucker, Peter Pollard: Identification of Cyanophage Ma-LBP and Infection of the Cyanobacterium Microcystis aeruginosa from an Australian Subtropical Lake by the Virus. In: ASM Applied and Environmental Microbiology, Band 71, Nr. 2, 3. Februar 2005, S. 629–635,doi:10.1128/AEM.71.2.629-635.2005
- NCBI: Cyanophage Ma-LEP, equivalent: Microcystis phage Ma-LEP (species)
- Kenneth W. Adolph, Robert Haselkorn: Isolation and characterization of a virus infecting a blue-green alga of the genus Synechococcus. In: Virology. 54, Nr. 1, 1973, S. 230–236. doi:10.1016/0042-6822(73)90132-3. PMID 4197413.
- NCBI: Cyanophage KBS-S-2A (species)
- NCBI: Cyanophage MED4-117 (species)
- NCBI: Cyanophage PSS2 (species)
- David W. Johnson, Malcolm Potts: Host Range of LPP Cyanophages. In: International Journal of Systematic Bacteriology. 35, 1985, S. 76–78. doi:10.1099/00207713-35-1-76.
- Robert S. Safferman, I. R. Schneider, R. L. Steere, M. E. Morris, T. O. Diener: Phycovirus SM-1: A virus infecting unicellular blue-green algae. In: Virology. 37, Nr. 3, 1969, S. 386–397. doi:10.1016/0042-6822(69)90222-0. PMID 5777559.
- K. G. Orkwiszewski, A. R. Kaney: Genetic transformation of the blue-green bacterium, Anacystis nidulans. In: Arch Mikrobiol. 98, Nr. 1, Juni 1974, S. 31–37. doi:10.1007/BF00425265. PMID 4209657.
- AlgaeBase: Microcystis
- Netzblaualge (Microcystis sp.), auf: PlingFactory (Fachbereich Biologie des Städtischen Gymnasiums Gevelsberg)
- NCBI: Cyanophage S-SM1 (species)
- John A. Fox, S. J. Booth, Eugene L. Martin: Cyanophage SM-2: A new blue-green algal virus. In: Virology. 73, Nr. 2, 1976, S. 557–560. doi:10.1016/0042-6822(76)90420-7. PMID 8869.
- NCBI: Cyanophage S-SM2 (species)
- M. M. Muradov, G. V. Cherkasova, D. U. Akhmedova, Faina Kamilova, R. S. Mukhamedov, A. A. Abdukarimov, A. G. Khalmuradov: Comparative characteristics of NP-1T cyanophages causing lysis of nitrogen-fixing Nostoc and Plectonema cultures. In: Microbiology. 59, Nr. 5, Januar 1990, S. 558–563. (Translation of Mikrobiologiya 59, S. 819–826)
- S. Ya. Koza'yakov: Cyanophages of the series A(L) specific for the blue-green alga Anabaena variabilis. In: Experimental Algology. Nr. 25, 1977, S. 151–171. Eksperimental'naya al'gologiya. Trudi Petergof Biological Institute, Leningrad State University
- C. Chénard C, J. F. Wirth, C. A. Suttle: Viruses Infecting a freshwater filamentous cyanobacterium (Nostoc sp.) encode a functional CRISPR array and a proteobacterial DNA polymerase B. In: mBio. 7, 2016, S. e00667-16. doi:10.1128/mBio.00667-16. PMID 27302758. PMC 4916379 (freier Volltext).
- Sabah A. A. Jassim, Richard G. Limoges: Impact of external forces on cyanophage–host interactions in aquatic ecosystems. In: World Journal of Microbiology and Biotechnology. 29, Nr. 10, 1. Oktober 2013, ISSN 0959-3993, S. 1751–1762. doi:10.1007/s11274-013-1358-5. PMID 23619821.
- Aaron Kaplan: Cyanophages: Starving the Host to Recruit Resources. In: Cell. 26, Nr. 12, 2016, S. R511–R513. doi:10.1016/j.cub.2016.04.030. PMID 27326715.
- K. G. Raghothama, A. S. Karthikeyan: Phosphate acquisition, in: Plant and Soil, Juli 2005, Band 274, Nr. 37,: S. 37–49, doi:10.1007/s11104-004-2005-6
- Jeremy A. Frank, Don Lorimer, Merry Youle, Pam Witte, Tim Craig, Jan Abendroth, Forest Rohwer, Robert A. Edwards, Anca M. Segall: Structure and function of a cyanophage-encoded peptide deformylase. In: The ISME Journal. 7, Nr. 6, 1. Juni 2013, ISSN 1751-7362, S. 1150–1160. doi:10.1038/ismej.2013.4. PMID 23407310. PMC 3660681 (freier Volltext).
- Tianchi Ni, Qinglu Zeng: Diel Infection of Cyanobacteria by Cyanophages. In: Frontiers in Marine Science. 2, 1. Januar 2016, ISSN 2296-7745. doi:10.3389/fmars.2015.00123.
- Alexander B. Westbye, Kevin Kuchinski, Calvin K. Yip, J. Thomas Beatty: The Gene Transfer Agent RcGTA Contains Head Spikes Needed for Binding to the Rhodobacter capsulatus Polysaccharide Cell Capsule. In: Journal of Molecular Biology. 428, Nr. 2B, 29. Januar 2016, S. 477–491. doi:10.1016/j.jmb.2015.12.010. PMID 26711507.
- NCBI: Synechococcus virus Syn5 (species)
- Desislava A. Raytcheva, Cameron Haase-Pettingell, Jacqueline Piret, Jonathan A. King: Two Novel Proteins of Cyanophage Syn5 Compose Its Unusual Horn Structure. In: ASM Journal of Virology. 88, Nr. 4, 15. Februar 2014, ISSN 0022-538X, S. 2047–2055. doi:10.1128/JVI.02479-13. PMID 24307583. PMC 3911526 (freier Volltext). Epub 31. Januar 2014, insbes. Fig. 1 (mit A: Photo, B: Schemazeichnung)
- Lauren McDaniel, Lee A. Houchin, Shannon J. Williamson, John P. Paul: Plankton blooms - Lysogeny in marine Synechococcus. In: Nature. 415, Nr. 6871, 2002, S. 496. bibcode:2002Natur.415..496M. doi:10.1038/415496a. PMID 11823851.
- Alice C. Ortmann, Janice E. Lawrence, Curtis A. Suttle: Lysogeny and lytic viral production during a bloom of the cyanobacterium Synechococcus spp.. In: Microbial Ecology. 43, Nr. 2, 2002, S. 225–231. doi:10.1007/s00248-001-1058-9. PMID 12023729.
- Ian J. Campbell, Jose Luis Olmos Jr., Weijun Xu, Dimithree Kahanda, Joshua T. Atkinson, Othneil Noble Sparks, Mitchell D. Miller, George N. Phillips Jr., George N. Bennett, Jonathan J. Silberg: Prochlorococcus phage ferredoxin: Structural characterization and electron transfer to cyanobacterial sulfite reductases – Phage Fd characterization and host SIR interactions, in: J. Biol. Chem., ASBMB Publications, 19. Mai 2020, doi:10.1074/jbc.RA120.013501, jbc.org (PDF). Siehe auch:
- Ocean virus hijacks carbon-storing bacteria, Quelle: Rice University
- National Science Foundation (NSF), 29. Mai 2020
- EurekAlert, 26 Mai 2020
- Ocean ScienceDaily, 26. Mai 2020
- Beneath the Ocean’s Surface, a Virus Is Hijacking the Most Abundant Organism on Earth, auf: SciTechDaily, 6. Juni 2020. Quelle: Rice University
- Ocean virus hijacks carbon-storing bacteria, Quelle: Rice University
- F. Partensky, W. R. Hess, D. Vaulot: Prochlorococcus, a Marine Photosynthetic Prokaryote of Global Significance. In: Microbiology and Molecular Biology Reviews. 63, Nr. 1, 1. März 1999, ISSN 1092-2172, S. 106–127. doi:10.1128/MMBR.63.1.106-127.1999. PMID 10066832. PMC 98958 (freier Volltext).
- Thibault Varin, Connie Lovejoy, Anne D. Jungblut, Warwick F. Vincent, Jacques Corbeila: Metagenomic profiling of Arctic microbial mat communities as nutrient scavenging and recycling systems. In: Limnology and Oceanography. 55, Nr. 5, 2010, S. 1901–1911. bibcode:2010LimOc..55.1901V. doi:10.4319/lo.2010.55.5.1901.
- Kai Cheng, Yijun Zhao, Xiuli Du, Yaran Zhang, Shubin Lan, Zhengli Shi: Solar radiation-driven decay of cyanophage infectivity, and photoreactivation of the cyanophage by host cyanobacteria. In: Aquatic Microbial Ecology. 48, Nr. 1, 20. Juni 2007, S. 13–18. doi:10.3354/ame048013.
- Kui Wang, K. Eric Wommack, Feng Chen: Abundance and Distribution of Synechococcus spp. and Cyanophages in the Chesapeake Bay. In: Applied and Environmental Microbiology. 77, Nr. 21, 1. November 2011, ISSN 0099-2240, S. 7459–7468. doi:10.1128/AEM.00267-11. PMID 21821760. PMC 3209163 (freier Volltext).
- Markus Weinbauer, Feng Chen, Steven Wilhelm: Virus-Mediated Redistribution and Partitioning of Carbon in the Global Oceans. In: Microbial Carbon Pump in the Oceans. 2011, S. 54–56.
- Bettina E. Schirrmeister, Alexandre Antonelli, Homayoun C. Bagheri: The origin of multicellularity in cyanobacteria. In: BMC Evolutionary Biology. 11, 1. Januar 2011, ISSN 1471-2148, S. 45. doi:10.1186/1471-2148-11-45. PMID 21320320. PMC 3271361 (freier Volltext).
- Birgitta Bergman, Gustaf Sandh, Senjie Lin, John Larsson, Edward J. Carpenter: Trichodesmium – a widespread marine cyanobacterium with unusual nitrogen fixation properties. In: FEMS Microbiology Reviews. 37, Nr. 3, 1. Mai 2013, ISSN 0168-6445, S. 286–302. doi:10.1111/j.1574-6976.2012.00352.x. PMID 22928644. PMC 3655545 (freier Volltext).
- A. K. Kashyap, A. N. Rai, Surendra Singh: Effect of cyanophage N-1 development on nitrogen metabolism of cyanobacterium Nostoc muscorum. In: FEMS Microbiology Letters. 51, Nr. 2–3, 1. Juni 1988, ISSN 0378-1097, S. 145–148. doi:10.1111/j.1574-6968.1988.tb02986.x.
- Lucas J. Beversdorf, Todd R. Miller, Katherine D. McMahon: The Role of Nitrogen Fixation in Cyanobacterial Bloom Toxicity in a Temperate, Eutrophic Lake. In: PLOS ONE. 8, Nr. 2, 6. Februar 2013, ISSN 1932-6203, S. e56103. bibcode:2013PLoSO...856103B. doi:10.1371/journal.pone.0056103. PMID 23405255. PMC 3566065 (freier Volltext).
- Jed A. Fuhrman, Curtis A. Suttle: Viruses in marine planktonic systems. In: Oceanography. 6, Nr. 2, 1993, S. 51–63. doi:10.5670/oceanog.1993.14.
- Laura S. Frost, Raphael Leplae, Anne O. Summers, Ariane Toussaint: Mobile genetic elements: the agents of open source evolution. In: Nature Reviews Microbiology. 3, Nr. 9, 2005, S. 722–732. doi:10.1038/nrmicro1235. PMID 16138100.