Retroviren
Retroviren (Retroviridae) sind eine große Familie behüllter Viren mit Einzel(+)-strängigem-RNA-Genom, deren Erbinformation (ss(+)-RNA) dementsprechend in Form von Ribonukleinsäure vorliegt. Anders als bei „normalen“ RNA-Viren aber muss die RNA von Retroviren zunächst einmal mittels reverser Transkription in ein DNA-Molekül umgeschrieben werden, bevor sie als solches in das Genom der Wirtszelle eingebaut und dort aktiv werden kann.
| Retroviren | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
HI-Virus (Grafik) | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Retroviridae | ||||||||||||||
| Links | ||||||||||||||
|
Retroviren können grob in einfache und komplexe Retroviren unterteilt werden. Des Weiteren unterscheidet man zwischen infektiösen exogenen Retroviren (teilweise mit XRV abgekürzt) und den endogenen Retroviren (ERV), die vertikal über die Keimbahn vererbt werden und auf diese Weise Bestandteil des Wirtszell-Genoms werden.
Retroviren infizieren vornehmlich tierische Zellen und sind bei Wirbeltieren allgegenwärtig: Sie infizieren Säugetiere, Vögel, Amphibien, Reptilien und Fische, sind dabei aber meist sehr wirtsspezifisch. Zu ihnen gehören die Erreger einiger weit verbreiteter Infektionskrankheiten, die sowohl bei Menschen als auch bei Tieren pandemisch bzw. epidemisch auftreten. Als Auslöser von Krankheiten beim Menschen sind unter anderem HIV und HTLV-1 bekannt.
Taxonomie

Historisch wurden die Retroviren zunächst nach ihrem elektronenmikroskopischen Erscheinungsbild in Typ A-, B-, C- oder D-Retroviren eingeteilt. Später folgte eine Klassifikation, die auch biochemische Eigenschaften und den Zelltropismus berücksichtigte. Die Klassifikation unterschied Onkornaviren, Spumaviren und die Lentiviren.
Innere Systematik
Die aktuelle, zurzeit verbindliche Taxonomie durch das International Committee on Taxonomy of Viruses (ICTV) unterteilt die Retroviren vor allem aufgrund ihrer genetischen Verwandtschaftsverhältnisse wie folgt in zwei Unterfamilien und elf Gattungen:
Familie: Retroviren (Retroviridae)
- Unterfamilie: Orthoretroviren (Orthoretrovirinae)
- Gattungen:
- Alpharetrovirus
- Betaretrovirus – mit vorgeschlagener Species „Humanes Mammatumorvirus“
- Gammaretrovirus
- Deltaretrovirus – mit Species Primate T-lymphotropic virus 1 (HTLV-1), Primate T-lymphotropic virus 2 (HTLV-2)
- Epsilonretrovirus
- Lentivirus – mit Species HIV-1, HIV-2, SIV, BIV, FIV
- Gattungen:
- Unterfamilie: Foamy- oder Spumaretroviren (Spumaretrovirinae)
- Gattungen:
- Bovines Foamyvirus (Bovispumavirus)
- Equines Foamyvirus (Equispumavirus)
- Felines Foamyvirus (Felispumavirus)
- Prosimiispumavirus
- Simiispumavirus
- Gattungen:
Beim Menschen sind bisher fünf Retroviren bekannt: Humanes T-lymphotropes Virus 1 (HTLV-1) und Humanes T-lymphotropes Virus 2 (HTLV-2), beide Deltaretroviren; Humanes Immundefizienz-Virus-1 (HIV-1), Humanes Immundefizienz-Virus Typ II (HIV-2), beide Lentiviren; sowie Xenotropic murine leukemia virus-related virus (XMRV), ein Gammaretrovirus. Die menschlichen Retroviren sind denen anderer Primaten so eng verwandt, dass häufig beide Gruppen unter der Bezeichnung Primaten-Retroviren zusammengefasst werden. Es wird davon ausgegangen, dass die entsprechenden menschlichen Retroviren durch Übertragung von Affen-Retroviren auf den Menschen entstanden sind. Bei HTLV-1 und HTLV-2 hat diese Übertragung wohl schon vor Jahrtausenden stattgefunden, für HIV-1 und HIV-2 wahrscheinlich im 20. Jahrhundert. Die Foamyviren wurden in verschiedene Gattungen aufgespalten und bilden jetzt eine Unterfamilie der Retroviren (ICTV-Stand November 2018).
Einfache und komplexe Retroviren
Die Unterteilung in einfache und komplexe Retroviren erfolgt nach der Genomorganisation bzw. der Translation von akzessorischen Proteinen. Dabei umfassen erstere Alpha-, Gamma-, Epsilon- und die meisten Betaretroviren, letztere die Deltaretroviren sowie Lenti- und Foamyviren.
Äußere Systematik und Pararetroviren
Die Retroviridae gehören mit weiteren Virusfamilien (Belpaoviridae, Metaviridae und Pseudoviridae) zu den revers transkribierenden RNA-Viren der Virusordnung Ortervirales, die noch die Caulimoviridae als revers transkribierenden DNA-Viren gehören. Die Caulimoviridae werden manchmal mit den nicht näher verwandten, aber ebenfalls revers transkribierenden DNA-Viren der Hepadnaviridae als Pararetroviren (Baltimore-Gruppe 7) klassifiziert.
Geschichte der Retrovirologie
Seit dem 19. Jahrhundert waren bei Haustieren Krankheiten wie die Bovine Leukose oder Jaagsiekte bei Schafen bekannt, ihre Ursache blieb jedoch unklar. 1904 wurde für die erste durch Retroviren verursachte Krankheit, die Ansteckende Blutarmut der Einhufer, durch zwei französische Tierärzte, Vallée und Carré, gezeigt, dass sie sich mit einem Filtrat auf andere Pferde übertragen ließ. Onkogene (Tumoren auslösende) Retroviren wurden seit 1908 untersucht, als die dänischen Pathologen Vilhelm Ellermann und Oluf Bang zeigten, dass sich durch zellfreie Filtrate Hühnerleukämie auf andere Hühner übertragen ließ und die damit die erste ansteckende Krebserkrankung beschrieben.[3] Dieses Virus ist heute als aviäres Leukosevirus (ALV) bekannt, zunächst wurde die Leukose jedoch nicht als echte Leukämie angesehen und Leukämien wurden nicht als echte Tumoren betrachtet. Diese frühen Untersuchungen blieben innerhalb der Wissenschaftsgemeinde weitgehend unbeachtet, erst viel später konnte ihre Bedeutung im Zusammenhang mit Retroviren erkannt werden. Peyton Rous stellte 1911 fest, dass mit filtrierten Extrakten aus Hühnersarkomen gesunde Hühner infiziert werden konnten, die daraufhin ebenfalls Tumoren entwickelten. Das Virus wurde später nach ihm Rous-Sarkom-Virus (RSV) genannt und Rous erhielt 1966, 54 Jahre nach seiner Erstbeschreibung, den Nobelpreis. Erst 1961 wurde festgestellt, dass Rous-Sarkom-Viren Ribonukleinsäure (RNA) enthalten, sie wurden daher (bis 1974) als RNA-Tumorviren bezeichnet.
Die Entdeckung, dass Viren Tumoren auslösen können, bestätigte sich 1936 auch bei Säugetieren: John J. Bittner beschrieb das Maus-Mammatumorvirus (MMTV). 1951 wurde das Murine Leukämievirus (MLV) isoliert und erstmals die vertikale Übertragung von Eltern auf die Nachkommen beschrieben. 1964 wurde von Howard M. Temin die Provirus-Hypothese vorgeschlagen, da beobachtet wurde, dass Zellen, die durch RSV „transformiert“ wurden (Tumor-Eigenschaften erhielten), auch in Abwesenheit des Virus die transformierten Eigenschaften beibehielten. Aus diesem Grund postulierte Temin in Anlehnung an temperente Bakteriophagen, von denen man bereits wusste, dass sie ins Genom ihres Wirts integrieren können, dass die RNA-Tumorviren dies ebenfalls tun. Bereits 1960 war von André Lwoff vorgeschlagen worden, dass DNA-Tumorviren (Polyomaviren) in das Genom ihres Wirts integrieren können. 1968 wurde gezeigt, dass diese Annahme zutrifft. Dass auch RNA-Tumorviren über die Keimbahn vererbt werden können, wurde weiterhin als bizarr betrachtet.[4]
Endogene Retroviren wurden gegen Ende der 1960er Jahre entdeckt. Die Vermutung, dass ganze virale Genome durch ihre Wirte nach den Mendelschen Regeln weitervererbt werden, war eine völlig neue Vorstellung, und die Provirus-Hypothese von Temin wurde immer noch nicht allgemein akzeptiert, teilweise sogar für unmöglich gehalten. Die Reverse Transkriptase, durch die RNA in DNA umgeschrieben wird, wurde 1970 nachgewiesen, und die Familie der RNA-Tumorviren als Folge 1974 in Retroviren umbenannt. Temins Provirus-Hypothese erwies sich nach der Entdeckung der Reversen Transkriptase endgültig als zutreffend.
Zu Beginn der 1970er Jahre wurden auch die ersten viralen Proteine beschrieben und im Verlauf der darauffolgenden Jahre der Replikationszyklus der Retroviren nach und nach in groben Zügen aufgeklärt. 1978 wurden die LTR-Regionen (Long Terminal Repeats) im Genom der Retroviren entdeckt, zwei Jahre später wurde ein Jumping scheme für den komplexen Vorgang der reversen Transkription vorgeschlagen. Die Technik der DNA-Sequenzierung, die Anfang der 1980er Jahre aufkam, führte 1981 zur ersten Publikation der vollständigen genomischen Sequenz eines Retrovirus, des Moloney murine leukemia virus.
1980 erfolgte die Erstbeschreibung des humanen T-Zell-Leukämie-Virus Typ 1 (HTLV-1), des ersten Retrovirus, das den Menschen infiziert, nachdem viele Jahre lang erfolglos in allen möglichen Tumorgeweben des Menschen nach Retroviren gesucht wurde. Kurze Zeit darauf entdeckten Luc Montagnier und Françoise Barré-Sinoussi (Nobelpreis für Medizin 2008) das HIV-1, HIV-2 folgte 1986. Spätestens seitdem 1988 klar wurde, dass HIV die Ursache der Immunschwächeerkrankung AIDS ist, entwickelte sich die Retrovirologie von einer eher exotischen Grundlagenforschung zu dem am intensivsten beforschten Gebiet in der Virologie mit großer Bedeutung für die Gesundheitswissenschaften.
Aufbau
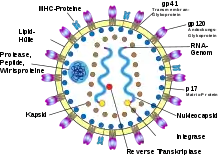
Viruspartikel
Infektiöse Retrovirus-Partikel haben einen Durchmesser von etwa 100 nm. Sie besitzen ein Kapsid, das von einer Virushülle umgeben ist, die aus der Zytoplasmamembran der Wirtszelle abgeschnürt wurde und mit viralen Glykoproteinen durchsetzt ist, sowie einen „Kern“ innerhalb des Kapsids aus weiteren Proteinen und einem Ribonukleinsäure-Komplex.
Genom
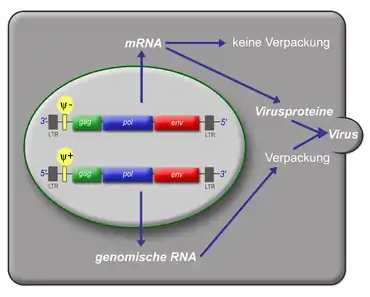
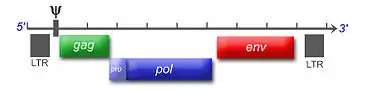
Das einzelsträngige RNA-Genom der Retroviren ist linear und 7–12 Kilobasenpaare (kb) groß. Retroviren sind die einzigen RNA-Viren, die diploid angelegt sind, d. h. jedes Retrovirus hat eine zusätzliche Kopie seines Genoms. Sie werden von den wirtseigenen Transkriptions-Enzymen übersetzt und synthetisiert und benötigen eine spezifische zelluläre tRNA. Das provirale Genom eines einfachen Retrovirus enthält in der Regel drei Gene und zwei Long Terminal Repeats (LTRs), die sich am Anfang und am Ende befinden und Informationen zur Steuerung der Expression der viralen Gene enthalten. Bei den drei Genen handelt es sich um gag (Gruppenspezifisches Antigen), pol (Polymerase) und env (envelope). gag codiert die Matrix-, Kapsid- und Nukleokapsidproteine. pol codiert die viralen Enzyme Protease, Reverse Transkriptase (mit RNase H) und Integrase. Bei den Beta- und Deltaretroviren hat die Protease ein eigenes Leseraster (pro) und bei den Alpharetroviren befindet sich die Information für die Protease im gag-Gen. env codiert die Proteine der Hülle. An regulatorischen Sequenzen gibt es im 5'-Bereich eine mit ψ (psi) bezeichnete Sequenz, die ein Signal für das Verpacken der RNA in die Viruspartikel ist, eine Primerbindungsstelle (PBS), an die sich die jeweilige tRNA anlagern kann und einen Promotor. Im 3'-Bereich finden sich ein oder mehr Polypurintrakte, die bei der reversen Transkription essentiell sind.
Komplexe Retroviren, wie z. B. das zu den Lentiviren gehörende HI-Virus, das zu den Deltaretroviren gehörende HTLV oder die Foamyviren enthalten noch weitere, regulatorische Gene, die als akzessorische Gene bezeichnet werden. Bei HIV-1 sind dies tat, rev, vif, nef, vpu und vpr, bei HTLV-1 rex, rof, tax, tof und bei den Foamyviren tas und bet.
Lebenszyklus
Der Lebenszyklus eines Retrovirus besteht aus mehreren Schritten: Infektion der Zelle, Reverse Transkription, Überwindung der Kernhülle, Integration ins Wirtsgenom, Expression der viralen Proteine und des RNA-Genoms und die Bildung neuer Viruspartikel.
Eintritt in die Wirtszelle
Nachdem das Glykoprotein der Virushülle spezifisch an seine(n) zellulären Rezeptor(en) gebunden hat, verschmilzt die virale Membran mit der Membran der Zelle und entlässt das Kapsid in das Innere der Zelle, das Cytoplasma. Was mit dem Kapsid im Cytoplasma geschieht, ist noch nicht in allen Einzelheiten geklärt, vermutlich zerfällt es in seine einzelnen Bausteine und gibt die beiden ssRNA-Stränge und die im Inneren enthaltenen Proteine, wie zum Beispiel die Reverse Transkriptase ins Cytoplasma der Wirtszelle frei.
Reverse Transkription
Hauptartikel: Reverse Transkriptase
Retroviren sind die einzigen einsträngig-plusstrangorientierten RNA-Viren, bei denen das Genom nicht sofort als Matrize (mRNA) bei der Infektion benutzt werden kann: Wenn das Virus diese RNA in die zu befallende Zelle eingebracht hat, muss die RNA in doppelsträngige DNA umgeschrieben werden. Dieser Vorgang wird reverse Transkription genannt. Dazu bringt das Virus die Reverse Transkriptase in seinen Viruspartikeln mit. Diese schreibt die einzelsträngige RNA des Virus zuerst in einzelsträngige DNA und anschließend in doppelsträngige DNA um. Bei der reversen Transkription werden die beiden LTR-Sequenzen generiert, die für den weiteren Ablauf der Infektion essenziell sind.
Normalerweise verläuft die Transkription an der DNA als Matrize, wobei ein komplementärer RNA-Strang synthetisiert wird; eine Ausnahme stellen die Retroviren und die Retroelemente (auch Klasse-I-Transposons genannt) dar. Da der Prozess durch die fehlende Korrekturlese-Fähigkeit der Reversen Transkriptase relativ ungenau ist, erfolgen häufige Mutationen des Virus. Diese ermöglichen eine schnelle Anpassung des Virus an antivirale Medikamente und damit eine Ausbildung von Resistenzen.
Überwindung der Kernhülle
Einige Gattungen der Retroviren, beispielsweise Gammaretroviren, können die Kernhülle nicht aktiv überwinden. Sie infizieren daher nur Zellen, die sich in Teilung befinden und nutzen den Moment der Zellteilung zur Integration, wenn das Genom nicht durch die Kernhülle geschützt ist. Andere Gattungen wie beispielsweise die Alpharetroviren und vor allem die Lentiviren können die Kernhülle aktiv überwinden und damit auch ruhende Zellen infizieren. Der Eintritt in den Zellkern erfolgt nach der Bildung des Präintegrationskomplexes (PIC) im Cytoplasma. Da die Kernporen kleiner sind als der PIC, der etwa die Größe eines Ribosoms hat, muss es sich um einen aktiven Transportprozess handeln. An diesem noch nicht vollständig verstandenen Prozess sind sowohl zelluläre als auch virale Proteine beteiligt. Ein weithin akzeptiertes Modell beschreibt den Eintritt in den Zellkern durch die Kernporen mit Hilfe von karyophilen Signalen der im PIC enthaltenen Proteine.[5]
Integration ins Wirtsgenom
Hauptartikel: Integrase
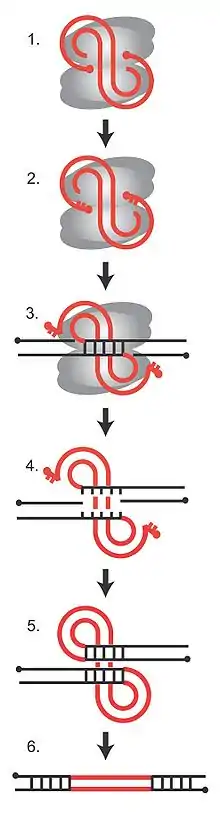
Die Integration des viralen Genoms in das Wirtsgenom ist ein essentieller Schritt im Replikationszyklus des Virus. Er wird katalysiert durch ein Enzym namens Integrase, das in allen Retroviren und Retrotransposons vorkommt. Die Integrase bindet an die virale und die Wirts-DNA und bildet mit diesen einen Komplex, der als Präintegrationskomplex (PIC, von engl. preintegration complex) bezeichnet wird. Theoretisch kann die Integration an jedem Ort im Wirtsgenom erfolgen, je nach Art des Retrovirus zeigen sich jedoch bestimmte bevorzugte chromosomale Bereiche. Was genau den Ort der Integration beeinflusst, ist noch nicht völlig geklärt, sicher ist jedoch, dass die Aminosäuresequenz der Integrase eine Rolle spielt, wodurch sich spezifische Interaktionsmöglichkeiten zwischen Integrase und Faktoren im Chromatin ergeben. In der Abbildung rechts ist die Abfolge der DNA-Strangbrüche und anschließender Neuzusammensetzung bei der Integration des viralen Genoms dargestellt. In grau sind die Integrase-Monomere, in rot die virale DNA und in schwarz die Wirts-DNA abgebildet, die roten Punkte schließlich die 5'-Enden der viralen DNA:
- Die virale cDNA ist an die Integrase gebunden und Teil des Präintegrationskomplexes.
- Die Integrase entfernt zwei Nukleotide von den 3'-Enden der viralen DNA, so dass 5'-Überhänge entstehen.
- Die Integrase schneidet die Wirts-DNA an einer zufälligen Stelle, so dass 5'-Überhänge von 4–6 Nukleotiden entstehen, und verbindet die verkürzten 3'-Enden der viralen DNA mit der Wirts-DNA.
- Die Basenpaarung der Wirts-DNA wird im betroffenen Bereich aufgelöst.
- DNA-Reparaturenzyme ergänzen die 4–6 Basen des jeweils anderen Strangs der Wirts-DNA und Ligasen verbinden ihre beiden noch freien Enden schließlich mit denen der viralen DNA.
- Während des Vorgangs verliert das Virusgenom die beiden endständigen Nukleotide, die 4–6 Basen der Wirts-DNA dagegen werden verdoppelt und flankieren anschließend das entstandene Provirus.
Expression und Partikelbildung
Nach der Integration werden zelluläre Transkriptionsfaktoren und die RNA-Polymerase II der Zelle rekrutiert, um die Provirus-DNA zu transkribieren. Die dafür notwendigen Promotor- und Enhancer-Strukturen liegen in der LTR des Provirus. Bei komplexen Retroviren wirken manche viralen Proteine (z. B. tat) zusätzlich als Transkriptionsverstärker. Es entstehen verschiedene mRNAs durch alternatives Spleißen.
Die entstehenden mRNAs werden ins Cytoplasma transportiert. Dort werden die verschiedenen viralen Proteine translatiert. Die Synthese der Env-Proteine erfolgt an der Membran des Endoplasmatischen Retikulums, so dass die Env-Proteine direkt in der Zellmembran verankert werden, wo sie sich zu Trimeren zusammenlagern. Alle anderen Virusproteine werden an freien Ribosomen synthetisiert. Die Gag- und Gag/Pol-Vorläuferproteine werden aminoterminal myristyliert und lagern sich an der Zellmembran an. An der Zellmembran findet dann die Partikelbildung statt: Gag- und Gag/Pol-Vorläuferproteine akkumulieren sich und interagieren außer miteinander auch mit Glykoproteinen der Zelle. Nur die ungespleißten mRNAs, von denen Gag und Pol translatiert wurden, besitzen das Verpackungssignal Psi und die Leader-Sequenz. Mit Hilfe des Psi-Signals binden die mRNAs an die Zinkfingermotive der Nucleokapsidproteine- so wird sichergestellt, dass nur ungespleißte und damit vollständige Genome in das Viruspartikel verpackt werden. Bei Kontakt mit den mRNAs stülpt sich die Membran an der Zelloberfläche ein und schnürt ein unreifes Viruspartikel ab. Erst innerhalb dieses Partikels beginnt die virale Protease, sich zu Dimeren zusammenzulagern, sich in einem autokatalytischen Schnitt aus den Vorläuferproteinen herauszuschneiden und dann die Gag- und Gag/Pol-Vorläuferproteine in die einzelnen Komponenten (Matrix-, Kapsid-, Nukleokapsid, Reverse Transkriptase und Integrase) zu spalten. Innerhalb des Partikels lagern sich die Kapsidproteine zu einem konischen Kapsid zusammen. Erst am Ende dieses Reifungsprozesses ist der Partikel infektiös.
Endogene Retroviren und evolutionäre Entwicklung
Hauptartikel: Retroelement, Retrotransposon, Endogenes Retrovirus
Der genaue Entstehungszeitpunkt der ersten retrovirusartigen Partikel ist unklar, die ältesten Sequenzen legen ein Alter von mindestens 250 Millionen Jahren nahe, vermutlich sind sie noch wesentlich älter.[6] Retroviren haben sich wahrscheinlich aus Retrotransposons entwickelt. Das bedeutet, dass sie eine infektiöse, weiterentwickelte Form der Retroelemente darstellen. Demnach wäre die reverse Transkription einer der ältesten Mechanismen in der Retrovirusentwicklung, der möglicherweise bereits in der RNA-Welt entstanden ist. Große Ähnlichkeit besteht jedenfalls zwischen Retroviren und den Transposons aus verschiedenen Lebewesen, beispielsweise den Ty-Elementen der Bäckerhefe und den Copia- und Ulysses-Elementen aus Drosophila melanogaster. Diese Retrotransposons codieren eine Reverse Transkriptase und haben eine ähnliche Struktur wie die Retroviren.
Hinweise, dass die Organisation der DNA bei Bakterien fundamental von der bei Archaeen und komplexen Zellen (Eukaryoten) abweicht, geben Anlass zu der These, dass deren zelluläre Urvorfahren (LUGA oder LUCA) noch der RNA-Welt angehört haben. Die Speicherung der Erbinformation in der DNA wird dann als eine Fähigkeit angesehen, die zunächst von Retroviren ‚erfunden‘ wurde und die dann zelluläre Organismen mehrmals durch Übertragung von solchen erworben haben. Daraus resultieren die Bakterien einerseits, die Archaeen und Eukaryoten andererseits. (Der grundsätzliche Aufbau der Ribosomen als Eiweißfabriken und der Genetische Code stimmen dagegen bei allen zellulären Organismen so gut überein, dass bereits der LUCA darüber verfügt haben muss.)[7]
Die Integration in das Genom ihres Wirts ist eines der ungewöhnlichsten und bemerkenswertesten Merkmale von Retroviren. Die Vielzahl an ähnlichen Sequenzen in Wirbeltieren und Retroviren zeigt, dass Retroviren in der Vergangenheit schon sehr oft auch die Zellen der Keimbahn ihrer Wirte infiziert haben. Derart an die Nachkommen vererbte Retroviren werden als endogene Retroviren (ERV) bezeichnet, um sie von den horizontal weitergegebenen, exogenen Retroviren zu unterscheiden. Durch die zunehmende Zahl sequenzierter Organismen wurden auch immer mehr endogene Retroviren entdeckt. Die Menge retroviraler DNA schwankt bei Wirbeltieren zwischen 5 und 10 %, das menschliche Genom besteht zu etwa 8 % aus retroviralen Sequenzen. Diese Daten geben einen Einblick in die lange Wirts-Virus-Coevolution.[8] Mit den Endogenen Retroviren wurden aus den infektiösen Viren, die aus Transposons entstanden, wieder Teile der Genome.
Bisher wurden im menschlichen Genom 31 verschiedene ERV-„Familien“ beschrieben, die wahrscheinlich auf 31 verschiedene Fälle von Keimbahninfektionen durch Retroviren zurückgehen (im Englischen als genome invasion event (Genominvasionsereignis) bezeichnet). Diesem Ausgangsereignis folgte eine Erhöhung der ERV-Kopienzahl, entweder durch Reinfektion der Keimbahnzellen oder durch Retrotransposition innerhalb der Zelle. Im Lauf der Generationen nimmt die Aktivität der ERVs immer weiter ab, da sich Mutationen ansammeln und ganze Abschnitte der ERVs verloren gehen können, bis schließlich die Aktivität der Viren ganz aufhört. Das Alter der einzelnen ERV-Familien oder ERV-Linien kann anhand der Größe und Form der Phylogenetischen Stammbäume der Linien abgeschätzt werden. Die meisten der humanen ERV-Linien (HERV) entstanden demnach vor der evolutionären Entwicklung von Altwelt- und Neuweltaffen vor etwa 25 bis 30 Millionen Jahren.
Durch Retroviren verursachte Krankheiten
Von Retroviren werden sehr viele verschiedene Lebewesen infiziert. Die betroffenen Spezies reichen von Muscheln bis zum Menschen, wobei die meisten unter den Wirbeltieren zu finden sind. Retroviren verursachen in ihren Wirten eine große Zahl verschiedenartiger Krankheiten, darunter Tumore (Lymphome, Sarkome), Neurologische Erkrankungen und Immunschwächen. Einige dieser Erkrankungen verursachen große Schäden in der Landwirtschaft, weil Nutztiere betroffen sind, oder sind die Ursache für menschliche Pandemien (AIDS). Andere Infektionen bleiben symptomlos, weshalb diese Retroviren als apathogen angesehen werden.
Manche durch Retroviren induzierte Krankheiten von Nagetieren stellen Modellsysteme dar, anhand derer die Infektionsmechanismen der Retroviren sowie die Entstehung der durch manche Retroviren verursachten Tumoren im Detail untersucht werden können. Die moderne Tumorbiologie fußt teilweise auf Daten, die aufgrund dieser Modelle erzeugt werden konnten.
Sehr viele heute bekannte Onkogene wurden zunächst bei Experimenten mit tierischen Retroviren, die diese Onkogene in ihr Genom aufgenommen hatten, entdeckt. Beispiele sind:[9][10][11]
- SRC – Rous-Sarkom-Virus (RSV), en. Rous sarcoma virus
- ABL – Abelson Murines Leukämievirus (Ab-MLV), en. Abelson murine leukemia virus
- KRAS – Kirsten Murines Sarkomavirus (Ki-MSV), auch Kirsten Murines Leukämievirus (Ki-MLV) genannt, en. Kirsten murine sarcoma virus (andere Spezies als (Ab-)MLV)[12]
- HRAS – Harvey Murines Sarkomavirus (Ha-MuSV), en. Harvey murine sarcoma virus[13]
- KIT – HZ4-feline sarcoma virus (HZ4-FeSV)
- ERBB – Erythroblastosis Aviäres Leukosevirus (Erythroblastosis-ALV), ALV en. Avian leucosis virus
- MYC – MC29 Myelocytoma Aviäres Leukosevirus (MC29-ALV), en. Avian myelocytomatosis virus 29
- CRK – CT10 Aviäres Sarkomavirus (CT10-ASV), en. Avian sarcoma virus CT10
- …
Literatur
- Reinhard Kurth, Norbert Bannert (Hrsg.): Retroviruses: Molecular Biology, Genomics and Pathogenesis. 1. Auflage. Caister Academic Press, Norfolk/UK 2010, ISBN 978-1-904455-55-4.
- John M. Coffin, Stephen H. Hughes, Harold Elliot Varmus (Hrsg.): Retroviruses. 1. Auflage. Cold Spring Harbor Laboratory Press, 1997, ISBN 0-87969-497-1. (online zugänglich bei NCBI Bookshelf)
- Jay A. Levy (Hrsg.): The Retroviridae. Volume 1–4. Springer US, ISBN 0-306-44074-1.
- Susanne Modrow, Dietrich Falke, Uwe Truyen: Molekulare Virologie. 2. Auflage. Spektrum Akademischer Verlag, Heidelberg, 2002, ISBN 3-8274-1086-X.
Weblinks
Einzelnachweise
- ICTV: ICTV Taxonomy history: Commelina yellow mottle virus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- Aris Katzourakis, Michael Tristem, Oliver G. Pybus, Robert J. Gifford: Discovery and analysis of the first endogenous lentivirus. In: Proc Natl Acad Sci U S A. 2007 Apr 10;104(15), S. 6261–6265. PMID 17384150
- V. Ellermann, O. Bang: Experimentelle Leukämie bei Hühnern. In: Zentralbl. Bakteriol. Parasitenkd. Infectionskr. Hyg. Abt. Orig. 46, 1908, S. 595–609.
- Robin A. Weiss: The discovery of endogenous retroviruses. In: Retrovirology. 2006 Oct 3;3, S. 67. Review. PMID 17018135
- Y. Suzuki, R. Craigie: The road to chromatin - nuclear entry of retroviruses. In: Nat Rev Microbiol. 2007 Mar;5(3), S. 187–196. Review. PMID 17304248
- Patric Jern, Göran O Sperber, Jonas Blomberg: Use of Endogenous Retroviral Sequences (ERVs) and structural markers for retroviral phylogenetic inference and taxonomy. In: Retrovirology. 2005, 2, S. 50 doi:10.1186/1742-4690-2-50 PMID 16092962.
- Patrick Forterre: Evolution – Die wahre Natur der Viren. In: Spektrum der Wissenschaft. Heft 8/2017, S. 37—41 (Online-Artikel veröffentlicht am 19. Juli 2017); Anmerkung: Der Autor spricht dabei von „mehreren viralen LUCAs“, scheint die Bezeichnung „LUCA“ folglich als Synonym für LCA/MRCA zu verwenden
- R. J. Gifford: Evolution at the host-retrovirus interface. In: Bioessays. 2006 Dec;28(12), S. 1153–1156. PMID 17117481
- J. M. Coffin, S. H. Hughes, H. E. Varmus: Retroviruses. CSHL Press, 1997, Kapitel Oncogenesis ISBN 0-87969-497-1, S. 482–484.
- Harold E. Varmus: Chancen und Probleme der personalisierten Krebstherapie, in: Lindauer Nobelpreisträgertagungen - The Lindau Nobel Laureate Meetings
- Karl-Henning Kalland, Xi-Song Ke, Anne Margrete Øyan: Tumour virology – history, status and future challenges. In: APMIS, Volume 117, Issue 5–6, May/June 2009, S. 382–399, doi:10.1111/j.1600-0463.2009.02452.x
- DJ Maudsley, AG Morris: Kirsten murine sarcoma virus abolishes interferon gamma-induced class II but not class I major histocompatibility antigen expression in a murine fibroblast line. In: J Exp Med., 1988 Feb 1, 167(2), S. 706–711, PMID 2831293
- ICTV: ICTV MSL including all taxa updates since the 2017 release.(MSL #33)