Polyomaviridae
Die Familie Polyomaviridae umfasst unbehüllte DNA-Viren, die bei verschiedenen Wirbeltieren (Säugetiere, Nagetiere und Vögel) und beim Menschen zu persistierenden Infektionen führen. Diese und die nahestehende Familie Papillomaviridae entstanden durch Aufspaltung der ehemaligen Familie Papovaviridae wurden vom International Committee on Taxonomy of Viruses (ICTV) im März 2020 als Papovaviricetes in den Rang einer Klasse erhoben, um die beiden Familien der Papovaviren zusammenzufassen.[1] Die ursprünglich einzige Gattung Polyomavirus wurde schon vorher ebenso aufgeteilt in vier Gattungen Alphapolyomavirus bis Deltapolyomavirus (s. u.). Der Name der Familie setzt sich aus dem griechischen πολύς (poly: viel, mehrere) und dem Suffix -oma aus der Benennung für Tumoren ab, da das erste identifizierte Virus der Familie, das Murine Polyomavirus, bei neugeborenen Mäusen zu verschiedenen Tumoren führt.
| Polyomaviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
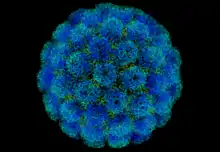
3D-Modell des SV40-Kapsids | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Polyomaviridae | ||||||||||||||
| Links | ||||||||||||||
|
2013 wurden Hybride beschrieben zwischen Papillomaviren und Polyomaviren.[2]
Morphologie
Die Virionen (Viruspartikel) der Polyomaviren bestehen aus einem nackten, etwa 40 bis 45 nm im Durchmesser großen Kapsid, das aus 72 Kapsomeren zusammengesetzt ist. Die Kapsomere sind in einer ikosaedrischen Symmetrie angeordnet (T=7). Die einzelnen Kapsomere werden an der Basis aus fünf Molekülen des Kapsidproteins VP1 gebildet (Pentamer), die jedoch zueinander nicht gleichförmig, sondern verdreht (windschief) angeordnet sind. Man spricht daher auch von einer verdrehten, ikosaedrischen Symmetrie (T=7d). An der Innenseite des Kapsids stabilisieren die Kapsidproteine VP2 und VP3 das VP1-Gerüst; sie interagieren auch mit der dsDNA im Inneren des Kapsids. Häufig werden abweichende Viruspartikel beobachtet, darunter leere normal strukturierte Kapside, sehr kleine, leere Kapside (Mikrokapside) und unregelmäßige röhrenförmige Strukturen, die aus den Kapsidproteinen in unterschiedlicher Zusammensetzung gebildet werden. Das VP1 Kapsidprotein kann sich ohne weitere Virusproteine spontan zu Virus-ähnlichen Partikeln zusammenlagern, die jedoch keine Nukleinsäure verpacken können. Im echten Virion macht das VP1 rund 70 % des gesamten Proteingehaltes aus.
Im Inneren der Kapside befindet sich der kovalent geschlossene DNA-Ring des Virusgenoms. Dieser ist wie bei den Papillomaviridae mehrfach verdrillt („supercoiled“) und bildet zusammen mit zellulären Histonen einen Nukleoproteinkomplex, der den eukaryotischen Nukleosomen strukturell sehr ähnelt. Von den fünf bekannten Histonen findet man die Histone H2a, H2b, H3 und H4.
Die Kapside sind sehr umweltstabil und können mit Diethylether, 2-Propanol oder Detergenzien (Seife) nicht inaktiviert werden. Sie sind hitzestabil bis 50 °C für 1 Stunde; bei gleichzeitiger Gegenwart von Magnesiumchlorid in 1 M Konzentration, sind die Kapside instabil, was ähnlich wie bei den Papillomviren auf die Abhängigkeit der Kapsidstruktur von zweiwertigen Kationen hindeutet.
Genom
Das Genom der Polyomaviren besteht aus einem einzelnen Molekül eines doppelsträngigen, kovalent geschlossenen DNA-Rings. Von einer nichtcodierenden, regulatorischen Region ausgehend sind die Offenen Leserahmen (ORFs) für die 5 bis 9 verschiedenen Virusproteine so angeordnet, dass die ORFs für die frühen Transkripte in einer Leserichtung laufen, die der späten Transkripte in die entgegengesetzte Richtung. Zu den frühen Transkripten gehören das große und eventuell auch kleine T-Antigen sowie andere regulatorische Proteine; die späten Transkripte codieren für die drei Strukturproteine VP1-3. Die Leserahmen überlappen sich mit teilweise verschiedenen Leserastern, so dass die Polyomaviren mit einer Genomgröße von etwa 4,7 bis 5,5 kBp für eine relativ hohe Zahl an Proteinen codieren. Wie die Papillomviren besitzen die Polyomaviren keine eigene DNA-Polymerase zur Vermehrung der viralen DNA, sie sind ebenfalls auf zelleigene Polymerasen angewiesen. Die frühen Virusproteine binden an die Enhancer und Promotoren für ihre eigenen Leserahmen in der regulatorischen Region. Dadurch wird im Laufe der Virusvermehrung die Produktion dieser Proteine zugunsten der späten Proteine unterdrückt. Die Untersuchung dieses Mechanismus beim SV-40 führte zur Entdeckung dieser regulatorischen Sequenzen bei Eukaryoten und zur Entwicklung des Enhancer-Konzeptes in der Molekularbiologie.
Biologische Bedeutung
Aviäre Polyomaviren lösen beispielsweise die Französische Mauser aus. Das BK-Virus (BKV, BK-Polyomavirus oder BKPyV) kann beim Menschen bei immunsuppressiver Behandlung nach Nierentransplantation zum Verlust des Transplantates führen. Das BK-Virus kann außerdem zur Infektion der Atemwege oder Zystitis bei Kindern führen, es kann eine hämorrhagische Zystitis bei Knochenmarkstransplantierten, eine Ureterstenose bei Nierentransplantierten und eine Meningoencephalitis bei AIDS-Patienten hervorrufen.
Die zu dieser Gattung gehörenden BK- und JC-Viren, die auch als Humanes Polyomavirus 1 und 2 (früher Polyomavirus hominis Typ 1 und 2) bezeichnet werden, persistieren im Nierengewebe; in der Normalbevölkerung können Antikörper gegen BK-Virus (BKV) zu 100 % und gegen JC-Virus (JCV) zu etwa 80 % nachgewiesen werden.
Die Tatsache, dass bei nicht erheblich vorgeschädigten Menschen und bei nicht erfolgter Doppelinfektion oder Sekundärinfektion eine Infektion mit diesen Viren nur extrem selten einen tödlichen Verlauf nimmt, zeigt zum Einen, dass diese krankheitsauslösenden Viren sehr stark an den Menschen als ihren Reservoirwirt angepasst sind. Die Schädigung seines Reservoirwirts ist für ein Virus kein vorteilhafter Effekt, da er zur eigenen Vermehrung auf diesen angewiesen ist. Die dennoch von diesem Virus beim Reservoirwirt ausgelösten Erkrankungen sind letztlich nur Nebeneffekte der Infektion. Zum Zweiten wird dadurch auch deutlich, dass sich der Mensch ebenfalls im Verlaufe vieler Generationen an dieses Virus anpassen konnte. Es besteht im Moment eine BK-Virus Durchseuchung der Bevölkerung von 80-90 %. Das JC-Virus führt bei zellulär Immunsupprimierten (AIDS St. C3) zur progressiv multifokalen Leukoenzephalopathie (PML). Die PML verläuft fast immer tödlich.
Das Simiane Virus 40 oder SV40 ist potenzieller Auslöser verschiedener Tumorerkrankungen. Teile der SV40-DNA finden in der Molekularbiologie Anwendung als besonders starker Promotor beziehungsweise Enhancer.
Systematik
Die frühere Gattung Polyomavirus wurde in vier Teile aufgespaltet, denen die Namen der griechischen Buchstaben Alpha bis Delta vorangestellt werden (ICTV Stand November 2018):[3][4][5]
- Familie Polyomaviridae
- Gattung Alphapolyomavirus[6]
- Spezies Maus-Polyomavirus
(Mus musculus polyomavirus 1, Murines Polyomavirus, alt Parotid tumor virus, MPyV) - Spezies Chlorocebus pygerythrus polyomavirus 1
(Meerkatzen-Polyomavirus-1, Vervet monkey polyomavirus 1, AGMPyV-1) - Spezies Chlorocebus pygerythrus polyomavirus 3
(Meerkatzen-Polyomavirus-3, Vervet monkey polyomavirus 3, AGMPyV-3) - Spezies Papio cynocephalus polyomavirus 1
(Pavian-Polyomavirus 1, BPyV-1) - Spezies Mesocricetus auratus polyomavirus 1
(Hamster-Polyomavirus, HaPyV) - Spezies Pan troglodytes polyomavirus 1 bis 7
(Schimpansen-Polyomavirus 1 bis 7) - Spezies Mus musculus polyomavirus 1 (Murines Polyomavirus, MPyV)
- Spezies Maus-Polyomavirus
- Gattung Betapolyomavirus[7] (zeitweilig in Gattungen Orthopolyomavirus und Wukipolyomavirus getrennt)
- Spezies Humanes Polyomavirus 1
(Polyomavirus hominis 1, BK-Virus, en. BK polyomavirus, offiziell Human polyomavirus 1, HPyV-1, BKPyV oder BKV) - Spezies Humanes Polyomavirus 2
(Polyomavirus hominis 2, JC-Virus, en. JC polyomavirus, offiziell Human polyomavirus 2, HPyV-2, JCPyV oder JCV) - Spezies Humanes Polyomavirus 3
(Polyomavirus hominis 3, KI-Virus, en. KI polyomavirus, offiziell Human polyomavirus 3, HPyV-3, KIPyV oder KIV) - Spezies Humanes Polyomavirus 4
(Polyomavirus hominis 4, WU-Virus, en. WU Polyomavirus, offiziell Human polyomavirus 4, HPyV-4, WUPyV oder WUV) - Spezies Humanes Polyomavirus 5
(Merkelzell-Polyomavirus, MCPyV) - Spezies Macaca mulatta polyomavirus 1
(Simian-Virus 40, MmPV1 bzw. SV40) - Spezies Chlorocebus pygerythrus polyomavirus 2
(Meerkatzen-Polyomavirus-2, Vervet monkey polyomavirus 2, AGMPyV-2) - Spezies Papio cynocephalus polyomavirus 2
(Pavian-Polyomavirus 2, BPyV-2)
- Spezies Humanes Polyomavirus 1
- Gattung Gammapolyomavirus[8] (veraltet Avipolyomavirus)
- Spezies Aves polyomavirus 1
(Avianes Polyomavirus 1, Wellensittich-Polyomavirus, Budgerigar fledgling disease virus, BFPyV oder BFDV) – Polyomavirus der Nestlingskrankheit der Wellensittiche - Spezies Corvus monedula polyomavirus 1
(Krähen-Polyomavirus) - Spezies Anser anser polyomavirus 1 (Hämorrhagisches Polyomavirus der Gänse, GHPV)
- Spezies Serinus canaria polyomavirus 1
(Finken-Polyomavirus)
- Spezies Aves polyomavirus 1
- Gattung Deltapolyomavirus[9]
- Spezies Humanes Polyomavirus 6 (Human polyomavirus 6, HPyV-6)
- Spezies Humanes Polyomavirus 7 (Human polyomavirus 7, HPyV-7)
- Spezies Humanes Polyomavirus 10 (Human polyomavirus 10, HPyV-10)
- Spezies Humanes Polyomavirus 11 (Human polyomavirus 11, HPyV-11)
- Keiner Gattung innerhalb der Polyomaviridae zugeordnet sind
- Spezies Bos taurus polyomavirus 1
(Bovines Polyomavirus, BPyV) - Spezies Kaninchen-Polyomavirus
(Rabbit kidney vacuolating virus, RKV) - Spezies Murines Pneumotropes Virus (MPtV)
- Spezies Baboon polyomavirus 1
(Simian-Virus 12, SV12)
- Spezies Bos taurus polyomavirus 1
Literatur
- J. Hou et al.: Family Polyomaviridae. In: C. M. Fauquet, M. A. Mayo et al.: Eighth Report of the International Committee on Taxonomy of Viruses. London / San Diego 2005, ISBN 0-12-249951-4, S. 231–238
- M. J. Imperiale, E. O. Major: Polyomaviridae. In: David M. Knipe, Peter M. Howley (eds.-in-chief): Fields’ Virology. Band 2. 5. Auflage. Philadelphia 2007, ISBN 0-7817-6060-7, S. 2263–2298
Weblinks
- Polyomaviridae (NCBI)
Einzelnachweise
- ICTV: ICTV Taxonomy history: Human polyomavirus 1, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- Annabel Rector, Marc Van Ranst: Animal papillomaviruses, Virology Band 445, Ausgabe 1–2, Oktober 2013, S. 213-223, doi:10.1016/j.virol.2013.05.007
- José Carlos Mann Prado, Telma Alves Monezi, Aline Teixeira Amorim, Vanesca Lino, Andressa Paladino, Enrique Boccardo: Human polyomaviruses and cancer: an overview. In: Clinics (Sao Paulo), 73(Suppl 1), S. e558s, doi:10.6061/clinics/2018/e558s, PMC 6157077 (freier Volltext), PMID 30328951 (online 26. September 2018), Fig. 1
- Ugo Moens, Sébastien Calvignac-Spencer, Chris Lauber, Torbjörn Ramqvist, Mariet C. W. Feltkamp, Matthew D. Daugherty, Ernst J. Verschoor, Bernhard Ehlers: ICTV Virus Taxonomy Profile: Polyomaviridae. In: J Gen Virol., 98(6), 22. Juni 2017, S. 1159–1160, doi:10.1099/jgv.0.000839, PMC 5656788 (freier Volltext), PMID 28640744
- dsDNA Viruses > Polyomaviridae. In: ICTV Report, Juni 2017, überarbeitet im Juli 2018, Tabelle 2A
- SIB: Alphapolyomavirus, auf: ViralZone
- SIB: Betapolyomavirus, auf: ViralZone
- SIB: Gammapolyomavirus, auf: ViralZone
- SIB: Deltapolyomavirus, auf: ViralZone