mRNA
Eine mRNA oder messenger-RNA[1] (englisch messenger ribonucleic acid), zu deutsch Boten-Ribonukleinsäure (auch Boten-RNS oder Boten-RNA),[2] ist eine einzelsträngige Ribonukleinsäure (RNA), die genetische Information für den Aufbau eines bestimmten Proteins in einer Zelle überträgt.
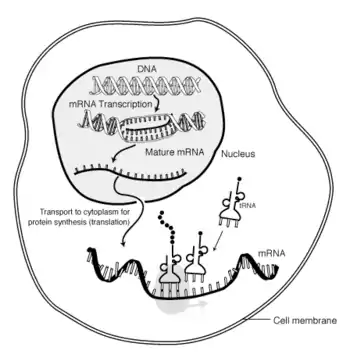
Danach wird die mRNA über Kernporen in das Cytoplasma exportiert. Dort findet die Proteinbiosynthese an Ribosomen statt. Hierbei wird die Nukleotidsequenz der mRNA mittels tRNAs in die Aminosäuresequenz des gebildeten Polypeptids übersetzt (Translation).
Eine mRNA kann mehrfach verwendet werden; schließlich wird sie abgebaut.
Zellen verändern Gestalt und Vorgänge durch ihre Proteine, die aus verschieden langen Ketten von bestimmten Aminosäuren in unterschiedlicher Reihenfolge bestehen. Die jeweilige Reihenfolge von Aminosäuren ist nicht zufällig, sondern wird als Bauplan vorgegeben. Die entsprechenden Vorlagen sind ein wesentlicher Teil der Erbinformation und liegen gespeichert in der Form von Desoxyribonukleinsäure (DNA) vor, jeweils in einem bestimmten DNA-Abschnitt (Gen) als Nukleotidsequenz. Beim Zugriff auf diese Information wird eine DNA-Sequenz abgelesen und in eine RNA-Sequenz umgeschrieben. Ein fertiges mRNA-Molekül trägt damit die Botschaft von der DNA – die bei Eukaryoten im Zellkern liegt – für den Proteinaufbau im Zellplasma und dient an den Ribosomen jeweils als Bauanleitung für ein bestimmtes Protein.
Grundlegend für solche Bauanweisungen ist die Reihenfolge von Nukleinbasen (Basensequenz) in den Genabschnitten der DNA-Moleküle. Damit die Information zum Ausdruck kommen kann (Genexpression), muss sie in die Sequenz der Nukleotide von mRNA-Molekülen umgeschrieben werden (Transkription). Hierbei wird anhand der DNA-Matrize ein RNA-Strang erstellt; dies geschieht unter enzymatischer Wirkung einer RNA-Polymerase. Als Botschaft einer mRNA kann die nun in deren Basensequenz enthaltene Information an Ribosomen abgelesen und mithilfe von tRNA-Molekülen übersetzt werden (Translation). Bei dieser ribosomalen Proteinbiosynthese gibt die codierende Basensequenz der mRNA dann die Reihenfolge von Aminosäuren (Aminosäurensequenz) in der neuaufgebauten Polypeptidkette vor – und damit die Primärstruktur eines bestimmten Proteins.
Bei Medikamenten, die auf mRNA basieren, produzieren Zellen nach der hierdurch eingebrachten Information dann das jeweils wirksame Protein. Dieses kann zum Beispiel bei mRNA-Impfstoffen als Antigen wirken.
Transkription
Bei der Transkription wird ein Abschnitt der DNA in einen RNA-Einzelstrang umgeschrieben. Dies geschieht unter Wirkung des Enzyms RNA-Polymerase. Der codogene DNA-Strang dient dabei als Matrize für den Aufbau eines RNA-Strangs, der dann als mRNA für Protein codiert. Bei Prokaryoten (Lebewesen ohne Zellkern) läuft dieser Vorgang der Transkription im Cytoplasma ab, bei Eukaryoten (Lebewesen mit Zellkern) wird das nukleäre Genom im Karyoplasma des Zellkerns einer Zelle transkribiert.
Der eigentliche Transkriptionsprozess durch eine RNA-Polymerase ist bei Prokaryoten und Eukaryoten der gleiche. Doch können in prokaryotischen Zellen (Prozyten) an der noch nicht vollständig synthetisierten mRNA schon Ribosomen ansetzen und mit der Translation beginnen. So kann noch während der Transkription zugleich die Synthese von Proteinen starten, was besondere Formen der Genregulation ermöglicht (siehe Attenuation). Bei Eukaryoten dagegen wird das primäre RNA-Transkript (prä-mRNA) zuvor noch im Zellkern verschiedenen Prozessen unterzogen (prozessiert, siehe unten) und erst danach als mRNA aus dem Kern exportiert ins Cytoplasma, wo sich die Ribosomen befinden.
Prokaryoten besitzen nur eine Art des Enzyms RNA-Polymerase für den Aufbau eines RNA-Polynukleotids. Eukaryoten verfügen dagegen über verschiedene Typen von RNA-Polymerasen, ihre RNA-Polymerase II katalysiert die Synthese von prä-mRNA.
| Enzym | Funktionelle RNA |
|---|---|
| RNA-Polymerase I | rRNA |
| RNA-Polymerase II | prä-mRNA (u. a.) |
| RNA-Polymerase III | tRNA (u. a.) |
| RNA-Polymerase IV, V | siRNA |
Ein wesentlicher Unterschied zwischen prokaryotischer und eukaryotischer mRNA besteht darin, dass prokaryotische mRNA zumeist polycistronisch ist, während eukaryotische mRNA gewöhnlich monocistronisch ist. Damit ist es Prokaryoten möglich, auf nur einem einzigen mRNA-Transkript die Informationen von mehreren – auf der DNA hintereinanderliegenden – Genen zu haben, und noch während der mRNA-Synthese (Transkription) bereits mit der Synthese der codierten Proteine zu beginnen (Translation). Ein solcher gemeinsam transkribierter Bereich auf der DNA, dessen Gene funktionell zusammengehören, heißt Operon.
Eukaryotische prä-mRNA-Prozessierung

In eukaryotischen Zellen entsteht eine reife mRNA durch Prozessierung ihrer Vorstufe, der hnRNA (englisch heterogeneous nuclear RNA) oder prä-mRNA (englisch precursor messenger RNA, pre-mRNA). Diese Prozessschritte laufen noch im Zellkern ab – erst danach kann die mRNA durch Kernporen ins Cytoplasma gelangen, wo dann an Ribosomen die Proteinbiosynthese stattfindet.
- Am 5′-Ende, es wird bei der Transkription zuerst synthetisiert, bekommt die RNA eine 5′-Cap-Struktur (englisch cap „Kappe“). Diese Kappe besteht aus einer modifizierten Form des Guanosins, dem 7-Methylguanosin (m7G). Die Cap-Struktur schützt die RNA vor dem Abbau durch Nukleasen und erlaubt den Cap-Binding-Komplex, der u. a. für den Kernexport wichtig ist. Auch nach dem Transport ins Cytosol spielt die 5′-Cap eine Rolle beim Erkennen der mRNA durch die kleine ribosomale Untereinheit und somit für den Beginn der Translation.
- Am 3′-Ende, bei der Transkription zuletzt synthetisiert, erfährt die RNA eine Polyadenylierung. Bei diesem Vorgang wird ein Poly-A-Schwanz aus 30 bis 200 Adenin-Nukleotiden angehängt. Dieser schützt die mRNA ebenfalls vor einem enzymatischen Abbau. Zusätzlich erleichtert er – auch durch Interaktionen mit der Kappe am anderen Ende – sowohl den Kernexport wie auch die Translatierbarkeit der mRNA.
- Durch Spleißen (englisch splicing) werden aus dem ursprünglichen Transkript (hnRNA) bestimmte RNA-Abschnitte als Introns entfernt, die so zur codierenden Information nicht mehr beitragen; die verbleibenden Abschnitte werden dabei miteinander verbunden und stellen dann als Exons die jeweils exprimierte genetische Information dar. Dieser Vorgang findet im Spleißosom statt, einem Komplex aus der hnRNA und den snRNPs (small nuclear Ribonucleoprotein Particle; gesprochen snurp) – bestehend aus den snRNAs U1, U2, U4, U5 und U6 und etwa 50 Proteinen – sowie weiteren Spleißfaktoren. Durch alternatives Splicing können damit aus derselben hnRNA unterschiedliche mRNAs entstehen, die translatiert auch zu unterschiedlichen Proteinen führen können.
An dieser Stelle greifen auch diverse Regulationsprozesse der Zelle ein. Über Antisense-RNA und RNA-Interferenz kann mRNA abgebaut werden und so die Translation verhindert werden. Weiterhin erfolgt in einigen Fällen die sogenannte RNA-Edition, wodurch Nukleotide in einer mRNA verändert werden. Ein Beispiel dafür ist die mRNA des Apolipoprotein B, die in manchen Geweben editiert wird, in anderen nicht. Durch die Edition entsteht hier ein zweites Stopcodon upstream, das bei der Translation zu einem kürzeren Protein (mit einer anderen Funktion) führt.
Translation
Bei der Translation wird die codierende Sequenz von Nukleinbasen der mRNA-Nukleotide in die Aminosäuresequenz der Polypeptidkette eines Proteins übersetzt (translatiert). Die Nukleotidsequenz eines Offenen Leserahmens wird hierbei in Dreierschritten abgelesen und jedem Basentriplett mittels tRNA-Molekülen jeweils eine bestimmte Aminosäure zugeordnet sowie diese mit der vorangegangenen über eine Peptidbindung verknüpft. Dieser Prozess findet an den Ribosomen im Cytoplasma statt und stellt die eigentliche Proteinbiosynthese dar. Bei eukaryotischen Zellen können die Ribosomen frei vorliegen oder sich der Membran des Endoplasmatischen Retikulums anheften.
Jedes Ribosom, das eine mRNA erkennt und an diese bindet, translatiert dann die codierende Nukleotidsequenz der mRNA gemäß dem genetischen Code in die entsprechende Aminosäurensequenz eines Proteins. Die hierfür nötigen Aminosäuremoleküle werden von tRNA-Molekülen herangetragen und dem Cytoplasma der Zelle entnommen. Während eine prokaryotische mRNA oft mehrere codierende Abschnitte enthält (polygenische mRNA), sind eukaryotische mRNAs gewöhnlich monocistronisch und enthalten so nur einen Abschnitt mit codierender Sequenz.
Ein Ribosom übersetzt jeweils nur eine mRNA und baut nach deren Vorgabe das codierte Polypeptid einmal auf. Danach löst sich das Ribosom von der mRNA. An eine mRNA können sich aber mehrere Ribosomen zugleich anlagern und jeweils eine Polypeptidkette synthetisieren. Ebenso kann eine mRNA mehrmals nacheinander ribosomal abgelesen werden. Die Anzahl der gebildeten Proteinmoleküle hängt also von der Zahl an durchgeführten Translationsvorgängen ab. Je länger ein mRNA-Molekül existiert, desto häufiger kann es hierfür benutzt werden.
Degradation
Später wird die mRNA durch eine Ribonuklease (RNase) enzymatisch abgebaut und in ihre Nukleotide zerlegt, die dann wieder zum Aufbau neuer RNA-Moleküle genutzt werden können. Mit dieser Zersetzung, auch Degradation genannt, endet die Lebensdauer eines mRNA-Moleküls. Die Dauer der Verfügbarkeit bis zum Abbau durch Nukleasen in der Zelle kann jedoch verschieden sein und ist für eine Regulation der Proteinbiosynthese bedeutsam. Der Abbauprozess findet in eukaryotischen Zellen oft in spezifischen Strukturen statt, den sogenannten P-bodies im Cytoplasma. Da hier mRNA auch – statt abgebaut zu werden – temporär zwischengelagert werden kann für eine erneute Translation, sind damit zusätzliche Regulationsweisen möglich.
Reinigung und Nachweis
RNA kann durch eine RNA-Reinigung isoliert werden, z. B. bei RNA mit einem Poly-A-Schwanz durch Verwendung einer Oligo-dT-Säule. Der Nachweis erfolgt per Northern Blot oder nach einer RT-PCR durch eine qPCR oder eine DNA-Sequenzierung.
mRNA-Impfstoffe
Da mRNA bereits in kurzer Zeit in den Wirtszellen enzymatisch abgebaut wird und außerdem über die Aktivierung von Toll-like-Rezeptoren zu einer Zytokin-Ausschüttung mit unerwünschten Wirkungen führen kann, wurde zunächst nur zögerlich an Medikamenten auf RNA-Basis geforscht.
Bei der Anwendung als Impfstoff wird eine mRNA verabreicht, die ein ausgewähltes Protein codiert. In den Zellen des Impflings wird anhand der aufgenommenen mRNA dieses Protein ribosomal synthetisiert. Anschließend kann es auf der Zelloberfläche extrazellulär dem Immunsystem präsentiert werden und als Antigen wirken. In der Folge können darüber unterschiedliche Immunantworten ausgelöst werden. Dieses Verfahren kann für verschiedene Zwecke eingesetzt werden, zum Beispiel in der Krebstherapie[3][4][5] sowie für Influenzaimpfstoffe[6] und für Tollwutimpfstoffe.[7]
Verwendet man statt normaler mRNA hingegen modRNA (nukleosid-modifizierte mRNA), lassen sich der rasche enzymatische Abbau und die Aktivierung von Toll-like-Rezeptoren verlangsamen bzw. verhindern, was die Entwicklung von Impfstoffen auf mRNA-Basis massiv beschleunigte.
Literatur
- James E. Darnell, Harvey Lodish, David Baltimore: Molekulare Zellbiologie. de Gruyter, Berlin u. a. 1993, ISBN 3-11-011934-X (4. Auflage. Harvey Lodish: Molekulare Zellbiologie. Spektrum Akademischer Verlag, Heidelberg u. a. 2001, ISBN 3-8274-1077-0).
- Benjamin Lewin: Molekularbiologie der Gene. Spektrum Akademischer Verlag, Heidelberg u. a. 1998, ISBN 3-8274-0234-4.
- William S. Klug, Michael R. Cummings, Charlotte A. Spencer: Genetik. 8., aktualisierte Auflage 2007, ISBN 978-3-8273-7247-5.
- G. Haimovich, D. A. Medina u. a.: Gene expression is circular: factors for mRNA degradation also foster mRNA synthesis. In: Cell. Band 153, Nummer 5, Mai 2013, S. 1000–1011, doi:10.1016/j.cell.2013.05.012. PMID 23706738.
Einzelnachweise
- Eintrag messenger-RNA im Lexikon der Biologie auf Spektrum.de.
- Moleküle auf Zellreise im UniSpiegel der Universität Heidelberg, Ausgabe Januar 2002.
- B. Weide, J. P. Carralot, A. Reese, B. Scheel, T. K. Eigentler, I. Hoerr, H. G. Rammensee, C. Garbe, S. Pascolo: Results of the first phase I/II clinical vaccination trial with direct injection of mRNA. In: Journal of immunotherapy. Band 31, Nummer 2, 2008 Feb-Mar, S. 180–188, doi:10.1097/CJI.0b013e31815ce501, PMID 18481387.
- B. Weide, S. Pascolo, B. Scheel, E. Derhovanessian, A. Pflugfelder, T. K. Eigentler, G. Pawelec, I. Hoerr, H. G. Rammensee, C. Garbe: Direct injection of protamine-protected mRNA: results of a phase 1/2 vaccination trial in metastatic melanoma patients. In: Journal of immunotherapy. Band 32, Nummer 5, Juni 2009, S. 498–507, doi:10.1097/CJI.0b013e3181a00068, PMID 19609242.
- M. Fotin-Mleczek, K. M. Duchardt, C. Lorenz, R. Pfeiffer, S. Ojkić-Zrna, J. Probst, K. J. Kallen: Messenger RNA-based vaccines with dual activity induce balanced TLR-7 dependent adaptive immune responses and provide antitumor activity. In: Journal of immunotherapy. Band 34, Nummer 1, Januar 2011, S. 1–15, doi:10.1097/CJI.0b013e3181f7dbe8, PMID 21150709.
- F. B. Scorza, N. Pardi: New Kids on the Block: RNA-Based Influenza Virus Vaccines. In: Vaccines. Band 6, Nummer 2, April 2018, S. , doi:10.3390/vaccines6020020, PMID 29614788, PMC 6027361 (freier Volltext).
- N. Armbruster, E. Jasny, B. Petsch: Advances in RNA Vaccines for Preventive Indications: A Case Study of A Vaccine Against Rabies. In: Vaccines. Band 7, Nummer 4, September 2019, S. , doi:10.3390/vaccines7040132, PMID 31569785, PMC 6963972 (freier Volltext).