Autolykiviridae
Die Autolykiviridae sind eine im Jahr 2021 vom International Committee on Taxonomy of Viruses (ICTV) offiziell bestätigte Familie von dsDNA-Viren, deren Wirte Bakterien sind (was sie als Bakteriophage klassifiziert), die aber im Gegensatz zu den Caudoviricetes keinen Kopf-Schwanz-Aufbau, sondern nur ein Kapsid mit ikosaedrischer Geometrie. Sie sind Mitglieder der Viruslinie, deren Hauptkapsidprotein (MCP) eine DJR-Faltung (englisch double jelly roll fold-Struktur) zeigt. Ihre nächsten derzeit (Mitte 2021) bekannten Verwandten sind die Corticoviridae. Zu ihren näheren Verwandten gehören unter anderem auch die Adenoviridae (Adenoviren).[2][3]
| Autolykiviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
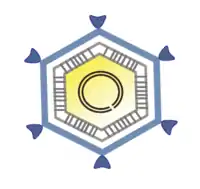
Schemazeichnung eines Virions | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Autolykiviridae | ||||||||||||||
| Links | ||||||||||||||
|
Forschungsgeschichte
Die ersten Hinweise auf Vertreter der neuen Familie fanden sich 2018 bei Metagenomanalysen von Kathryn M. Kauffman, Natalya Yutin, Disa Bäckström und Kollegen.[4][5] Bis zur offiziellen Bestätigung der Familie gelang es aber bereits, von einzelnen Vertretern TEM-Aufnahmen zu machen (siehe §Bildergalerie).[3][4]
Etymologie
Die Familie ist benannt nach der Figur des Autolykos (altgriechisch Αὐτόλυκος, latinisiert Autolycus) aus der griechischen Mythologie, einem Meister der Diebesfertigkeit, der als schwer zu fangen galt.[3] Genause schwer waren die Vertreter dieser Familie zu fassen, was ihre Entdeckung lange verzögert hatte, trotz ihrer ubiquitären Verbreitung[6] in den Ozeanen (siehe §Ökologie).[4]
Morphologie
Die Autolykiviridae gehören zu den zuvor schlecht untersuchten nicht-taxonomischen Gruppe der Bakteriophagen ohne Kopf-Schwanz-Aufbau (d. h. nicht zu den Caudoviricetes), sind aber auch nicht filamentös (wie etwa die Tubulavirales). Sie zeigen ein isometrisches (gleichförmiges) ikosaedrisches Kapsid. Seltene Aufnahmen zeigen eine Art Schwanz oder Tubulus, ähnlich wie bei den Tectiviridae, wenn sie an der Zellmembran der Wirtszelle anliegen (d. h. zu Beginn der Infektion).[4]
Genom
Die Autolykiviridae haben ein lineares ssDNA-Genom (unsegmentiert, d. h. monopartit) mit einer Länge von grob 10 kbp (Kilobasenpaare).[3]
Proteom
Die Autolykiviridae kodieren neben dem DJR-MCP eine Verpackungs-ATPase (en. packaging ATPase, für die Assemblierung d. h. den Zusammenbau der Viruspartikel) der FtsK-HerA-Superfamilie (vgl. Monodnaviria, Yaravirus) sowie für eine DNA-Polymerase mit Protein-Primer, bezeichnet als pDNApol. Diese Eigenschaften kennzeichnen sie als Mitglied der Klasse Tectiliviricetes, das pDNApol unterscheidet sie aber von ihren nächsten Verwandten, den Corticoviridae. Sie enthalten wie andere Mitglieder der Tectiliviricetes Lipidmembranen in ihren Kapsiden, zudem sind wie bei den Tectiviridae terminale Proteine kovalent an ihr Genom gebunden.[3]
Wirte und Systematik
Die derzeit (Mitte 2021) bekannten Autolykiviridae wurden in küstennahen marinen heterotrophen Bakterien der Gattung Vibrio (Vibrionen) isoliert. Tests zeigten aber, dass einige Arten der Autolykiviridae in der Lage sind, sich in mindestens 6 verschiedenen Spezies der Bakterienfamilie Vibrionaceae zu replizieren.[3]
Die Systematik (Stand ICTV MSL#36, 1. Halbjahr 2021) und Wirte sind wie folgt[2][7][3] (auf NCBI sind derzeit (13. Juni 2021) die einzelnen Stämme noch jeweils eigenen provisorischen Spezies zugeordnet und nicht in die ICTV-bestätigten offiziellen Spezies gruppiert.[8]):
Klasse: Tectiliviricetes
- Familie: Autolykiviridae mit 2 Gattungen
- Gattung: Livvievirus mit 2 Spezies
- Spezies: Livvievirus viph1249a (ehem. Gruppe E) – isoliert auf Vibrio cyclitrophicus
- Stamm: Vibrio phage 1.249.A._10N.261.55.B9 (Referenzstamm)
- Stamm: Vibrio phage 1.249.B._10N.261.55.B9
- Spezies: Paulavirus viph1044o (sic, richtiger wäre Livvievirus viph1044o, ehem. Gruppe D) – ursprünglich für die Gattung Paulavirus vorgeschlagen – isoliert auf Vibrio lentus und V. sp. F12
- Stamm: Vibrio phage 1.044.O._10N.261.51.B8 (Referenzstamm)
- Stamm: Vibrio phage 1.043.O._10N.261.52.C7
- Stamm: Vibrio phage 1.048.O._10N.286.46.A10 identisch Vibrio phage 1.102.O._10N.261.45.E3
- Stamm: Vibrio phage 1.057.O._10N.261.46.B12
- Stamm: Vibrio phage 1.062.O._10N.286.55.C3
- Stamm: Vibrio phage 1.095.O._10N.286.46.E10
- Stamm: Vibrio phage 1.107.A._10N.286.52.E10
- Stamm: Vibrio phage 1.107.B._10N.286.52.E10
- Stamm: Vibrio phage 1.107.C._10N.286.52.E10
- Gattung: Paulavirus mit 3 Spezies
- Spezies: Paulavirus viph1008o (ehem. Gruppe A) – isoliert auf Vibrio lentus, V. cyclitrophicus, und V. splendidus, kann auch Enterovibrio-Arten infizieren.
- Stamm: Vibrio phage 1.008.O._10N.286.54.E5 (Referenzstamm)
- Stamm: Vibrio phage 1.040.O._10N.286.45.B9
- Stamm: Vibrio phage 1.011.O._10N.286.49.B11
- Stamm: Vibrio phage 1.069.O._10N.286.49.F11
- Stamm: Vibrio phage 1.125.O._10N.286.49.F5
- Stamm: Vibrio phage 2.092.O._10N.286.52.B7
- Spezies: Paulavirus viph1020o (ehem. Gruppe B) – isoliert auf Vibrio tasmaniensis, infiziert auch V. lentus
- Stamm: Vibrio phage 1.020.O._10N.222.48.A2 (Referenzstamm)
- Spezies: Paulavirus viph1080o (ehem. Gruppe C) – isoliert auf Vibirio kanaloae und V. lentus.
- Stamm: Vibrio phage 1.080.O._10N.286.48.A4 (Referenzstamm)
- Stamm: Vibrio phage 1.141.A._10N.261.49.B
Obwohl die Autolykiviridae offenbar eine Schwesterfamilie der Corticoviridae darstellen, hat das ICTV die neue Familie nicht in deren Ordnung Vinavirales aufgenommen. Die Autolykiviridae verbleiben daher zunächst ohne Ordnungszuweisung, und die Corticoviridae monotypisch in ihrer Ordnung Vinavirales. Als provisorische Bezeichnung für eine beide Familien umfassende Klade wurde PM2-Gruppe (en. PM2 group) vorgeschlagen, benannt nach der Corticoviridae-Spezies Pseudoalteromonas-Phage PM2.[5]
Ökologie
Die Autolykiviren wurden als Hauptvernichter (en. principal killers) der marinen Vibrionaceen identifiziert, was auf einen viel größeren ökologischen Einfluss der schwanzlosen Bakteriophagen hinweist, als bisher angenommen.[5][6]
Bildergalerie
Offenbar gibt es zur Zeit (14. Juni 2021) noch keine gemeinfreien Bilder oder Schemazeichnungen von Autolykiviridae. Eine Reihe von TEM-Aufnahmen und Genomkarten finden sich bei den folgenden Quellen:
- Vorschlag an das ICTV, docx-Datei, Fig. 1a und b (Genomkarten der fünf 2021 bestätigten Spezies unter c)[3]
- Kauffman et al. (2018), Fig. 1a und b, sowie Extended Data Fig. 1.[4]
- David L. Chandler (2018), MIT News und Stanislav Mihulka (2018), OSEL.[6]
- Ignacio-Espinoza und Fuhrmann (2018), Schemazeichnung in Fig. 1 (rechts) und Link mit TEM-Thumbnail.[9]
- Scientists Find New Type of Virus in World’s Oceans: Autolykiviridae, auf: sci-news vom 25. Januar 2018
Weblinks
- Dann Turner, Andrew M. Kropinski, Evelien M. Adriaenssens: A Roadmap for Genome-Based Phage Taxonomy, in: MDPI Viruses, Band 13, Nr. 506, 18. März 2021, doi:10.3390/v13030506
- Sari Mäntynen, Lotta-Riina Sundberg, Hanna M. Oksanen, Minna M. Poranen: Half a Century of Research on Membrane-Containing Bacteriophages: Bringing New Concepts to Modern Virology, in: MDPI Viruses, Band 11, Nr. 76, 18. Januar 2019, doi:10.3390/v11010076
- Forscher entdecken ein mysteriöses Virus, das die Ozeane dominiert, auf: Business Insider vom 29. Januar 2018 (deutsch)
- Peter Dockrill: Never-Before-Seen Viruses With Weird DNA Were Just Discovered in The Ocean, auf: sciencealert vom 25. Januar 2018
Einzelnachweise
- Sari Mäntynen, Lotta-Riina Sundberg, Hanna M. Oksanen, Minna M. Poranen: Half a Century of Research on Membrane-Containing Bacteriophages: Bringing New Concepts to Modern Virology, in: MDPI Viruses, Band 11, Nr. 76, 18. Januar 2019, doi:10.3390/v11010076
- ICTV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- Kauffman KM, Hussain FA, Yang J, Arevalo P, Brown JM, Chang WK, VanInsberghe D, Elsherbini J, Sharma RS, Cutler MB, Kelly L, Polz MF: Official taxonomy updates – prokaryote viruses: Autolykiviridae, Create one new family (Autolykiviridae) of non-tailed dsDNA bacterial viruses in the double jelly roll fold major capsid lineage. [ZIP (docx, xlsx)]
- Kathryn M. Kauffman, Fatima A. Hussain, Joy Yang, Philip Arevalo, Julia M. Brown, William K. Chang, David VanInsberghe, Joseph Elsherbini, Radhey S. Sharma, Michael B. Cutler, Libusha Kelly, Martin F. Polz: A major lineage of non-tailed dsDNA viruses as unrecognized killers of marine bacteria, in: Nature 554, S. 118–122, 24. Januar 2018, doi:10.1038/nature25474, PMID 29364876, ResearchGate.
TEM-Aufnahmen: Siehe auch:- Karla Lant: Undercover Marine Bacteria Killers: New Lineage of Non-tailed Viruses Identified, auf: Environmental Monitor vom 28. Februar 2018
- Natalya Yutin, Disa Bäckström, Thijs J. G. Ettema, Mart Krupovic, Eugene V. Koonin: Vast diversity of prokaryotic virus genomes encoding double jelly-roll major capsid proteins uncovered by genomic and metagenomic sequence analysis, in: BMC Virology Journal, Band 15, Nr. 67, 10. April 2018, doi:10.1186/s12985-018-0974-y, PMID 29636073, PMC 5894146 (freier Volltext)
- David L. Chandler: New type of virus found in the ocean: The unusual characteristics of these abundant, bacteria-killing viruses could lead to evolutionary insights, auf: MIT News vom 24. Januar 2018. Dazu:
- Stanislav Mihulka: Nově objevené viry jsou lstiví zabijáci bakterií, auf: OSEL vom 27. Januar 2018 (tschechisch)
- ICTV: ICTV Taxonomy history: Autolykiviridae, EC 52, Online meeting, October 2020; Email ratification March 2021 (MSL #36)
- NCBI: Autolykiviridae (family)
- Julio Cesar Ignacio-Espinoza, Jed A. Fuhrman: A non-tailed twist in the viral tale, in: Nature, Band 554, S. 38–39, 24. Januar 2018, doi:10.1038/d41586-018-00923-8