Klebsiella pneumoniae
Klebsiella pneumoniae ist ein fakultativ anaerobes, gramnegatives Stäbchenbakterium aus der Gattung Klebsiella, das in der Lage ist, den Zweifachzucker Lactose (Milchzucker) abzubauen. Außerdem kann es den in der Luft vorhandenen Stickstoff zu Ammoniak bzw. Ammonium reduzieren, dieser Stoffwechselweg wird als Stickstofffixierung bezeichnet. 1984 wurde die Art in drei Unterarten (Subspezies) aufgeteilt. Das Bakterium ist überall verbreitet. Beim Menschen gehört es zu den normalen Bewohnern des Darms. In anderen Körperregionen kann es jedoch als Krankheitserreger auftreten, auch bei Tieren. Das Genom des Bakterienstammes Klebsiella pneumoniae subsp. pneumoniae DSM 30104 wurde im Jahr 2012 vollständig sequenziert.
| Klebsiella pneumoniae | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
.jpg.webp)
Klebsiella pneumoniae | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Klebsiella pneumoniae | ||||||||||||
| (Schroeter 1886) Trevisan 1887[1] |
Unter den Vertretern der Gattung ist Klebsiella pneumoniae von besonderer medizinischer Bedeutung, für diese Art sind im Krankenhaus erworbene Lungenentzündungen (nosokomiale Pneumonien) und andere Infektionen typisch. Sie verfügt über mehrere Virulenzfaktoren und es sind multiresistente Bakterienstämme bekannt, d. h. sie sind gegen viele Antibiotika resistent, so dass die Arzneimittel bei einer Infektion mit diesen Bakterienstämmen nicht mehr wirken. Personen mit geschwächtem Immunsystem oder mit akuten Infektionen sind gefährdet, auch die Stärke der Kontamination kann entscheiden.
Merkmale
Erscheinungsbild
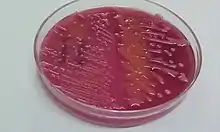
Die Zellen von Klebsiella pneumoniae erscheinen im lichtmikroskopischen Bild als kurze Stäbchen mit einer Länge von 1–2 µm und einer Breite von 0,5–0,8 µm. Sie liegen einzeln oder in Paaren vor und sind von einer Schleimkapsel (Glykokalyx) umgeben.[2] In der Gram-Färbung werden sie rosa bis rot angefärbt, sie sind gramnegativ. Wie für die Gattung Klebsiella typisch, sind sie nicht aktiv beweglich (motil), besitzen also keine Flagellen (Geißeln). Die Zelloberfläche ist jedoch mit Fimbrien besetzt.[2] Auf einem Nährboden gewachsene Bakterienkolonien weisen keine besondere Färbung auf, sie sind konvex erhaben, in der Aufsicht rund und mit einem Durchmesser von 3–4 mm eher groß, typisch ist ihr schleimiges Aussehen.[3] Dieses wird durch die Anhäufung extrazellulärer Polysaccharide verursacht, die zusammen mit dem vorhandenen Wasser einen Biofilm bilden.[2]
Wachstum und Stoffwechsel
Wie bei den Vertretern der Enterobacteriaceae üblich, verlaufen der Katalase-Test positiv und der Oxidase-Test negativ.[4] Klebsiella pneumoniae ist fakultativ anaerob, d. h. sie kann mit oder ohne Sauerstoff wachsen. Sie ist in der Lage, das Disaccharid Lactose zu verwerten.[3] Weitere Informationen sind im Abschnitt Biochemische Nachweise zu finden.
Außerdem gehört sie zu den stickstofffixierenden Mikroorganismen, sie kann elementaren, molekularen Stickstoff (N2) zu Ammoniak (NH3) bzw. Ammonium (NH4+) reduzieren und damit biologisch verfügbar machen. Dies erfolgt mit Hilfe des Enzymkomplexes Nitrogenase in einem anoxischen Milieu, da der Enzymkomplex durch Sauerstoff inaktiviert wird. Klebsiella pneumoniae ist diazotroph, kann also mit N2 als Stickstoffquelle wachsen, um daraus zelleigene Stoffe wie Aminosäuren aufzubauen.[3]
Für die Kultivierung sind einfache Nährmedien geeignet, beispielsweise Casein-Soja-Pepton-Agar (CASO-Agar), auch auf Columbia-Blutagar lassen sich die Bakterien anzüchten.[5] Häufig werden Selektivnährmedien verwendet, die zur Isolierung und Unterscheidung von Vertretern der Enterobakterien geeignet sind, beispielsweise MacConkey-Agar und Eosin-Methylen-Blau-Agar (EMB), die beide Lactose enthalten. Für eine weitere Selektion wird ein Nährmedium empfohlen, das als Kohlenstoffquelle (organische Verbindung zur Energiegewinnung) nur Citrat und Inositol enthält, es basiert auf dem Simmons Citrat-Agar mit einem Zusatz von 1 % Inositol.[3] Klebsiella pneumoniae ist mesophil, optimales Wachstum erfolgt bei einer Temperatur von 30–37 °C, nach Inkubation über ein bis zwei Tage sind Kolonien sichtbar.[5] Wachstum erfolgt auch noch bei 41 °C, nicht jedoch bei 5 °C. Bakterienstämme, die aus medizinischem Untersuchungsmaterial isoliert wurden, wachsen meist optimal bei 37 °C, jedoch verlaufen verschiedene Nachweisreaktionen zur Identifizierung besser bei einer Inkubationstemperatur von 30 °C.[3]
Chemotaxonomie
Bestandteile der Bakterienzelle wirken als Antigene, bei Klebsiella sind dies 77 verschiedene K-Antigene (K verweist auf die Kapsel), sowie 9 somatische O-Antigene. Von diagnostischer Bedeutung sind die K-Antigene, durch serologische Untersuchung lassen sich die verschiedenen Serotypen unterscheiden, was u. a. bei der Aufklärung von epidemiologischen Zusammenhängen angewandt wird. Es gibt jedoch ebenfalls ein ELISA-Verfahren zum Nachweis der O-Antigene.[3] Die Bestimmung kann auch mit Hilfe genetischer Untersuchungen erfolgen.
Genetik
Der GC-Gehalt, also der Anteil der Nukleinbasen Guanin und Cytosin in der Bakterien-DNA, liegt beim Bakterienstamm DSM 30104 (aus der Stammsammlung DSM Deutsche Sammlung von Mikroorganismen und Zellkulturen) bei 57,0 Molprozent.[6] DSM 30104 ist der Typusstamm der Subspezies Klebsiella pneumoniae subsp. pneumoniae und damit auch der Spezies, er wurde aus menschlichem Blut isoliert.[7] Das Genom wurde im Jahr 2012 vollständig sequenziert.[6]
Es liegt als ringförmigen Bakterienchromosom vor und weist eine Größe von 5.512 Kilobasenpaaren (kb) auf, was in etwa mit der Genomgröße von Escherichia coli vergleichbar ist. Es sind 5.425 codierende Gene vorhanden, außerdem wurden 77 tRNAs identifiziert. Die Gene wurden mit der Antibiotic Resistance Genes Database (ARDB, Antibiotikaresistenz-Gendatenbank) verglichen, es konnten 15 Gene identifiziert werden, die eine Resistenz vermitteln, u. a. für eine Klasse A Beta-Lactamase und eine Effluxpumpe. Zehn weitere Gene codieren für Genprodukte, die die β-Lactamase-Fähigkeiten des Bakteriums erweitern, darunter das als ampC bezeichnete Gen, welches für das als AmpC-Beta-Lactamase (in diesem Fall eine Cephalosporinase) bezeichnete Enzym codiert und das als gloB bezeichnete Gen, welches für eine als Metallo-β-Lactamase (in diesem Fall eine Carbapenemase) bezeichnete Enzym codiert.[6] Seitdem wurden über 4.200 Genome (bezogen auf das zirkuläre Bakterienchromosom) dieser Spezies sequenziert, außerdem 913 Annotationen von Plasmiden durchgeführt (Stand 2018).[8]
Plasmide tragen häufig die genetische Information für eine Antibiotikaresistenz (siehe unten) des Bakteriums, die Genprodukte sind Enzyme, die eine bestimmte chemische Struktur eines Antibiotikums verändern und dadurch die Wirkung des Arzneistoffes verhindern. Bei Klebsiella pneumoniae sind dies plasmidcodierte Beta-Lactamasen, wie die SHV-1, TEM-1, TEM-2 oder weitere ESBL (Extended Spectrum β-Lactamasen).[3] Seit Beginn des 21. Jahrhunderts beobachtet man auch Resistenzen gegen Carbapeneme, verursacht durch Carbapenemasen (carbapenem-hydrolyzing beta-lactamase), die nach dem produzierenden Bakterium als KPC (Klebsiella pneumoniae Carbapenemasen) bezeichnet werden, verschiedene Varianten werden KPC-1, KPC-2 oder KPC-3 genannt.[9] Die Besonderheit von Plasmiden ist, dass sie durch horizontalen Gentransfer zwischen verschiedenen Bakterienarten ausgetauscht werden und somit die Antibiotikaresistenz „übertragen“ wird. Ein klinischer Fallbericht der Übertragung eines Plasmids mit dem Resistenzgen blaKPC-3 von K. pneumoniae auf K. aerogenes ist dort im Artikel beschrieben.
Die Untersuchung der Nukleotidsequenz einzelner Gene ergab, dass die Art Klebsiella pneumoniae eine große Diversität aufweist. Weitere genetische Untersuchungen, beispielsweise eine Abwandlung des PCR-Verfahrens mit zufällig vervielfältigter polymorpher DNA (RAPD), bestätigen das Vorkommen von drei unterschiedlichen phylogenetischen Gruppen, die als KpI, KpII und KpIII bezeichnet werden. Sie sind nicht mit den drei Subspezies identisch.[3] Weiterführende genetische Untersuchungen in den letzten Jahren, wie Sequenzierung der 16S ribosomalen RNA (rRNA) und Multi-Locus Sequenzanalyse (MLSA) bestimmter Gene haben dazu geführt, die Vertreter der Gruppe KpII als Klebsiella quasipneumoniae zu klassifizieren bzw. die Stämme der phylogenetischen Gruppe KpIII als Klebsiella variicola.[10]
Pathogenität
Die drei Subspezies von K. pneumoniae werden durch die Biostoffverordnung in Verbindung mit der TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466 der Risikogruppe 2 zugeordnet. Bei K. pneumoniae subsp. pneumoniae und K. pneumoniae subsp. rhinoscleromatis findet sich außerdem die Bemerkung ht, sie zeigt an, dass das Bakterium pathogen für Mensch und Wirbeltiere ist, jedoch in der Regel keine Übertragung zwischen beiden Wirtsgruppen erfolgt.[11]
K. pneumoniae verfügt über mehrere Virulenzfaktoren. Die Kapsel (Glykokalyx) schützt vor Phagozytose durch die Phagozyten, Zellen des Immunsystems. Dabei stört sie das an der Abwehr von Mikroorganismen beteiligte Komplementsystem, indem dessen Aktivierung oder die Aufnahme bereits freigesetzter Polypeptide, wie C3b verhindert wird.[2] Adhäsine ermöglichen das Anheften an die Wirtszellen. Einige Adhäsine von K. pneumoniae wirken gleichzeitig als Hämagglutinine und sind den Fimbrien (Pili) zuzuordnen. Die Typ-1-Fimbrien führen bei Erythrozyten von Meerschweinchen zu einer sichtbaren Verklumpung (Agglutination), sie heften sich an humane Epithelzellen des Darms oder Epithelzellen der ableitenden Harnwege. K. pneumoniae-Isolate aus medizinischen Proben bilden mehr Typ-1-Fimbrien aus als Isolate aus Umweltproben.[3] Auch Typ-3-Fimbrien kommen vor, sie vermitteln die Anheftung der Bakterien an das pflanzliche Wurzelsystem, sowie beim Menschen an Endothelzellen, Epithelzellen der Lungenbläschen und der ableitenden Harnwege und an das Kollagen Typ V. Welche Rolle die Typ-3-Fimbrien bei der Infektion von Menschen spielen, ist noch Gegenstand der Forschung. Es wird vermutet, dass sie für die Kolonisation von invasiven medizinischen Geräten, die über eine längere Zeit im Körper verbleiben, verantwortlich sind.[3]
Die Lipopolysaccharide (LPS) der Äußeren Membran wirken als Antigene, die nach außen gerichteten Polysaccharidketten werden als O-Antigene bezeichnet (man vergleiche das bei den Salmonellen angewendete Kauffmann-White-Schema). K. pneumoniae besitzt neun verschiedene O-Antigene, wobei O1 am häufigsten vorkommt.[3] Die O-Antigene stören ebenfalls die Reaktionskaskade des Komplementsystems.[2] Außerdem ist O1 an der Nekrose von infiziertem Gewebe beteiligt.[3] Auch die bakteriellen Siderophore sind für die Pathogenität von Bedeutung. Sie dienen dazu, die Zellen mit für den Stoffwechsel essentiellen Eisen-Ionen zu versorgen, indem sie Fe3+-Ionen binden. K. pneumoniae bildet Enterobactin (Enterochelin), während nur einige Stämme zusätzlich noch Aerobactin produzieren. Bei den Serotypen K1 und K2 hat man ein Plasmid gefunden, auf dem die genetische Information für das Hydroxamat Aerobactin codiert ist. Werden diese Gene in einen Stamm ohne Plasmid mit Hilfe der Transformation übertragen, so weisen die transformierten Zellen eine um den Faktor 100 gesteigerte Virulenz auf. Auch Yersiniabactin, ein für Yersinia-Arten typisches Siderophor wird von einigen Stämmen gebildet.[3]
Biochemische Nachweise
K. pneumoniae ist nah mit K. aerogenes (früher in die Gattung Enterobacter gestellt) und Enterobacter cloacae verwandt. Die Bakterien zeigen eine ausgesprochene Vielseitigkeit in Bezug auf die Verwertung diverser Kohlenhydrate und weisen bis auf wenige Ausnahmen gleiche biochemische Merkmale auf, wie beispielsweise die vorhandenen Enzyme und die daraus resultierenden Stoffwechseleigenschaften.[4]
Vertreter der Gattung Klebsiella führen als typische Gärung die 2,3-Butandiol-Gärung zur Energiegewinnung durch, im Voges-Proskauer-Test wird Acetoin, ein Zwischenprodukt der 2,3-Butandiol-Gärung, nachgewiesen. Vertreter der verwandten Gattungen Enterobacter und Klebsiella reagieren hierbei positiv. Dies gilt prinzipiell auch für K. pneumoniae,[2][3] allerdings zeigen die Subspezies oder einzelne Bakterienstämme unterschiedliche Reaktionen, also auch ein negatives Ergebnis im VP-Test. Der Typusstamm DSM 30104 ist im Gegensatz zur Beschreibung der Art VP-negativ (produziert also kein Acetoin aus Pyruvat), dafür zeigt er im Methylrot-Test ein positives Ergebnis, was typisch für Vertreter der gemischten Säuregärung ist.[5] Diese Unterschiede im physiologischen Phänotyp spiegeln die genetische Diversität der Bakterienart wider. Auch weitere biochemische Merkmale sind innerhalb der Art nicht eindeutig festzulegen. So ist der Indol-Test als Unterscheidungsmerkmal zwischen K. pneumoniae (Indol-negativ) und Klebsiella oxytoca (Indol-positiv) grundsätzlich geeignet, allerdings gibt es auch einige Indol-positive Stämme von K. pneumoniae.[3]
Weitere Nachweise
Anstatt das Bakterium nachzuweisen, beschränkt man sich oft auf die Bestimmung des Serotyps oder den Nachweis einzelner Virulenzfaktoren oder Resistenzgene. Die K- und O-Antigene können sowohl „konventionell“ serologisch (in der englischsprachigen Literatur als serotyping bezeichnet) wie seit Verbreitung der molekularbiologischen Methoden auch durch diese bestimmt werden, beispielsweise mit Hilfe der Multi-Locus Sequenzanalyse (MLSA). Durch Vergleich mit den zahlreichen sequenzierten Genomen der Art konnte gezeigt werden, dass bei Stämmen mit den Kapsel-Antigenen K1 oder K2 fast immer der Serotyp O1 auftritt. Die Serotypen K1 und K2 werden als hypervirulent angesehen.[12] Die Bestimmung der Kapsel-Antigene kann ebenfalls durch die Multiplex-PCR (es wird mehr als ein Genomabschnitt nachgewiesen) und die Pulsed-Field-Gelelektrophorese (PFGE) erfolgen.[13]
Die Identifizierung mit Hilfe der MALDI-TOF-Methode in Kombination mit Massenspektrometrie (MS) ist grundsätzlich geeignet, Klebsiella nachzuweisen, ist aber nicht immer zuverlässig in Bezug auf die Unterscheidung nah verwandter Gattungen, beispielsweise zu Raoultella. Die Spektren vieler gramnegativer Spezies, die zu den Enterobakterien gehören, zeigen eine große Übereinstimmung (Stand 2013), was die Identifizierung erschwert. Eine andere systematische Untersuchung von in einer flüssigen, bluthaltigen Nährlösung kultivierten Bakterien zeigte, dass insbesondere Bakterien mit einer Kapsel nicht korrekt identifiziert werden.[14] Hingegen kann beim Nachweis der Antibiotikaresistenz MALDI-TOF angewendet werden, um das Fehlen oder verringerte Vorhandensein von Proteinen in der äußeren Membran (englisch: outer membrane proteins, OMP) zu detektieren. Bei K. pneumoniae ist hier OmpK36 von Bedeutung, ein wichtiges Membranporin, durch das β-Lactam-Antibiotika in die Zelle gelangen. Bei resistenten Stämmen fehlt es oder wird in geringer Zahl ausgebildet.[14]
Systematik
Der deutsche Mikrobiologe Carl Friedländer beschrieb diese Bakterien erstmals 1883 als Erreger einer seltenen Form der Lungenentzündung (Friedländer-Pneumonie). Er nannte sie damals noch „Diplococcus“. Kurz danach wurden die „Friedländer-Bakterien“ als „Bacterium pneumoniae crouposae“ Zopf 1885, „Hyalococcus pneumoniae“ Schroeter 1886 und „Bacillus pneumoniae“ (Schroeter 1886) Flügge 1886 beschrieben.[1]
1984 wurde die Art in drei Unterarten (Subspezies) aufgeteilt:[1]
- Klebsiella pneumoniae subsp. ozaenae (Abel 1893) Ørskov 1984, comb. nov. → Basonym: Klebsiella ozaenae (Abel 1893) Bergey et al. 1925 (Approved Lists 1980)
- Klebsiella pneumoniae subsp. pneumoniae (Schroeter 1886) Ørskov 1984, subsp. nov.
- Klebsiella pneumoniae subsp. rhinoscleromatis (Trevisan 1887) Ørskov 1984, comb. nov. → Basonym: Klebsiella rhinoscleromatis Trevisan 1887 (Approved Lists 1980)
Klebsiella pneumoniae (Schroeter 1886) Trevisan 1887 ist durch den Typusstamm ATCC 13883 (= CCUG 225 = CIP 82.91 = DSM 30104 = HAMBI 450 = IAM 14200 = IFO (NBRC) 14940 = JCM 1662 = LMG 2095 = NCTC 9633) definiert, nach der Unterteilung in drei Subspezies ist dies auch der Typusstamm für K. pneumoniae subsp. pneumoniae (Schroeter 1886) Ørskov 1984.[1][5]
Die Unterteilung in drei Subspezies beruht auf Merkmalen der Pathogenese und nicht auf der ausreichenden Unterscheidbarkeit der DNA-Sequenz. Durch genetische Untersuchungen wurde 2003 gezeigt, dass innerhalb der Art mindestens drei unterschiedliche phylogenetische Gruppen existieren, dies sind jedoch nicht die hier erwähnten Unterarten. Bei durch K. pneumoniae verursachten Lungenentzündungen sind zwar Stämme, die zur phylogenetischen Gruppe KpI gehören, vorherrschend, aber Vertreter der Gruppen KpII (K. quasipneumoniae) und KpIII (K. variicola) kommen ebenfalls vor.[3]
Bedeutung
Vorkommen und Ökologie
Klebsiella pneumoniae ist ubiquitär verbreitet. Ihre natürlichen Habitate sind Gewässer, Abwasser (besonders von Industrieanlagen der Papierherstellung und Fruchtverarbeitung), Erdboden und Pflanzen, dort insbesondere die Wurzeln. Die Bakterien heften sich mit Hilfe ihrer Fimbrien (vor allem der Typ-3-Fimbrien) an die Wurzelhaare der Rhizodermis, um dort Stickstoff zu fixieren. Die Stickstofffixierung erfolgt – anders als bei den Rhizobien (Knöllchenbakterien), die ausschließlich in Symbiose mit Pflanzen aus der Familie der Leguminosen leben – bei K. pneumoniae assoziativ mit verschiedenen höheren Pflanzen (Gefäßpflanzen), manchmal als Endophyt. So wurde K. pneumoniae auch von Blättern der Reispflanze, dem Gewebe der Halme von Zea mays (Mais) und verrottendem Holz isoliert. Bei Literaturangaben zum Vorkommen ist jedoch zu beachten, dass die Identifizierung nicht immer zuverlässig war, so dass vereinzelt Isolate auch anderen Klebsiella-Arten bzw. Vertretern der Gattung Raoultella (die früher zur Gattung Klebsiella gestellt waren) zuzurechnen sind.[3]
Das Bakterium besiedelt auch Tiere und Menschen, u. a. den Darm, K. pneumoniae wurde von zahlreichen Vertretern der Säugetiere und Insekten isoliert. Bei Stuten (weiblichen Hauspferden) kann sie eine Metritis, eine Entzündung der Gebärmutter, verursachen. Dies gilt für Stämme mit dem Kapselantigen K1, K2 und K5, durch die es zu Epidemien gekommen ist. Hingegen werden Stämme mit dem Kapselantigen K7 eher als opportunistische Erreger betrachtet. Neben dem menschlichen Darm, wo es zur Darmflora gehört, findet sich das Bakterium auch im Nasenrachenraum (Nasopharynx). Vor dem Auftreten multiresistenter Stämme wurde angenommen, dass die Besiedelung der Patienten oder des Krankenhauspersonals das Reservoir ist, durch das eine Infektion mit K. pneumoniae verursacht werden kann. Untersuchungen bei epidemischen Ausbrüchen in Krankenhäusern – also bei nosokomialen Infektionen – mit Hilfe serologischer (Bestimmung der K-Antigene) oder molekularbiologischer Methoden zeigen ein anderes Bild. Häufig lässt sich später ein bestimmter antibiotikaresistenter Klon identifizieren, der für die Infektionen verantwortlich war.[3] Sofern ein Screening der Patienten bei Aufnahme und in regelmäßigen Abständen im Hinblick auf die Besiedelung mit K. pneumoniae erfolgt, kann bewiesen werden, dass dieser Klon nicht ursprünglich bei dem Patienten zu finden war. Untersuchungen in italienischen Krankenhäusern ergaben 2012 die Verbreitung der multiresistenten Klone ST101, ST258 und ST512.[15]
In Israel wurde eine Symbiose zwischen dem Bakterium, Larven des Steppenrüsslers Conorhynchus pistor und der Pflanze Salsola inermis beobachtet. Die Larven des Rüsselkäfers leben in Lehmkokons an den Wurzeln der Pflanze, die Bakterien leben im Verdauungstrakt der Käferlarven. Die Ausscheidungen der Käferlarven versorgen die Pflanze mit Stickstoff. Es wurde beobachtet, dass der Befall von den Käferlarven eine positive Auswirkung auf die Pflanzen hat.[16]
Medizinische Bedeutung
Bei durch Klebsiella pneumoniae (Klebsiella pneumoniae subsp. pneumoniae) ausgelösten Erkrankungen handelt es sich häufig um Infektionen der ableitenden Harnwege (Harnwegsinfekt, HWI bzw. CTI als Abkürzung im englischen Sprachraum) oder Atemwege (Pneumonien).[2] Eine Pneumonie (auch die Friedländer-Pneumonie) wird hauptsächlich durch Serotypen mit dem K1-Antigen verursacht, daneben können auch K2, K3, K4, K5 und K6 beteiligt sein.[3] Zunehmend ist das Bakterium bekannt dafür, nosokomiale Pneumonien bei immuninkompetenten stationären Patienten auszulösen.
Nosokomiale Infektionen („Krankenhausinfektionen“) bei immunsupprimierten Patienten werden häufig durch Anwendung invasiver medizinischer Verfahren, z. B. beim Einführen von Kathetern oder der intensivmedizinischen Beatmung verursacht. Die nosokomial erworbenen Infektionskrankheiten umfassen neben Pneumonien ebenfalls Harnwegsinfekte, Wundinfektionen, Bakteriämien bis zur Sepsis sowie Cholezystitis.[2] Neben immunsupprimierten Patienten sind Neugeborene gefährdet, in den letzten Jahren kam es weltweit zu mehreren epidemischen Ausbrüchen in Neugeborenen-Intensivstationen, mit Todesfolgen.[17] Die klinische Bedeutung von (nosokomialen) Infektionen mit K. pneumoniae ist verknüpft mit der verbreiteten Multiresistenz des Bakteriums. K. pneumoniae gehört zu den fünf häufigsten Krankheitserregern der bakteriellen Sepsis (6,7 % der Fälle, 2006 veröffentlicht) bzw. der nosokomial erworbenen Pneumonie (10,1 % der Fälle, 2010 veröffentlicht).[9] Sie gehört zur sogenannten ESKAPE-Gruppe.
Ein aktueller Ansatz zur Unterscheidung der Klebsiella pneumoniae-Bakterienstämme im Hinblick auf ihre medizinische Relevanz, der auch durch genetische Untersuchungen bestätigt wird, ist die Einteilung in folgende drei Gruppen: Opportunistische, hypervirulente und multiresistente Stämme.[18] Die opportunistischen Erreger infizieren insbesondere Patienten mit einem geschwächten Immunsystem und sind typisch für nosokomiale Infektionen (siehe oben). Hypervirulente K. pneumoniae sind sogenannte community acquired (deutsch „ambulant erworbene“) Stämme, die gesunde Menschen außerhalb von Einrichtungen des Gesundheitswesens besiedeln bzw. infizieren. Sie verursachen schwere Infektionen, wie beispielsweise Pyogenen Leberabszess, Endophthalmitis (Infektion im Auge) und Meningitis (Hirnhautentzündung), diese werden seit den 1990er Jahren vermehrt aus Asien und den Pazifikanrainerstaaten gemeldet.[18] Insbesondere die Serotypen K1 und K2 werden als hypervirulent bezeichnet. Genetische Vergleichsuntersuchungen zeigen, dass bei ihnen besonders viele Virulenzfaktoren auftreten, u. a. eine große Anzahl an Siderophoren, die Bildung von Colibactin (schädigt die DNA) sowie das Regulatorgen rmpA (mucosity regulator, bezieht sich auf die Schleimkapsel).[12] Für multiresistente K. pneumoniae ist die Produktion von Carbapenemasen typisch, dadurch wird die Behandlung der Infektion zunehmend schwierig (vergleiche folgende Abschnitte).
Klebsiella pneumoniae und Autoimmunerkrankungen
Es gibt Studien,[19] die darauf hinweisen, dass durch natürliche Abwehrreaktionen gebildete und gegen Klebsiella pneumoniae gerichtete IgA-Antikörper mit Strukturen des humanen Zelloberflächenproteins HLA-B27 kreuzreagieren. HLA-B27 reguliert wichtige Funktionen des menschlichen Immunsystems. Klebsiella pneumoniae steht im Verdacht, über diesen Mechanismus auch Autoimmunreaktionen wie z. B. Spondylitis ankylosans (Morbus Bechterew) auszulösen.
Antibiotikaresistenzen
K. pneumoniae verfügt über eine natürliche Antibiotikaresistenz gegen Benzylpenicillin, Aminopenicilline (z. B. Ampicillin und Amoxicillin) und Carboxypenicilline (z. B. Carbenicillin und Ticarcillin), allesamt β-Lactam-Antibiotika. Die Resistenz beruht auf der im Bakterienchromosom codierten Klasse A Beta-Lactamase. Zusätzlich dazu auftretende, erworbene Resistenzen sind häufig durch plasmidcodierte ESBL (Extended Spectrum β-Lactamasen) wie die SHV-1, TEM-1 oder TEM-2 bedingt (siehe Abschnitt Genetik). Dadurch ist K. pneumoniae resistent gegen weitere Penicilline sowie Cephalosporine der 3. Generation (z. B. Cefotaxim und Ceftazidim). Ende der 1990er waren in den USA 24 % der K. pneumoniae-Stämme resistent gegen Ceftazidim.[3] An dem deutschen epidemiologische Überwachungsprogramm Surveillance der Antibiotika-Anwendung und der bakteriellen Resistenzen auf Intensivstationen (SARI) beteiligte Intensivstationen meldeten einen Anstieg der Resistenz gegenüber Cephalosporinen der 3. Generation von 2,2 % (2000) auf 16,8 % (2010).[9] Seit Beginn des 21. Jahrhunderts beobachtet man auch Resistenzen gegen Carbapeneme, verursacht durch Carbapenemasen (eine weitere Gruppe der β-Lactamasen), die nach dem produzierenden Bakterium als KPC (Klebsiella pneumoniae Carbapenemasen) bezeichnet werden (vergleiche nächster Abschnitt).
Carbapenemresistente Klebsiella pneumoniae-Stämme (KPC)
Erstmals 2001 wurde an einen bestimmten Klebsiella pneumoniae-Stamm die Bildung einer Carbapenemase (carbapenem-hydrolyzing beta-lactamase) beobachtet: Diese bewirkt eine Resistenz der Klebsiellen gegenüber bestimmten Antibiotika, den Carbapenemen. Zu diesen gehören z. B. die Arzneistoffe Imipenem und Meropenem. Die Aktivität der Carbapenemase wird jedoch in Gegenwart von Clavulansäure unterdrückt. Der untersuchte carbapenemresistente Klebsiella pneumoniae-Stamm (carbapenem-resistant Klebsiella pneumoniae, CRKP) „1534“ zeigte weiterhin Resistenz gegen alle Cephalosporine und Aztreonam und ist damit weitgehend unempfindlich gegen viele moderne Antibiotika.[20] Es sind verschiedene Varianten der Klebsiella pneumoniae Carbapenemasen bekannt, wie etwa KPC-1, KPC-2 und KPC-3.[21][22]
Multiresistente Stämme
Carbapenemase-produzierende Enterobacteriaceae (CPE) werden in Deutschland als 3MRGN oder 4MRGN (multiresistente gramnegative Bakterien) klassifiziert.[9] In der Erregergruppe CPE ist K. pneumoniae überproportional vertreten, durch horizontalen Gentransfer sind die KPC jedoch auch bei verwandten Arten der Enterobakterien anzutreffen, beispielsweise bei Escherichia coli, Serratia marcescens, Enterobacter cloacae, Citrobacter freundii sowie bei K. oxytoca und K. aerogenes.[15] Da meist bei Auftreten einer ESBL oder einer Carbapenemase auch eine Resistenz gegen Fluorchinolone auftritt, sind diese Stämme gegenüber allen vier im MRGN-System definierten Antibiotikaklassen resistent und werden als 4MRGN bezeichnet.[9] Das Robert Koch-Institut (RKI) berichtete 2012 über den ersten Fall aus Deutschland, bei dem aus der Operationswunde an der Hüfte eines Patienten ein multiresistenter Stamm isoliert wurde. Das Antibiogramm bestätigte das Isolat als 4MRGN, zusätzlich wurde auch eine Resistenz gegenüber Colistin nachgewiesen, das in solchen Fällen eigentlich als Reserveantibiotikum verwendet wird.[23]
2012 warnte die beim RKI eingerichtete KRINKO (Kommission für Krankenhaushygiene und Infektionsprävention), „(…) dass sich Deutschland gerade am Beginn einer Entwicklung befindet, bei der es zur Zunahme Carbapenem-resistenter K. pneumoniae-Stämme kommt.“[9] International betrachtet sind CPE schon weit verbreitet oder zumindest ein vermehrt auftretendes Problem. Dies zeigt beispielsweise das international agierende Überwachungssystem European survey on carbapenemase-producing Enterobacteriaceae (EuSCAPE). An diesem Surveillance-System sind die 28 Mitgliedstaaten der Europäischen Union, sieben (potenzielle) Beitrittskandidaten, Island, Norwegen und Israel beteiligt. Ein 2013 veröffentlichter Zwischenbericht gibt an, dass 29 dieser 38 Staaten ein nationales Surveillance-System für Carbapenemase-produzierende Enterobacteriaceae (CPE) unterhalten, in allen nationalen Programmen wird auch Klebsiella pneumoniae überwacht. 33 der „nationalen Experten“ gaben dabei an, dass in ihrem Land K. pneumoniae die Bakterienart ist, die am häufigsten unter den CPE nachgewiesen wird.[24]
Beispiele von Infektionen mit multiresistenten Stämmen
Im Washoe County, Nevada, USA wurde ein multiresistenter Klebsiella pneumoniae-Stamm an einer Patientin entdeckt, die zuvor in Indien mehrere Krankenhausaufenthalte hatte. Das Bakterium war gegen alle zugelassenen Antibiotika resistent. Im Labor zeigte einzig Fosfomycin eine Wirkung, das allerdings nur oral eingenommen werden konnte, da es in den USA keine Zulassung für eine intravenöse Behandlung hatte, wie sie die Patientin benötigte. Die Patientin verstarb, nachdem die Verabreichung aller 26 in den USA zugelassenen Antibiotika, u. a. auch Fosfomycin in oraler Form, keine Wirkung erzielte.[25] Im Mai 2017 fand man im Universitätsklinikum Frankfurt, dem größten Krankenhaus Hessens, bei fünf Patienten den Erreger Klebsiella pneumoniae 4MRGN.[26] 2011 und 2012 gab es im Klinikum Bremen-Mitte mehrere Todesfälle auf der Neugeborenen-Intensivstation durch multiresistente Stämme.[27]
Weblinks
- Taxonomy Browser Klebsiella pneumoniae. In: Website des National Center for Biotechnology Information (NCBI). Abgerufen am 30. Juni 2018.
- Deutsche Sammlung von Mikroorganismen und Zellkulturen (DSMZ): Klebsiella pneumoniae subsp. pneumoniae, Type Strain. In: Website BacDive. Abgerufen am 30. Juni 2018.
Quellen
Literatur
- Sylvain Brisse, Francine Grimont, Patrick A. D. Grimont: The Genus Klebsiella (Chapter 3.3.8). In: Martin Dworkin, Stanley Falkow, Eugene Rosenberg, Karl-Heinz Schleifer, Erko Stackebrandt (Hrsg.): The Prokaryotes. A Handbook on the Biology of Bacteria, Band 6: Proteobacteria: Gamma Subclass. 3. Auflage. Springer-Verlag, New York 2006, ISBN 978-0-387-25496-8, S. 159–196, doi:10.1007/0-387-30746-X_8.
- Empfehlung der Kommission für Krankenhaushygiene und Infektionsprävention (KRINKO) beim Robert Koch-Institut (RKI): Hygienemaßnahmen bei Infektionen oder Besiedlung mit multiresistenten gramnegativen Stäbchen. In: Bundesgesundheitsblatt. Band 55, Oktober 2012, S. 1311–1354, doi:10.1007/s00103-012-1549-5 (PDF, Volltext (PDF; 1,1 MB) ); hier speziell Kapitel 2.1.2 Klebsiella spp. S. 1317.
Einzelnachweise
- Jean Euzéby, Aidan C. Parte: Genus Klebsiella. In: List of Prokaryotic names with Standing in Nomenclature (LPSN). Abgerufen am 30. Juni 2018.
- Subhash Chandra Parija: Coliforms: Klebsiella, Enterobacter (Chapter 31). In: Textbook of Microbiology and Immunology. 2. Auflage. Elsevier India, Haryana 2012, ISBN 978-81-312-2810-4, S. 261–264.
- S. Brisse, F. Grimont, P. A. D. Grimont: The Genus Klebsiella. In: The Prokaryotes. A Handbook on the Biology of Bacteria, Band 6. 2006, S. 159–196.
- Shoshana Bascomb, Stephen P. Lapage, W. R. Willcox, M. A. Curtis: Numerical Classification of the Tribe Klebsielleae. In: Journal of General Microbiology. Band 66, Nr. 3, 1971, S. 279–295, doi:10.1099/00221287-66-3-279.
- Deutsche Sammlung von Mikroorganismen und Zellkulturen (DSMZ): Klebsiella pneumoniae subsp. pneumoniae, Type Strain. In: Website BacDive. Abgerufen am 30. Juni 2018.
- J. H. Lee, I. S. Cheon, B. S. Shim, D. W. Kim, S. W. Kim, J. Chun, M. Song: Draft genome sequence of Klebsiella pneumoniae subsp. pneumoniae DSM 30104T. In: Journal of Bacteriology. Band 194, Nr. 20, Oktober 2012, S. 5722–5723, doi:10.1128/JB.01388-12, PMID 23012294, PMC 3458660 (freier Volltext).
- Sample from Klebsiella pneumoniae subsp. pneumoniae DSM 30104. In: Website des National Center for Biotechnology Information (NCBI). Abgerufen am 1. Juli 2018.
- Klebsiella pneumoniae. In: Website Genome des National Center for Biotechnology Information (NCBI). Abgerufen am 1. Juli 2018.
- Empfehlung der Kommission für Krankenhaushygiene und Infektionsprävention (KRINKO) beim Robert Koch-Institut (RKI): Hygienemaßnahmen bei Infektionen oder Besiedlung mit multiresistenten gramnegativen Stäbchen. In: Bundesgesundheitsblatt - Gesundheitsforschung - Gesundheitsschutz. Band 55, 2012, S. 1311–1354, doi:10.1007/s00103-012-1549-5.
- S. Brisse, V. Passet, P. A. D. Grimont: Description of Klebsiella quasipneumoniae sp. nov., isolated from human infections, with two subspecies, Klebsiella quasipneumoniae subsp. quasipneumoniae subsp. nov. and Klebsiella quasipneumoniae subsp. similipneumoniae subsp. nov., and demonstration that Klebsiella singaporensis is a junior heterotypic synonym of Klebsiella variicola. In: International Journal of Systematic and Evolutionary Microbiology. Band 64, September 2014, S. 3146–3152, doi:10.1099/ijs.0.062737-0.
- TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466: Einstufung von Prokaryonten (Bacteria und Archaea) in Risikogruppen. In: Webseite der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA). 25. August 2015, S. 115, 164, abgerufen am 29. März 2018 (letzte Änderung vom 31. März 2017).
- R. Follador, E. Heinz, K. L. Wyres, M. J. Ellington, M. Kowarik, K. E. Holt, N. R. Thomson: The diversity of Klebsiella pneumoniae surface polysaccharides. In: Microbial Genomics. Band 2, Nr. 8, August 2016, S. e000073, doi:10.1099/mgen.0.000073, PMID 28348868, PMC 5320592 (freier Volltext).
- Jane F. Turton, Hatice Baklan, L.K. Siu, Mary E. Kaufmann, Tyrone L. Pitt: Evaluation of a multiplex PCR for detection of serotypes K1, K2 and K5 in Klebsiella sp. and comparison of isolates within these serotypes. In: FEMS Microbiology Letters. Band 284, Nr. 2, Juli 2008, S. 247–252, doi:10.1111/j.1574-6968.2008.01208.x.
- Andrew E. Clark, Erin J. Kaleta, Amit Arora, Donna M. Wolk: Matrix-assisted laser desorption ionization-time of flight mass spectrometry: a fundamental shift in the routine practice of clinical microbiology. In: Clinical Microbiology Reviews. Band 26, Nr. 3, Juli 2013, S. 547–603, doi:10.1128/CMR.00072-12, PMID 23824373, PMC 3719498 (freier Volltext) (Review).
- Carolina Venditti, Laura Villa u. a.: Isolation of KPC 3-producing Enterobacter aerogenes in a patient colonized by MDR Klebsiella pneumoniae. In: New Microbiologica. Band 39, Nr. 4, Oktober 2016, S. 310–313, PMID 27284988.
- Oren Shelef, Yael Helman, Ariel-Leib-Leonid Friedman, Adi Behar, Shimon Rachmilevitch: Tri-Party Underground Symbiosis between a Weevil, Bacteria and a Desert Plant. In: PLOSone. Band 8, Nr. 11, 2013, S. 1-7 (Volltext online).
- Arbeitsgruppe Neonatologische Intensivmedizin der KRINKO, Leitung Arne Simon: Risikocharakterisierung intensivmedizinisch behandelter Früh- und Neugeborener und Daten zur Ist-Situation in deutschen neonatologischen Intensivpflegestationen 2013. (PDF; 345 kB) Robert Koch-Institut (RKI), 23. Oktober 2013, S. 1–52, abgerufen am 1. Juli 2018.
- Rebekah M. Martin, Michael A. Bachman: Colonization, Infection, and the Accessory Genome of Klebsiella pneumoniae. In: Frontiers in Cellular and Infection Microbiology. Band 8, 2018, S. 4, doi:10.3389/fcimb.2018.00004, PMID 29404282, PMC 5786545 (freier Volltext) (Review).
- M. Ogasawara, D. H. Kono, D. T. Yu: Mimicry of human histocompatibility HLA-B27 antigens by Klebsiella pneumoniae. In: Infection and Immunity. Band 51, Nummer 3, März 1986, S. 901–908, PMC 260984 (freier Volltext).
- H. Yigit, A. M. Queenan u. a.: Novel carbapenem-hydrolyzing beta-lactamase, KPC-1, from a carbapenem-resistant strain of Klebsiella pneumoniae. In: Antimicrobial Agents and Chemotherapy. Band 45, Nummer 4, April 2001, S. 1151–1161, doi:10.1128/AAC.45.4.1151-1161.2001. PMID 11257029. PMC 90438 (freier Volltext).
- Ashfaque Hossain u. a.: Plasmid-Mediated Carbapenem-Hydrolyzing Enzyme KPC-2 in an Enterobacter sp. In: Antimicrob Agents Chemother. Band 48, Nummer 11, November 2004, S. 4438–4440, doi:10.1128/AAC.48.11.4438-4440.2004, PMC 525415 (freier Volltext).
- Ben M. Lomaestro: The Spread of Klebsiella pneumoniae Carbapenemase–Producing K. pneumoniae to Upstate New York. In: Clinical Infectious Diseases. Band 43, 2006, S. e26–e28.
- Fallbericht des Nachweises eines multiresistenten Klebsiella pneumoniae-Isolates bei einem Patienten. In: Robert Koch-Institut (Hrsg.): Epidemiologisches Bulletin. Nr. 45, 12. November 2012, S. 453–455 (rki.de [PDF; 94 kB; abgerufen am 2. Juli 2018]).
- Hajo Grundmann, Corinna Glasner, Anna-Pelagia Magiorakos, Liselotte Högberg-Diaz, Dominique L. Monnet, Barbara Albiger: Carbapenemase-producing bacteria in Europe. (PDF; 3,3 MB) ECDC Technical Report. Europäisches Zentrum für die Prävention und die Kontrolle von Krankheiten (ECDC), November 2013, S. 1–18, abgerufen am 24. Mai 2018 (englisch).
- L. Chen, R. Todd, J. Kiehlbauch, M. Walters, A. Kallen: Notes from the Field: Pan-Resistant New Delhi Metallo-Beta-Lactamase-Producing Klebsiella pneumoniae – Washoe County, Nevada, 2016. In: Morbidity and Mortality Weekly Report (MMWR), Nummer 66, 2017, S. 33, doi:10.15585/mmwr.mm6601a7.
- Nach Klebsiella-Befall: Intensivstation an Uni-Klinikum weiter gesperrt. faz.net, 8. Mai 2017, abgerufen am 1. Juli 2018.
- Klebsiella – der Todes-Keim auf der Frühchenstation. welt.de, 3. November 2011, abgerufen am 1. Juli 2018.