Nitrogenase
Nitrogenase ist ein Enzymkomplex, der in der Lage ist, elementaren, molekularen Stickstoff (N2) zu reduzieren und damit in eine biologisch verfügbare Form umzuwandeln. Diesen Vorgang bezeichnet man als Stickstofffixierung. Nitrogenasen sind bei verschiedenen Bakterien und einigen Archaeen vorhanden. Stickstofffixierende Aktinomyceten sind ebenso bekannt wie Cyanobakterien (zum Beispiel Anabaena) und Proteobakterien (zum Beispiel Azotobacter).
| Nitrogenase | ||
|---|---|---|
| Bezeichner | ||
| Gen-Name(n) | nif, anf, vnf | |
| Enzymklassifikation | ||
| EC, Kategorie | 1.18.6.1, Oxidoreduktase | |
| Substrat | 8 Ferredoxinred. + 8 H+ + N2 + 16 ATP + 16 H2O | |
| Produkte | 8 Ferredoxinox. + H2 + 2 NH3 + 16 ADP + 16 Phosphat | |
| Vorkommen | ||
| Übergeordnetes Taxon | Bakterien, Archaeen | |
Struktur und Eigenschaften
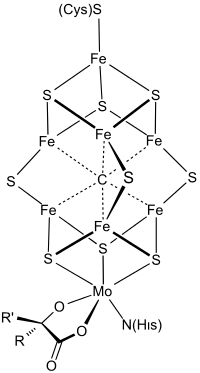
Der Enzymkomplex besteht aus zwei enzymatisch aktiven Proteinen, der Dinitrogenase (Heterotetramer α2β2) und der Dinitrogenase-Reduktase (Homodimer). Die Reaktion findet in der Dinitrogenase statt, die Reduktase überträgt die Elektronen, die sie von Ferredoxin erhält, mittels eines [4Fe-4S]-Eisen-Schwefel-Clusters. Das aktive Zentrum der Dinitrogenase besteht aus einem weiteren [8Fe-7S]-Eisen-Schwefel-Cluster, sowie dem Eisen-Schwefel-Molybdän-Cofaktor (FeMoCo). Lange Zeit war die Identität des zentralen Atoms des FeMoCo-Faktors unklar. Sowohl ein Kohlenstoff-, ein Sauerstoff- als auch ein Stickstoffatom waren plausible Kandidaten. Zwei unabhängige X-Ray-Emission-Spectroscopy-Untersuchungen ergaben 2011, dass es sich bei dem interstitiellen Atom im Zentrum des Clusters um ein Kohlenstoffatom handelt.[1][2] Durch neuere Computersimulationen wurde zudem 2017 die Festigkeit des zentralen Kohlenstoffs bestimmt.[3] Einige Bakterien sind in der Lage, bei Molybdänmangel alternative Cofaktoren herzustellen, die statt Molybdän Vanadium oder nur Eisen enthalten.[4][5][6][7] Diese Strukturen sind im Vergleich zu FeMoco jedoch deutlich instabiler.[8] Die Nitrogenase der meisten Bakterien ist wie alle Enzyme mit Eisen-Schwefel-Clustern extrem sauerstoffempfindlich. Um das Enzym gegen Sauerstoff zu schützen, haben Bakterien verschiedene Anpassungen entwickelt, etwa dicke Schleimkapseln oder besonders dickwandige Zellen. Bakterien, die oxygene Photosynthese betreiben, trennen stickstofffixierende Zellen (Heterozysten) räumlich von Sauerstoff freisetzenden Zellen oder sie assimilieren Stickstoff nur nachts, wenn die Lichtreaktion der Photosynthese ruht. Einige Bakterien können Stickstoff nur in Symbiose fixieren, zum Beispiel Rhizobien (Knöllchenbakterien), die mit Pflanzen (oft Leguminosen) zusammenleben. Da die Pflanzen selbst nicht in der Lage sind, elementaren, molekularen Stickstoff zu fixieren, sind sie auf das Produkt der bakteriellen Nitrogenase angewiesen. Das gebildete Ammoniak ist Ausgangsstoff für die Bildung von Glutaminsäure und Glutamin.
Katalysierte Reaktion
Da N2 ein sehr stabiles und inertes Molekül ist (die Bindungsenergie beträgt 945 kJ/mol), wird eine große Menge an Energie benötigt, um die Dreifachbindung zwischen den beiden Stickstoff-Atomen zu spalten. Die hierfür benötigte Energie, liefert der Energieträger ATP. Als Produkt dieser Reaktion entsteht Ammoniak.
Die Summengleichung der durch Nitrogenase katalysierten Reaktion lautet:
Die Aktivität der Nitrogenase ist nicht auf Stickstoff beschränkt, das Enzym reduziert auch andere Dreifachbindungen, zum Beispiel die von Ethin, Cyanid, Aziden oder Stickstoffoxiden. Unter natürlichen Bedingungen hat diese Eigenschaft vermutlich keine Bedeutung. Die Reduktion von Ethin zu Ethen wird jedoch genutzt, um Nitrogenase experimentell nachzuweisen.
Regulation
Der gesamte Prozess der biologischen Stickstofffixierung ist relativ komplex und erfordert das Zusammenwirken mehrerer Enzyme, von denen die Nitrogenase das wichtigste ist. Die Gene dieser Enzyme unterliegen einer strengen Regulation. Ihre Transkription wird zum Beispiel durch Sauerstoff und Stickstoffverbindungen wie Nitrat und einige Aminosäuren abgeschaltet. Die Abschaltung durch Stickstoffverbindungen ist vorteilhaft, weil die Nutzung von Stickstoff aus diesen Quellen wesentlich weniger Energie verbraucht.
Die Nitrogenase-Aktivität wird bei Klebsiella pneumoniae über das nifLA-Operon auf transkriptionaler Ebene reguliert. Das Protein NifL ist dabei ein Sensor für O2, NifA ist unter anderem ein transkriptionaler Aktivator für das nifHDKY-Operon, welches die strukturellen Elemente der Nitrogenase codiert. Ist das Bakterium Sauerstoff ausgesetzt, bilden NifL und NifA ein Heterodimer, NifA kann in Folge die Aktivatorfunktion nicht ausüben, Nitrogenase wird nicht exprimiert. Dieser Effekt ist sinnvoll, da Nitrogenase durch O2 inhibiert werden würde und eine Expression bei Anwesenheit von O2 reine Energieverschwendung wäre.[9] Die Expression der Gene nifL und nifA selbst wird von NtrC gestartet. Dieser Faktor signalisiert den Bedarf, Stickstoff zu fixieren.[10]
Siehe auch
Einzelnachweise
- T. Spatzal, M. Aksoyoglu, L. Zhang, S. L. A. Andrade, E. Schleicher, S. Weber, D. C. Rees, O. Einsle: Evidence for Interstitial Carbon in Nitrogenase FeMo Cofactor. In: Science. 334, 6058, S. 940, doi:10.1126/science.1206445.
- K. M. Lancaster, M. Roemelt, P. Ettenhuber, Y. Hu, M. W. Ribbe, F. Neese, U. Bergmann, S. DeBeer: X-ray Emission Spectroscopy Evidences a Central Carbon in the Nitrogenase Iron-Molybdenum Cofactor. In: Science. 334, 2011, S. 974–977, doi:10.1126/science.1206445.
- J. Grunenberg: Der interstitiell gebundene Kohlenstoff der Nitrogenase ist deutlich stabiler als bisher angenommen. In: Angewandte Chemie. 2017, 129, S. 7394–7397, doi:10.1002/ange.201701790.
- PROSITE documentation PDOC00085. Swiss Institute of Bioinformatics (SIB), abgerufen am 20. September 2011 (englisch).
- PROSITE documentation PDOC00580. Swiss Institute of Bioinformatics (SIB), abgerufen am 20. September 2011 (englisch).
- R. N. Pau, M. E. Eldridge, D. J. Lowe, L. A. Mitchenall, R. R. Eady: Molybdenum-independent nitrogenases of Azotobacter vinelandii: a functional species of alternative nitrogenase-3 isolated from a molybdenum-tolerant strain contains an iron-molybdenum cofactor. In: Biochem. J.. 293 (Pt 1), Juli 1993, S. 101–7. PMID 8392330. PMC 1134325 (freier Volltext).
- A. N. Glazer, K. J. Kechris: Conserved amino acid sequence features in the alpha subunits of MoFe, VFe, and FeFe nitrogenases. In: PLoS ONE. 4, Nr. 7, 2009, S. e6136. doi:10.1371/journal.pone.0006136. PMID 19578539. PMC 2700964 (freier Volltext).
- Heldt, Piechulla: Pflanzenbiochemie, Springer, 2017, ISBN 978-3-662-44397-2, S. 300
- The use of cloned nif (Nitrogen fixation) DNA to investigate transcriptional regulation of nif expression in Klebsiella pneumoniae
- Positive control and autogenous regulation of the nifLA promoter in Klebsiella pneumoniae