tRNA
Die Kurzform tRNA steht für transfer-RNA. Transfer-RNAs sind kurze Ribonukleinsäurestränge (RNA), die in jeder Zelle bei der Bildung von Proteinen (Proteinbiosynthese) eine wichtige Rolle spielen, indem sie einzelne Aminosäuremoleküle transferieren. Die Länge reifer tRNA-Stränge liegt in der Regel zwischen 73 und 95 Nukleotiden. Über das Basentriplett ihres Anticodons vermitteln sie passend zu einem Codon auf der mRNA bei der Translation am Ribosom jeweils die richtige Aminosäure entsprechend dem Genetischen Code.
Struktur
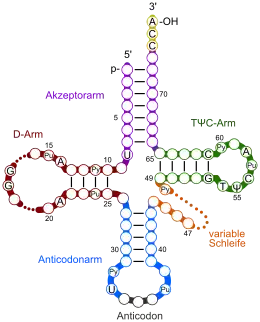
Pu: Purinbase
Py: Pyrimdinbase
T: Thymidin
Ψ: Pseudouridin
In der Regel bestehen tRNA-Moleküle aus 73 bis 95 Nukleotiden eines einzelnen RNA-Stranges. Neben den vier Grundbausteinen der RNA (Adenosin, Uridin, Cytidin und Guanosin) enthält tRNA eine Reihe unterschiedlich modifizierter Standardbasen. So hat man beispielsweise die Nukleoside Dihydrouridin (D), Inosin (I), 2-Thiouridin (s2U)[1], 4-Thiouridin (s4U),[2] Pseudouridin (Ψ), N4-Acetylcytidin (ac4C) und 5-Methyl-Uridin (Thymidin) (T) identifiziert.
In jedem tRNA-Molekül treten Paarungen konjugierender Nukleinbasen auf, sie bilden so doppelsträngige Bereiche aus. In einer zweidimensionalen Darstellung hat tRNA eine kleeblattartige Sekundärstruktur. An dieser Struktur lassen sich ein Stamm und drei Schleifen unterscheiden: eine Dihydrouridin-, eine TΨC- und eine Anticodonschleife (siehe Abbildung).
- D-Arm (Dihydrouridin-Arm): Die sogenannte Dihydrouridin-Schleife am Ende des D-Arms verdankt ihren Namen den häufig in ihr enthaltenen Dihydrouracilresten. In Abbildungen wird sie in der Regel links dargestellt. Jedoch enthält nicht in jedem Fall ein Dihydrouridin-Arm auch die namensgebenden Dihydrouracilreste.[3] Die Dihydrouridin-Schleife dient vor allem der Erkennung der tRNA durch die Aminoacyl-tRNA-Synthetase.
- Anticodonarm: Auf der Anticodonschleife befindet sich ein spezifisches Basentriplett, das sogenannte Anticodon. Die Schleife besteht immer aus fünf konjugierenden Paaren und sieben ungepaarten Basen, von denen drei das Anticodon bilden. Dieses interagiert mit dem komplementären Codon der abzulesenden mRNA (vgl. Abschnitt Funktion).
- T-Schleife (TΨC-Schleife): Die in Abbildungen rechts dargestellte TΨC-Schleife enthält sieben ungepaarte Basen, von denen drei die namensgebenden Sequenz TΨC bilden. Dabei steht T für (Ribo-)Thymidin, Ψ für Pseudouridin und C für Cytidin.
- Variable Schleife: Zwischen dem TΨC-Arm und dem Anticodonarm liegt die sogenannte variable Schleife; je nach tRNA ist diese unterschiedlich lang.
- Akzeptorstamm: Der Akzeptorstamm ist an seinem 5’-Ende phosphoryliert. Sein 3’-Ende zeigt immer die Sequenz CCA-3’. Das Adenosin (A) am 3’-Ende präsentiert seine Ribose, was eine wichtige Rolle spielt. Denn an diesem Ende, der 3’-OH-Gruppe der Ribose, wird die Carboxygruppe einer korrespondierenden Aminosäure verestert und damit an die tRNA gebunden (vgl. Abschnitt Funktion).
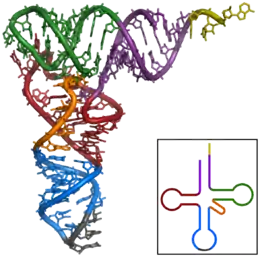
Am häufigsten findet man die modifizierten Basen unter den nicht konjugierten Basen der Kleeblattstruktur. Die tatsächliche dreidimensionale Tertiärstruktur ähnelt eher einer „L“-Form, deren zwei vorkragenden Abschnitte von dem Aminosäuren-Akzeptorstamm und der Anticodonschleife gebildet werden. Diese funktionell wichtigen Regionen liegen also so weit wie möglich voneinander entfernt. Die dreidimensionale Struktur von tRNA aus Hefe wurde bereits 1974 in einer Auflösung von 300 pm (3 Å) unabhängig von zwei Gruppen um Rich und Klug aufgeklärt.[4][5] 2000 wurde eine Struktur mit einer verbesserten Auflösung von 1,93 Å publiziert.[6]
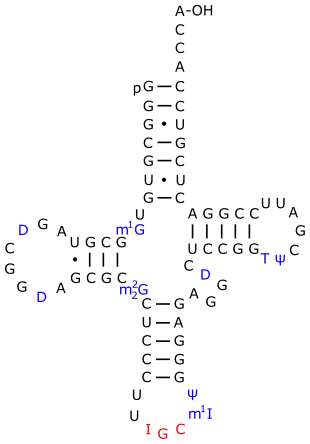 Eine tRNAAla aus S. cerevisiae
Eine tRNAAla aus S. cerevisiae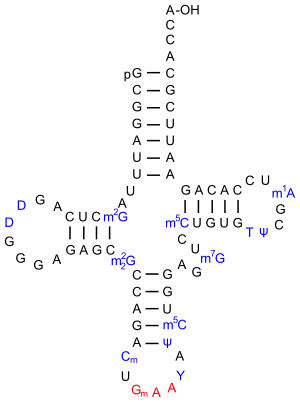 Eine tRNAPhe aus S. cerevisiae[6]
Eine tRNAPhe aus S. cerevisiae[6]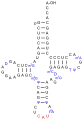 Eine tRNAMet, nicht die Initiator-tRNA (siehe unten), aus S. cerevisiae
Eine tRNAMet, nicht die Initiator-tRNA (siehe unten), aus S. cerevisiae
Funktion
Bei der Translation in der Proteinbiosynthese muss im Ribosom entsprechend dem genetischen Code zu jedem Basentriplett auf der mRNA die passende Aminosäure an die Peptidkette angehängt werden. Diese Aufgabe wird durch die tRNA vermittelt. Dazu gibt es für jede Aminosäure mindestens eine, häufig aber auch mehrere verschiedene tRNAs.
Beladung der tRNA mit einer Aminosäure
Unter ATP-Verbrauch werden tRNAs abhängig von ihrer Sequenz durch die jeweilige Aminoacyl-tRNA-Synthetase am 3’-Ende spezifisch mit der zugehörigen Aminosäure beladen. Dazu wird an die 3’-Hydroxygruppe der Ribose des Adenosins die Carboxygruppe der Aminosäure in Esterbindung angehängt, so dass eine Aminoacylgruppe entsteht. Häufig erkennt die Aminoacyl-tRNA-Synthetase dazu das Anticodon auf der tRNA. Es können aber auch andere Strukturelemente bei der Erkennung eine Rolle spielen, hauptsächlich der Akzeptorstamm.
Ein besonderer Fall sind die tRNAs, die mit Alanin beladen werden (tRNAAla). Unabhängig vom Organismus weist die tRNAAla ein G–U-Basenpaar an den Positionen 3 bzw. 70 im Akzeptorstamm auf.[7] Wird eine dieser beiden Basen mit einer anderen durch (zielgerichtete) Mutagenese ausgetauscht, kann die resultierende tRNA nicht mehr durch die Alanyl-tRNAAla-Synthetase mit Alanin beladen werden.
Zunächst wurde angenommen, dass in allen Organismen für jede Aminosäure genau eine Aminoacyl-tRNA-Synthetase existiert, die alle zur entsprechenden Aminosäure gehörenden tRNAs beladen kann. Später wurde aber entdeckt, dass einigen Organismen eine oder mehrere Aminoacyl-tRNA-Synthetasen fehlen, diese aber dennoch die entsprechenden Aminosäuren in ihre Proteine einbauen können und auch die nötigen tRNAs besitzen. Diese müssen also einen anderen Mechanismus zur Beladung dieser tRNAs besitzen. So fehlt in vielen Archaea, Bakterien, Chloroplasten und Mitochondrien eine Aminoacyl-tRNA-Synthetase für Glutamin. Stattdessen bindet die Aminoacyl-tRNA-Synthetase für Glutamat dieses sowohl an tRNA für Glutamat als auch an tRNA für Glutamin. Das „fehlerhaft“ an die tRNA für Glutamin gebundene Glutamat wird anschließend durch eine Transamidase (Glu-tRNAGln-Amidotransferase)[8] in Glutamin umgewandelt.[9]
Einbau von Aminosäuren in die naszierende Kette
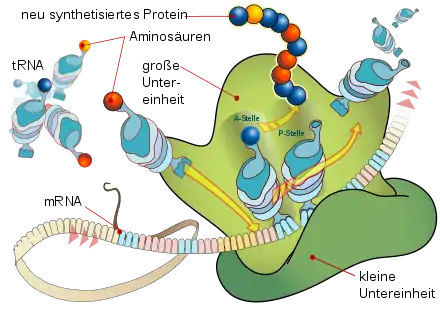
Die aminoacylierten tRNAs werden von den Ribosomen für die Proteinbiosynthese genutzt. Passt das Anticodon zum entsprechenden Basencodon der Boten-RNA (mRNA), so kann sich die tRNA dort anlagern, und die herantransportierte Aminosäure an das entstehende Protein anknüpfen.
Entsprechend dem genetischen Code müsste für jedes Basentriplett, das eine Aminosäure codiert und kein Stopcodon darstellt, eine tRNA existieren – in der Regel also 61.[10] Es wurde aber festgestellt, dass die Zahl der tRNA davon deutlich nach unten abweicht. Die genaue Anzahl unterscheidet sich in den Organismen, jedoch sind es nicht weniger als 31 und nicht mehr als 41.[11][12] Dennoch werden in allen Organismen alle Basentripletts zur Proteincodierung verwendet. Diese Abweichung wird durch die Wobble-Theorie erklärt.
| Base des Anticodons (tRNA) | Base des Codons (mRNA) |
|---|---|
| C | G |
| A | U |
| G | U oder C |
| I | U, C oder A |
| U | A oder G; in Chloroplasten und Mitochondrien auch U oder C[13] |
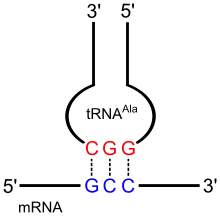
Diese auf Francis Crick zurückgehende Theorie besagt, dass die ersten beiden Basen des Codons und die letzten beiden Basen des Anticodons nach „klassischer“ Watson-Crick-Basenpaarung Wasserstoffbrückenbindungen ausbilden; G paart damit immer mit C bzw. A mit U.[14] Nach Cricks Untersuchungen kann die Möglichkeit der Basenpaarung zwischen der dritten Base des Codons und der ersten Base des Anticodons erweitert werden und folgt nicht dieser strengen Regel. Man spricht auch von der „Wobble-Position“.
Wenn beispielsweise die erste Base des Anticodons ein U ist, kann diese ein A oder G des Codons erkennen. Manchmal ist diese erste Base auch ein Inosin, es vermag sowohl mit U, C als auch A des Codons zu interagieren. Die Ergebnisse sind in rechts stehender Tabelle zusammengefasst.
Häufig ist die erste Base des Anticodons modifiziert. Dies wirkt sich auf die Erkennung der korrespondierenden dritten Base des Codons aus.[13] Ist beispielsweise die erste Base des Anticodons ein Lysidin (k2C), wird diese in Bakterien nur von einem A erkannt. In Mitochondrien mancher Eukaryoten basenpaart 5-Formylcytidin (f5C) mit A oder G. In Stachelhäutern und den Mitochondrien von Tintenfischen hat man entdeckt, dass 7-Methylguanosin (m7G) mit allen vier Standardbasen interagieren kann.
Demnach sind durch die Tatsache, dass der genetische Code ein degenerierter Code ist, bei dem mehrere Basentripletts für die gleiche Aminosäure codieren können, häufig nur zwei Basen für die Erkennung nötig, da sich Basentripletts für die gleiche Aminosäure oft nur in einer Base unterscheiden.
Initiator-tRNAs
Bei der Synthese eines Polypeptids stellt der Startvorgang (Initiation) andere Anforderungen an die tRNA als die Verlängerung (Prolongation) der Kette. Indem die Synthese in der Regel bei einem Codon AUG (codierend für Methionin) startet, benötigt die Zelle für das Startcodon AUG eine spezielle zur Initiation geeignete tRNA. Diese auch Initiator-tRNA genannte tRNAi unterscheidet sich von einer anderen methioninspezifischen tRNAMet, die für die Erkennung des Codons AUG innerhalb eines Leserasters notwendig ist. Obwohl beide tRNAs von derselben Methionyl-tRNA-Synthetase mit Methionin beladen werden, kann tRNAMet nicht für das Startcodon verwendet werden, sondern nur tRNAiMet.[15]
In Bakterien, beispielsweise E. coli, wird die Initiator-tRNA als tRNAifMet angegeben.[16] Das „f“ im Namen gibt an, dass die mit Methionin beladene tRNA mit einem Formylrest modifiziert wird. Diese Reaktion katalysiert eine Methionyl-tRNA-Formyltransferase (EC 2.1.2.9), dabei wird N10-Formyltetrahydrofolat (10-CHO-THF) zu Tetrahydrofolsäure (THF) umgesetzt. Die tRNAifMet unterscheidet sich in wichtigen Punkten von der tRNAMet, so dass letztere nicht von der Formyltransferase erkannt und daher nur tRNAifMet für die Initiation verwendet wird. So findet keine Basenpaarung zwischen dem ersten Cytosin und dem Adenin im Akzeptorstamm statt (vgl. Bild). Im Anticodonstamm gibt es drei aufeinanderfolgende G–C-Basenpaare. Schließlich hat die Initiator-tRNA in der D-Schleife eine CCU-Sequenz. Zusammen sorgen diese Unterschiede für eine leicht geänderte Konformation.[17]
Die Initiator-tRNA in Eukaryoten, sowie in den Archaeen, wird nicht formyliert. Nach der Translation kann das gebildete Protein modifiziert werden. So wird in Bakterien der N-terminale Formylrest in der Regel abgespalten. Auch das Methionin am N-Terminus des Polypeptids kann dann abgespalten werden, ebenso in Eukaryoten und Archaeen.
 eine tRNAifMet aus E. coli. Die Bereiche, die sich von der tRNAMet unterscheiden, sind in grün hervorgehoben.
eine tRNAifMet aus E. coli. Die Bereiche, die sich von der tRNAMet unterscheiden, sind in grün hervorgehoben.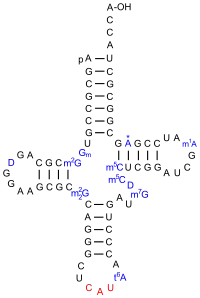 eine tRNAiMet aus S. cerevisiae[18]
eine tRNAiMet aus S. cerevisiae[18]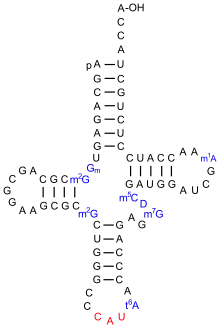 eine tRNAiMet des Menschen
eine tRNAiMet des Menschen
Biosynthese von tRNA
tRNA-Gene
Organismen variieren in der Anzahl ihrer tRNA-Gene auf ihrem Genom. Der Fadenwurm Caenorhabditis elegans, ein typischer Modellorganismus für genetische Studien, besitzt 29.647[19] Gene in seinem Zellkern-Genom, wobei 620 davon für tRNA codieren.[20][21] Die Backhefe besitzt 275 tRNA-Gene in ihrem Genom.
In Prokaryoten sind meist mehrere Gene in Operons zusammengefasst. Die Gene können für Proteinsequenzen oder verschiedene RNA-Produkte codieren, darunter auch tRNA. In der Regel werden so mehrere tRNA auf einem Operon zusammengefasst und diese können auch proteincodierende Gene enthalten.
Bei Eukaryoten muss zwischen tRNA-Genen auf der DNA im Zellkern und tRNA-Genen auf der DNA von Mitochondrien (sowie DNA-haltigen Hydrogenosomen) oder Plastiden wie Chloroplasten unterschieden werden. In Plastiden sind die Gene ähnlich wie bei Prokaryoten in Operons organisiert (siehe Endosymbiontentheorie). Die Ausstattung mit tRNA-Genen (z. B. 30 tRNA-Gene bei Marchantia polymorpha) beinhaltet alle tRNA-Gruppen, die für die Proteinsynthese in Plastiden erforderlich sind.
Jedoch liegt nicht immer ein vollständiger Satz an tRNA-Genen in Mitochondrien vor, so dass – abhängig von der Spezies – tRNAs aus dem Cytosol in das Organell importiert werden müssen. Während in fast allen Opisthokonta (z. B. im Menschen oder in der Hefe) alle mitochondrialen tRNA-Gene durch die mitochondriale DNA codiert werden, müssen beispielsweise in Nesseltieren oder Apicomplexa die meisten tRNAs importiert werden.[22]
Transkription der tRNA
Prokaryoten besitzen nur eine RNA-Polymerase. Diese transkribiert den Leserahmen eines Operons, so dass in der Regel eine polycistronische mRNA entsteht. Aus dieser müssen ggf. die tRNAs durch endonukleolytische Schnitte in den intercistronischen Bereichen herausgelöst werden.
In Eukaryoten wird die tRNA im Zellkern durch RNA-Polymerase III transkribiert.[23] Proteincodierende Gene werden dort hingegen durch RNA-Polymerase II transkribiert. Die tRNA in Plastiden wird durch eine RNA-Polymerase transkribiert, die der von Prokaryoten ähnelt (siehe auch Endosymbiontentheorie). Diese RNA-Polymerase wird auch im Plastidengenom codiert, während eine weitere aus dem Cytoplasma importiert werden muss. In den Mitochondrien gibt es zwei RNA-Polymerasen, die beide importiert werden müssen. Sie sind mit dem kerncodierten plastidären Enzym verwandt.
In Archaeen existiert für die Transkription wie bei den Prokaryoten nur eine RNA-Polymerase. Im Vergleich zu Prokaryoten ist diese aber komplexer. Größe, Anzahl der Untereinheiten und deren Aminosäuresequenzen sind eher mit den eukaryotischen RNA-Polymerasen vergleichbar.
Reifung der tRNA-Vorläufer
Die durch die Transkription entstehenden prä-tRNAs können Introns beinhalten. In Bakterien findet das Spleißen dieser Introns autokatalytisch statt, wogegen die Introns in Eukaryoten und Archaeen durch tRNA Splicing-Endonuklease entfernt werden.[24]
Bei Eukaryoten findet sowohl die tRNA-Synthese als auch erste posttranskriptionale Prozessierungen wie das Trimmen der 5'- und 3'-Enden, Basenmodifikationen und das posttranskriptionelle Anfügen der 3'-CCA-Sequenz im Zellkern statt. Der Export in das Cytosol wird durch Proteine wie Los1 vermittelt. Im Cytosol erfolgt an der Außenmembran von Mitochondrien das Spleißen, falls Introns vorhanden sind, und auch weitere Modifikationen, bis die tRNA gereift ist.[25]
Entstehung der tRNAs
Die obere Hälfte der tRNA (aus dem D-Arm und dem Akzeptor-Stamm mit der 5'-terminierenden Phosphateinheit und der 3'-terminierenden CCA-Gruppe) und die untere Hälfte (aus dem T-Arm und dem Anticodon-Arm) sind in Struktur und Funktion unabhängige Einheiten. Es wird vermutet, dass sich die obere Hälfte zuerst entwickelt hat, wobei die CCA-Gruppe ursprünglich – d. h. in einer frühen RNA-Welt – tRNA-ähnliche Moleküle zum Zweck der Replikation markiert haben könnte (ein solcher Marker heißt im Englischen genomic tag). Die untere Hälfte könnte später als eine Erweiterung entstanden sein, z. B. als die Proteinbiosynthese startete und die RNA-Welt in eine Ribonukleoprotein-Welt (RNP-Welt) überging. Ein solches Szenario wird im Englischen genomic tag hypothesis genannt. Tatsächlich haben tRNA-ähnliche Moleküle noch heute eine wichtige katalytische Rolle (ergo als Ribozyme) bei der Replikation. Sie können als molekulare (oder chemische) Fossilien betrachtet werden, die wie die „lebenden Fossilien“ Hinweise auf den frühen Verlauf der Evolution geben.[26]
Variationen von tRNA-Formen im Tierreich
Die oben dargestellte „klassische“ Form der tRNA mit dreiarmiger „Kleeblatt“-Struktur wurde bei bisherigen Untersuchungen bei den meisten tRNAs bestätigt. In einigen Stämmen wurden aber stark modifizierte mitochondriale tRNAs (mt-tRNAs) gefunden, bei denen verschiedene Teile der normalen Struktur stark modifiziert oder ganz verloren waren. Dabei erscheint es unwahrscheinlich, dass es sich um durch Mutationen funktionslos gewordene Pseudogene handelt. Bei einer tRNA-Sequenz fehlt der D-Arm im gesamten Tierreich,[27] was darauf hindeutet, dass diese Sequenz bereits beim Vorfahren aller heutigen Tiere modifiziert war. Besonders stark modifizierte mitochondriale tRNA-Strukturen wurden bei Nematoden[28] und Spinnentieren[29] identifiziert. Einige mt-tRNAs haben dabei große Teile der üblichen Strukturen verloren, ohne ihre Funktionsfähigkeit einzubüßen. Meist ist anstelle der verlorengegangenen Struktur eine Ersatzstruktur, z. B. eine zusätzliche Schleife (TV-Ersatzschleife), ausgebildet. Warum der Verlust einer sonst hoch konservierten Struktur, deren Mutation allen Annahmen nach fast immer letal sein müsste, hier ausnahmsweise doch möglich ist, ist bisher nicht aufgeklärt.
Siehe auch
Literatur
- Reginald Garrett, Charles M. Grisham: Biochemistry. (International Student Edition). 4. Auflage. Cengage Learning Services, 2009, ISBN 978-0-495-11464-2.
- Peter Karlson, Detlef Doenecke, Jan Koolman, Georg Fuchs, Wolfgang Gerok: Karlsons Biochemie und Pathobiochemie. 15. Auflage. Georg Thieme, 2005, ISBN 3-13-357815-4, S. 142.
- Dieter Soll (Hrsg.), Uttam L. RajBhandary (Hrsg.), T. L. Rajbhandary: tRNA: Structure, Biosynthesis, and Function. Asm Press, 1995, ISBN 1-55581-073-X.
- E. M. Phizicky, A. K. Hopper: tRNA biology charges to the front. In: Genes Dev. 24(17), 2010, S. 1832–1860. PMID 20810645; PMC 2932967 (freier Volltext).
- M. A. Rubio, A. K. Hopper: Transfer RNA travels from the cytoplasm to organelles. In: Wiley Interdiscip Rev RNA. 2(6), 2011, S. 802–817. PMID 21976284; doi:10.1002/wrna.93
- Paul Schimmel: The emerging complexity of the tRNA world: mammalian tRNAs beyond protein synthesis. In: Nature Reviews. Molecular Cell Biology. 6. September 2017, doi:10.1038/nrm.2017.77, PMID 28875994.
- Kunal Chatterjee, Regina T. Nostramo, Yao Wan, Anita K. Hopper: tRNA dynamics between the nucleus, cytoplasm and mitochondrial surface: Location, location, location. In: Biochimica Et Biophysica Acta. 27. November 2017, doi:10.1016/j.bbagrm.2017.11.007, PMID 29191733.
Weblinks
- The RNA modification database. (Memento vom 7. April 2013 im Internet Archive) Umfangreiche Datenbank über modifizierte Basen in tRNAs
- Datenbank mitochondrialer tRNAs aus Säugern
- The Genomic tRNA Database (GtRDB)
- Alexandra Kühnlein et al.: tRNA sequences can assemble into a replicator. In: eLife, 10, 2. März 2021, S. e63431, doi:10.7554/eLife.63431. Dazu:
- Origin of life: The chicken-and-egg problem. ScienceDaily, 2. März 2021
Einzelnachweise
- R. K. Kumar, D. R. Davis: Synthesis and studies on the effect of 2-thiouridine and 4-thiouridine on sugar conformation and RNA duplex stability. In: Nucleic Acids Research. Band 25, Nummer 6, März 1997, S. 1272–1280. PMID 9092639, PMC 146581 (freier Volltext).
- Yuchen Liu et al.: Biosynthesis of 4-Thiouridine in tRNA in the Methanogenic Archaeon Methanococcus maripaludis. In: The Journal of Biological Chemistry, 287, 17. August 2012, S. 36683–36692, doi:10.1074/jbc.M112.405688
- H. Grosjean, R. Gupta, E. S. Maxwell: Modified nucleotides in archaeal RNAs. In: P. Blum (Hrsg.): Archaea: new models for prokaryotic biology. Caister Academic Press, Norfolk, UK 2008, S. 171–196.
- S. H. Kim et al.: Three-dimensional tertiary structure of yeast phenylalanine transfer RNA. In: Science, 185(149), 1974, S. 435–440. PMID 4601792; doi:10.1126/science.185.4149.435.
- J. D. Robertus et al.: Structure of yeast phenylalanine tRNA at 3 Å resolution. In: Nature. 250(467), 1974, S. 546–551. PMID 4602655; doi:10.1038/250546a0.
- H. Shi, P. B. Moore: The crystal structure of yeast phenylalanine tRNA at 1.93 Å resolution: a classic structure revisited. In: RNA, 6(8), 2000, S. 1091–1105. PMID 10943889; PMC 1369984 (freier Volltext).
- Reginald Garrett, Charles M. Grisham: Biochemistry. (International Student Edition). 4. Auflage. Cengage Learning Services, 2009, ISBN 978-0-495-11464-2, S. 958.
- K. Horiuchi, M. Harpel, L. Shen, Y. Luo, K. Rogers, R. Copeland: Mechanistic Studies of Reaction Coupling in Glu-tRNAGln Amidotransferase. In: Biochemistry. Band 40, Nr. 21, Mai 2001, S. 6450–6457, doi:10.1021/bi002599l.
- Katharina Munk (Hrsg.): Taschenlehrbuch Biologie: Genetik. 1. Auflage. Thieme Verlag, 2010, ISBN 978-3-13-144871-2, S. 188.
- Peter Karlson, Detlef Doenecke, Jan Koolman, Georg Fuchs, Wolfgang Gerok: Karlsons Biochemie und Pathobiochemie. 15. Auflage. Thieme, 2005, S. 142.
- F. Crick: The origin of the genetic code. In: J Mol Biol. Band 38, Nr. 3, 1968, S. 367–379, doi:10.1016/0022-2836(68)90392-6, PMID 4887876.
- H. Lodish et al.: Molecular Biology of the Cell. 5. Auflage. WH Freeman, New York 2004, ISBN 0-7167-3136-3. (Excerpt) (englisch)
Harvey Lodish et al.: Molekulare Zellbiologie. Spektrum Akademischer Verlag, 2001, ISBN 3-8274-1077-0. - A. Ambrogelly et al.: Natural expansion of the genetic code. In: Nature Chemical Biology. 3 (1), 2007, S. 29–35. PMID 17173027; doi:10.1038/nchembio847.
- Reginald Garrett, Charles M. Grisham: Biochemistry. (International Student Edition). 4. Auflage. Cengage Learning Services, 2009, ISBN 978-0-495-11464-2, S. 959.
- Reginald Garrett, Charles M. Grisham: Biochemistry. (International Student Edition). 4. Auflage. Cengage Learning Services, 2009, ISBN 978-0-495-11464-2, S. 977.
- Reginald Garrett, Charles M. Grisham: Biochemistry. (International Student Edition). 4. Auflage. Cengage Learning Services, 2009, ISBN 978-0-495-11464-2, S. 966.
- Donald Voet, Judith G. Voet: Biochemie. Wiley-VCH, 1994, ISBN 3-527-29249-7, S. 928.
- S. E. Kolitz, J. R. Lorsch: Eukaryotic initiator tRNA: finely tuned and ready for action. In: FEBS Letters. 584 (2), 21. Januar 2010, S. 396–404 (doi:10.1016/j.febslet.2009.11.047, PMC 2795131 (freier Volltext). PMID 19925799).
- WormBase: Release Notes – WS187. 25. Januar 2008.
- J. Spieth, D. Lawson: Overview of gene structure. In: WormBook, 2006, S. 1–10. PMID 18023127; PDF (englisch)
- L. H. Hartwell et al.: Genetics: From Genes to Genomes. 2. Auflage. McGraw-Hill, New York 2004, S. 264.
- André Schneider: Mitochondrial tRNA import and its consequences for mitochondrial translation. In: Annu Rev Biochem. 80, 2011, S. 1033–1053. PMID 21417719; doi:10.1146/annurev-biochem-060109-092838
- R. J. White: Regulation of RNA polymerases I and III by the retinoblastoma protein: a mechanism for growth control? In: Trends in Biochemical Sciences. 22(3), 1997, S. 77–80. PMID 9066256; doi:10.1016/S0968-0004(96)10067-0
- J. Abelson, C. R. Trotta, H. Li: tRNA Splicing. In: J Biol Chem, 273(21), 1998, S. 12685–12688. PMID 9582290; jbc.org (PDF; englisch)
- A. K. Hopper, D. A. Pai, D. R. Engelke: Cellular dynamics of tRNAs and their genes. In: FEBS Lett. 584(2), 2010, S. 310–317. PMID 19931532; PMC 2818515 (freier Volltext).
- Nancy Maizels, Alan M. Weiner: The Genomic Tag Hypothesis – What Molecular Fossils Tell Us about the Evolution of tRNA. In: The RNA World. 2. Auflage. Cold Spring Harbor Laboratory Press, 1999, ISBN 0-87969-561-7. rna.cshl.edu (PDF; 280 kB)
- D. R. Wolstenholme: Animal mitochondrial DNA: structure and evolution. In: D. R. Wolstenholme, K. W. Jeon (Hrsg.): Mitochondrial genomes. Academic Press, New York 1992, S. 173–216.
- Y. Watanabe et al.: Primary and higher order structures of nematode (Ascaris suum) mitochondrial tRNAs lacking either the T or D stem. In: Journal of Biological Chemistry, 269(36), S. 22902–22906. PMID 8077242; jbc.org (PDF; englisch)
- Susan E. Masta, Jeffrey L. Boore: Parallel Evolution of Truncated Transfer RNA Genes in Arachnid Mitochondrial Genomes. In: Molecular Biology and Evolution, 25(5), 2008, S. 949–959. PMID 18296699; oxfordjournals.org (PDF; englisch)