Atmungskette
Die Atmungskette ist ein Teil des Energiestoffwechsels der meisten Lebewesen. Einerseits wird mit dem Ausdruck Atmungskette ein Stoffwechselweg bezeichnet, nämlich eine Kette von nacheinander stattfindenden biochemischen Redoxreaktionen, die den Lebewesen zur Energiegewinnung dient, andererseits auch die Gesamtheit der an dem Stoffwechselweg teilnehmenden Proteinkomplexe.
| Übergeordnet |
| Elektronentransportkette Zellatmung Oxidative Phosphorylierung |
| Untergeordnet |
| Atmungskette der Zellmembran Atmungskette der Mitochondrienmembran |
| Gene Ontology |
|---|
| QuickGO |
| Übergeordnet |
| Zellmembran Mitochondrienmembran |
| Untergeordnet |
| Komplex I Komplex II Komplex III Komplex IV |
| Gene Ontology |
|---|
| QuickGO |
Die Atmungskette ist ein Spezialfall einer Elektronentransportkette und bildet zusammen mit der Chemiosmosis den Prozess der oxidativen Phosphorylierung.
Durch NADH, FMNH2 und FADH2 angelieferte Elektronen werden in einer Reihe von Redoxvorgängen auf ein Oxidationsmittel übertragen. So wird – insbesondere bei Eukaryoten – die exergonische Reaktion von Wasserstoff (H2) und Sauerstoff (1/2 O2) zu Wasser in Einzelschritte aufgeteilt. Anstelle einer unter Umständen explosionsartigen Wärmeentwicklung wird die freiwerdende Energie dazu genutzt, aus ADP und Phosphat die universelle „Energiewährung“ der Zelle, ATP, zu synthetisieren (oxidative Phosphorylierung). Die an die Wasserstoff- und Elektronenüberträger NADH und FADH2 gebundenen Elektronen und der daran gebundene Wasserstoff entstammen der Oxidation externer Elektronendonatoren, etwa – mittels des Citratzyklus – dem Abbau von Fettsäuren und der Glykolyse.
Bei Eukaryoten befindet sich die Atmungskette in der inneren Membran der Mitochondrien, bei Prokaryoten in der Zellmembran. Dort kommen auch andere Elektronendonatoren als Fette und Zucker sowie andere Elektronenakzeptoren als Sauerstoff vor.
Atmungskette als Elektronentransportkette
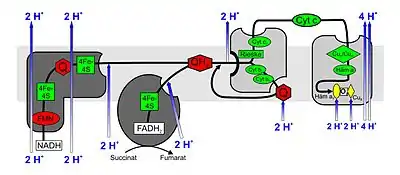
Elektronentransportketten bestehen aus einer Reihe hintereinander geschalteter Redox-Moleküle, die in der Lage sind, Elektronen aufzunehmen bzw. abzugeben. Über diese Kette werden Elektronen von höheren Energieniveaus auf niedrigere weitergegeben, sie fallen sozusagen in Stufen bergab, wobei die einzelnen Redox-Moleküle ein zunehmend niedriges Energieniveau haben. Bei Eukaryoten sind an der Reaktionskette nacheinander die Enzym-Komplexe I bis IV und die Wasserstoff- bzw. Elektronenüberträger Ubichinon (Coenzym Q) und Cytochrom c, die in die innere Mitochondrienmembran eingelagert sind, beteiligt. Die an der Elektronentransportkette beteiligten Proteine (Komplexe I–IV) sowie die Elektronenüberträger Ubichinon und Cytochrom c, bilden ein (komplexes) Redoxsystem.
Der Elektronentransport ist mit der Aufnahme und Abgabe von Protonen verbunden. Durch die räumliche Organisation dieser Prozesse oder mittels durch den Elektronenfluss verursachte konformationelle Änderungen der Proteinstruktur werden Protonen (H+) aus der Matrix (dem Inneren) der Mitochondrien in den Intermembranraum (zwischen innerer und äußerer Mitochondrienmembran, siehe unten) „transportiert“ (real oder als Nettoeffekt). Dabei entsteht ein „elektrochemischer Protonengradient“ (zusammengesetzt aus dem Konzentrationsunterschied der Protonen und dem durch den Austransport der positiv geladenen Protonen erzeugten negativen Membranpotential im Inneren der Mitochondrien). Die Energie dieses „elektrochemischen Protonengradienten“ (proton motive force, „pmf“) wird nach der mittlerweile allgemein akzeptierten chemiosmotischen Theorie von Peter D. Mitchell von einer ATP-Synthase durch Rückdiffusion von Protonen in der inneren Mitochondrienmembran dazu genutzt, ATP aus ADP und anorganischem Phosphat zu synthetisieren (in seltenen Fällen auch Fructose). Diese Phosphorylierung wird wegen der Kopplung an die Atmungskette Atmungskettenphosphorylierung oder oxidative Phosphorylierung (Abkürzung: Oxphos) genannt.[1]
Funktionen der Komplexe der Atmungskette
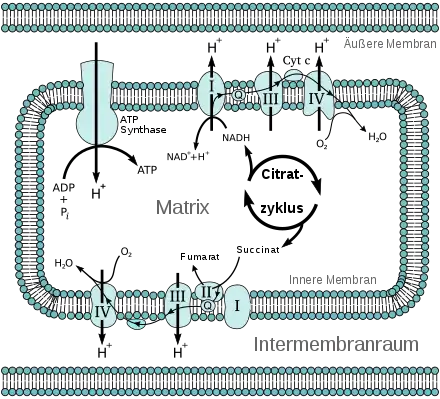
Ein Mitochondrium enthält außer seiner äußeren noch eine innere Membran. Den Raum zwischen diesen beiden Membranen nennt man Intermembranraum (perimitochondrialer Raum).
Drei der vier Komplexe der Atmungskette durchspannen jeweils die innere Mitochondrienmembran vollständig (integral), Komplex II hingegen endet „blind“ (peripher). Es wird ein Protonenkonzentrationsunterschied zwischen dem Intermembranraum und dem Inneren (Matrix) des Mitochondriums erzeugt, der dann in der ATP-Synthase zur Synthese von ATP genutzt wird.
Komplex I
NADH: Ubichinon–Oxidoreduktase oder NADH-Dehydrogenase. Dieser sehr große Enzymkomplex (940 kDa) reduziert mittels NADH, vor allem aus dem Citratzyklus, Ubichinon (UQ oder Q) zum Ubihydrochinon, auch Ubichinol (UQH2 oder QH2) genannt. Der Komplex besteht aus zwei Teilen, die zusammen seine charakteristische L-Form ergeben. In einer Einheit werden flavinhaltige Nukleotide (FMN) sowie Eisen-Schwefel-Zentren als prosthetische Gruppen benötigt, um die Redoxreaktion zu katalysieren. Durch den mit den Redoxreaktionen verbundenen Elektronentransport werden pro oxidiertem NADH netto 3–4 Protonen in den Intermembranraum gepumpt. Es wird vermutet, dass die Kopplung mit dem Protonentransport durch konformationelle Änderung des Enzyms erfolgt.[2]
Komplex II
Succinat: Ubichinon–Oxidoreduktase oder Succinat-Dehydrogenase. Der Komplex II ist das Enzym Succinat-Dehydrogenase aus dem Citratzyklus. Bei der Reaktion im Citratzyklus wird Succinat zu Fumarat oxidiert. FAD liegt als prosthetische Gruppe im Enzym vor. Es überträgt seine Elektronen im Komplex II auf Ubichinon, das zu Ubihydrochinon reduziert wird. Auch Komplex II enthält Eisen-Schwefel-Zentren, wie Komplex I; allerdings werden keine Protonen in den Intermembranraum gepumpt.
Komplex III
Ubihydrochinon (Ubichinol): Cytochrom c–Oxidoreduktase oder Cytochrom-c-Reduktase. An Komplex III trägt der Q-Zyklus durch asymmetrische Absorption und Freigabe von Protonen zur Erzeugung des Protonenkonzentrationsunterschieds bei. Bei der Oxidation von Ubichinol (QH2) zu Ubichinon (Q) werden in einem Zyklus pro abgegebenem Elektron (vom Ubichinol) ein Molekül Cytochrom c reduziert und zwei Protonen in den Intermembranraum abgegeben. Durch das zweite Elektron wird an einer anderen Bindestelle auf der Seite der mitochondrialen Matrix ein weiteres Ubichinon erst zum freien Radikal Ubisemichinon (QH), dann zu QH2 reduziert, wobei zwei Protonen aus der Matrix aufgenommen werden.
Nach den beiden Halbzyklen sind pro Ubichinol-Molekül vier Protonen in den Intermembranraum freigesetzt, zwei Protonen aus der mitochondrialen Matrix entfernt und zwei Cytochrom c reduziert. Im Komplex III findet somit eine Umleitung von einem Zwei-Elektronen-Transporter (Ubichinol) auf einen Ein-Elektronen-Transporter (Cytochrom c) statt.[1][3]
Komplex IV
Cytochrom c: O2-Oxidoreduktase oder Cytochrom-c-Oxidase. Im Komplex IV wird Cytochrom c oxidiert und Sauerstoff zu Wasser reduziert. Die dabei freigesetzte Energie wird genutzt, um Protonen vom Matrixraum in den Intermembranraum zu pumpen.
Am Komplex IV wird Cytochrom c oxidiert und dabei ein Elektron auf den Komplex übertragen. Nach der sukzessiven Übertragung von vier Elektronen (e−) kann ein gebundenes Sauerstoffmolekül zu zwei Wassermolekülen (H2O) reduziert werden. Die dabei benötigten vier Protonen (H+) werden aus der Matrix entzogen. Die bei der Reduktion von Sauerstoff zu Wasser frei werdende Energie wird vom Enzym genutzt, um weitere vier Protonen pro Sauerstoffmolekül von der Matrix über die innere Mitochondrienmembran in den Intermembranraum zu pumpen. Dies geschieht mittels Änderungen der räumlichen Struktur: In einer Konformation hat ein Protein eine hohe Affinität zu H+ und nimmt daher ein Proton auf. In der entgegengesetzten Konformation besteht eine niedrige Affinität, und das Proton wird auf der Membran-Außenseite freigesetzt.[4]
Die Cytochrom-c-Oxidase ist ein Transmembranprotein mit zwei Häm a-Molekülen (Häm a und Häm a3) als prosthetische Gruppen und zwei Kupfer-Zentren (CuA und CuB) als Kofaktoren. Das Enzym ist für nahezu sämtlichen Sauerstoffverbrauch (Bildung von Wasser aus Sauerstoff und Wasserstoff in der Atmungskette) aller sauerstoffatmenden Organismen verantwortlich.
Hemmstoffe der Atmungskette
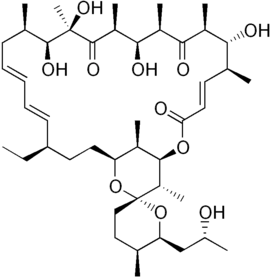
Es wurden eine Reihe von Hemmstoffen identifiziert, die die Elektronentransportkette an unterschiedlichen Stellen inhibiert:[5]
- Rotenon und Amobarbital (Amytal) hemmen den Komplex I (NADH Oxidase). Da aber der Elektronentransfer von FADH2 an Komplex II nicht beeinflusst wird, kann die oxidative Phosphorylierung noch stattfinden.
- Malonat und eine Reihe von Fungiziden (SDHI) hemmen den Komplex II (Succinat-Dehydrogenase).
- Das Antibiotikum Antimycin A hemmt den Komplex III (Cytochrom c-Reduktase); es blockiert die Übertragung von Elektronen vom Cytochrom b auf Cytochrom c1; die Komponenten der Atmungskette vor dem Wirkungsort von Antimycin A im Komplex III bleiben reduziert, alle dahinter bleiben oxidiert. Dadurch werden der Verbrauch von Sauerstoff an Komplex IV und die Synthese von ATP im Komplex V gehemmt.[6] Auch die Fungizide aus der Klasse der Strobilurine hemmen den Komplex III an der Stelle Qo.
- Cyanide, Azide und Kohlenmonoxid hemmen den Komplex IV (Cytochrom c-Oxidase); diese Moleküle blockieren die Bindungsstelle für Sauerstoff. Infolgedessen führt dies zu einem Elektronenstau, wodurch die Komponenten der Atmungskette vollständig reduziert vorliegen und die Atmungskette zum Erliegen kommt. Vergiftungen mit Cyaniden lassen die Venen rot erscheinen, weil der Sauerstoff vom Hämoglobin aus den Arteriolen nicht in die Zellen diffundieren kann, da es dort nicht verbraucht wird. So gelangt sauerstoffreiches Blut in die Venen.
- das Antibiotikum Oligomycin hemmt die ATP-Synthase durch Binden an deren Steg (Fo-Teil, o für olygomycinsensitiv), wodurch der Protonengradient wesentlich langsamer abgebaut wird. Infolgedessen nimmt der Elektronenfluss zum Erhalt dieses Gradienten deutlich ab, und der Sauerstoffverbrauch sinkt. Oligomycin dient auch als Entkoppler. In einschlägiger Literatur wird nicht zwischen F0 (Null) und Fo unterschieden.
- Biguanide wie das Diabetes-Medikament Metformin wirken vermutlich durch eine Hemmung des Komplex I.[7]
Alle bisher angesprochenen Hemmstoffe der Atmungskette führen zu einem verminderten Sauerstoffverbrauch. Anders verhalten sich Hemmstoffe, die als Entkoppler bezeichnet werden.
Entkoppler der Atmungskette
Entkoppler (Protonophore) bauen das mitochondriale Membranpotential (Protonengradient) ab. Damit unterbrechen sie die Verknüpfung von Oxidation und Phosphorylierung. Dies hat zur Folge, dass der Elektronentransport und die Funktionsweise der Komplexe I bis IV vollständig abläuft, der bei diesem Prozess aufgebaute Protonengradient aber durch den Entkoppler aufgehoben wird. Damit kann keine Synthese von ATP erfolgen. Dadurch, dass Protonen nicht mehr gegen einen elektrochemischen Gradienten transloziert werden müssen, laufen die Oxidationsschritte in den Komplexen I bis IV viel schneller ab. Dies führt gleichzeitig zu einem erhöhten Sauerstoffverbrauch.
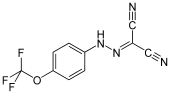
- natürliche Entkoppler:
- Thermogenin, siehe auch braunes Fettgewebe.
- künstliche Entkoppler:
- 2,4-Dinitrophenol ist eine schwache Säure. Sie wird über der inneren Mitochondrienmembran aufgrund des reichen Vorkommens von H+-Ionen (niedriger pH) protoniert, kann dann die Membran passieren und wird intramitochondrial (höherer pH) deprotoniert.
- Carbonylcyanid-m-chlorphenylhydrazon (CCCP)
- Carbonylcyanid-p-trifluoromethoxyphenylhydrazon (FCCP)
- Pentachlorphenol (PCP)
Literatur
- M. Saraste: Oxidative phosphorylation at the fin de siecle. In: Science, Band 283, 1999, Nr. 5407, S. 1488–1493, PMID 10066163.
- Löffler, Petrides: Biochemie des Menschen. 7. Auflage.
Weblinks
- Oxidative phosphorylation – Reference pathway. KEGG (graphische Darstellung der mitochondrialen Atmungsketten-Komplexe, englisch)
- Mitochondrial Pathways: Oxidative Phosphorylation. (englisch)
- Pedro Silva: The chemical logic behind … Respiration and Fermentation. (englisch)
- Elektronentransportkette (animierte Darstellung)
- Electron Transport Chain. Jassal / reactome
Einzelnachweise
- Bruce Alberts u. a.: Molecular biology of the cell. 4. Auflage. New York 2002, S. 773–793
- R. G. Efremov, R. Baradaran, L. A. Sazanov: The architecture of respiratory complex I. In: Nature, Band 465, Nummer 7297, Mai 2010, S. 441–445, doi:10.1038/nature09066. PMID 20505720.
- J.M. Berg, J.L. Tymoczko, L. Stryer: Biochemie. 6. Auflage. Spektrum-Verlag, 2007.
- Bruce Alberts u. a.: Molecular biology of the cell. 4. Auflage. New York 2002, S. 791
- Todd A. Swanson, Sandra I. Kim, Marc J. Glucksman: BRS Biochemistry, Molecular Biology, and Genetics. 5. Auflage. Lippincott Raven, 2010, ISBN 978-0-7817-9875-4, S. 89.
- L. S. Huang, D. Cobessi u. a.: Binding of the respiratory chain inhibitor antimycin to the mitochondrial bc1 complex: a new crystal structure reveals an altered intramolecular hydrogen-bonding pattern. In: Journal of molecular biology. Band 351, Nummer 3, August 2005, S. 573–597, doi:10.1016/j.jmb.2005.05.053. PMID 16024040. PMC 1482829 (freier Volltext).
- H. R. Bridges, V. A. Sirviö u. a.: Molecular features of biguanides required for targeting of mitochondrial respiratory complex I and activation of AMP-kinase. In: BMC biology. Band 14, August 2016, S. 65, doi:10.1186/s12915-016-0287-9, PMID 27506389, PMC 4977651 (freier Volltext).