Ribosomale RNA
Die ribosomale Ribonukleinsäure (rRNA) ist die Ribonukleinsäure, aus der zusammen mit Proteinen die Ribosomen aufgebaut sind.
Eigenschaften
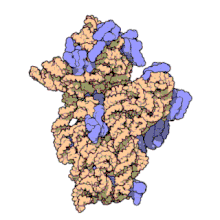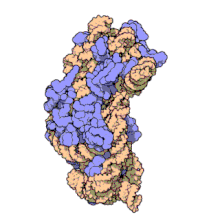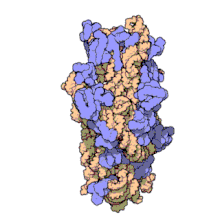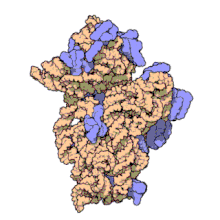
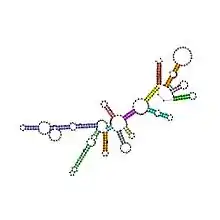
Ribosomale RNA wird im Nucleolus durch Transkription anhand einer DNA-Vorlage erzeugt (der rDNA). Hierbei bewirkt zunächst eine RNA-Polymerase den Aufbau zahlreicher RNA-Stränge gleicher Zusammensetzung.[2] Im Nucleolus wird deren Nukleotidsequenz verändert, wobei manche Teile (ITS-Sequenzen) entfernt, über 200 Nukleinbasen enzymatisch modifiziert werden.[3] Die rRNA bindet anschließend an ribosomale Proteine (ca. 50 Proteine bei Prokaryoten, ca. 80 bei Eukaryoten), wodurch Ribosomen entstehen. Als RNA-bindende Proteine gehören diese Proteinkomplexe zu den Ribonukleoproteinen.
Das Ribosom ist der Ort der Proteinbiosynthese. Drei oder vier verschiedene rRNA-Moleküle sind am Aufbau eines Ribosoms beteiligt. Die ribosomale Ribonukleinsäure hat in diesem Verbund neben einer enzymatischen Funktion (als Ribozym) auch strukturelle und Erkennungsfunktionen. So wird z. B. die Peptidbindung aufeinanderfolgender Aminosäuren von der rRNA katalysiert, während enzymatische Aktivität in Lebewesen sonst meist von Proteinen ausgeübt wird. Um den hohen Bedarf der Zelle an rRNA decken zu können – die rRNA kann bis zu 90 % der Gesamt-RNA einer Zelle ausmachen – findet sich die rDNA-Vorlage mehrfach bis vielfach auf den Chromosomen.
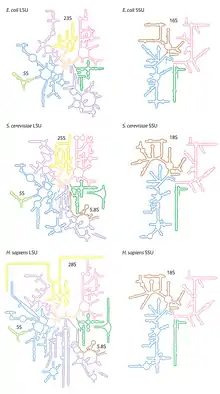
Ribosomen von Prokaryoten enthalten drei unterschiedlich große rRNA-Moleküle,[4][5] die der Eukaryoten vier.[6][Anm. 1] Dabei organisiert sich die rRNA bei allen zellulären Organismen grundsätzlich in zwei ribosomalen Untereinheiten:
- eine große (englisch large subunit, LSU), mit zwei rRNAs bei Prokaryoten und drei rRNAs bei Eukaryoten
- eine kleine (en. small subunit, SSU) mit immer nur einer einzigen rRNA.
Die Größe der rRNAs wird herkömmlich nach ihrem Sedimentationsverhalten in Svedberg (S) angegeben. Diese variiert mit der Anzahl der Nukleinbasen bzw. Nukleotiden der betreffenden rRNA-Moleküle je nach Spezies. Die Angaben der Tabelle sind insofern Beispiele:
| Ribosom | Untereinheit | rRNA | Nukleotide |
|---|---|---|---|
| 70S | LSU: 50S | 23S | 2900 nt |
| 5S | 120 nt | ||
| SSU: 30S | 16S | 1500 nt |
| Ribosom | Untereinheit | rRNA | Nukleotide |
|---|---|---|---|
| 80S | LSU: 60S | 28S | 4718 nt |
| 5,8S | 160 nt | ||
| 5S | 120 nt | ||
| SSU: 40S | 18S | 1874 nt |
Die eukaryotische 5,8S- und 28S-rRNA entsprechen zusammen in Größe und Funktion in etwa der prokaryotischen 23S-rRNA, zuzüglich so genannter Expansionssegmente (en. expansion segment, ES, genauer ES39), die an der Oberfläche des Ribosoms lokalisiert sind und von denen ursprünglich annahm, dass sie nur in Eukaryoten vorkommen. Inzwischen hat man jedoch bei den Lokiarchaeota und Heimdallarchaeota (aus der Gruppe der Asgard-Archaeen, die als die nächsten archaealen Verwandten von Eukaryoten angesehen werden, siehe Eozyten-Hypothese) zwei große (supersized) ESs in ihren 23S-rRNAs gefunden.[7][8] Beim halophilen Archaeon Halococcus morrhuae [en] (Haloarchaeen, Euryarchaeota) enthält die 5S-rRNA ebenfalls eine 108-Nukleotid-Insertion.[9][10]
Die eukaryotische SSU enthält in ihrer 18S-rRNA ebenfalls ESs. SSU-ESs sind aber im Allgemeinen kleiner als LSU-ESs.[11]
Prokaryotische rRNA
Die 16S-rRNA macht zusammen mit verschiedenen Proteinen ca. 2/3 der Masse der kleineren 30S-Untereinheit der prokaryotischen Ribosomen aus und hat eine wichtige Funktion in der Initiationsphase der Translation: Durch Basenpaarung bindet das 3'-Ende der 16S-rRNA an die Shine-Dalgarno-Sequenz der mRNA. Dadurch wird das Startcodon der mRNA in die richtige Position im Ribosom gebracht. Diese Position heißt P-Stelle.
Eukaryotische rRNA
Die Regionen der Chromosomen, die die rDNA beherbergen, lagern sich im Zellkern während der Interphase des Zellzyklus zu einem oder mehreren Nucleoli, auch Kernkörperchen genannt, zusammen. Die rRNA wird dort von der RNA-Polymerase I transkribiert, während mRNA durch RNA-Polymerasen|RNA-Polymerase II transkribiert wird. Dabei wird zunächst eine 45S-Prä-rRNA erzeugt, deren Prozessierung die 18S-, 5,8S- und 28S-rRNAs in gleicher Anzahl liefert. Lediglich die 5S-rRNA wird davon unabhängig an anderer Stelle und zwar durch die RNA-Polymerase III transkribiert. Diese und die ribosomalen Proteine (die im Cytosol synthetisiert wurden) werden zum Kernkörperchen gebracht, wo sich daraus die große und die kleine Untereinheit des Ribosoms bilden, um anschließend aus dem Kern ausgeschleust zu werden. Nackte, d. h. nicht von Proteinen bedeckte RNA würde zu schnell beschädigt werden. Ein spezieller Regulationsmechanismus sorgt dafür, dass die 5S-rRNA in der passenden Menge gebildet wird.
Anwendungen
Die Sequenz der rRNA wird im Zuge phylogenomischer Untersuchungen zur Bestimmung von Verwandtschaftsgraden bestimmt (rRNA und ITS).[12][13][14] Dadurch kann ein phylogenetischer Baum erstellt werden. Andere Marker sind der Elongationsfaktor Tu (EfTu), das Gen der Untereinheit I der Cytochrom-c-Oxidase, das Gen des Cytochrom b der Cytochrom-c-Reduktase, die Gene für die ATP-Synthetase und die Gene von Hitzeschockproteinen.
Die Antibiotika-Klassen der Tetracycline und der Aminoglycoside binden an rRNA.[15][16]
Phylogenetik
Ribosomale RNA erlangte in den letzten Jahrzehnten enorme Bedeutung als Werkzeug zur Aufklärung der Stammesgeschichte, Evolution des Lebens und der Erforschung verwandtschaftlicher Beziehungen unter den Organismen. Die Analyse der rRNA ist heute eine anerkannte Methode zur Einordnung einer Art in den universellen Stammbaum des Lebens und zur Ermittlung der nächstverwandten Arten. So ist die erwähnte Ähnlichkeit der RNA aus Mitochondrien und Plastiden eine starke Stütze der Endosymbiontenhypothese für diese Organellen.
Ribosomale RNA war wahrscheinlich bereits Bestandteil der ersten lebenden Einheiten auf der Erde und damit der Vorfahren aller heute lebenden Organismen (Endosymbiontentheorie). Sie gehört zur Grundausstattung jeder heute lebenden Zelle. Gleichzeitig hat sie in allen Organismen die gleiche Funktion und die Gene der rRNA unterliegen wahrscheinlich nur selten einem horizontalen Gentransfer. Man geht deshalb davon aus, dass die rRNA-Moleküle in allen Organismen mit vergleichbarer Geschwindigkeit evolvieren und nicht nur die Entwicklungsgeschichte des jeweiligen rRNA-Genes, sondern die eines gesamten Organismus widerspiegeln. Sie gelten als ideale „molekulare Chronometer“, mit deren Hilfe sich verwandtschaftliche Beziehungen unter den Organismen rekonstruieren lassen.
RNA ist ein instabileres Molekül als DNA und ihre Analyse daher technisch aufwendiger. Deshalb arbeitet man in der Praxis fast immer mit den Genen der rRNA, also der rDNA und leitet hiervon die Sequenz der rRNA ab.
Erkenntnisse

Die auf Basis der ribosomalen RNA entwickelten Stammbäume gelten inzwischen als zuverlässig und die meisten der hiermit errechneten Verwandtschaftsbeziehungen wurden auch mit anderen Methoden bestätigt. Dennoch kann die Anwendung der rRNA-Methode nicht allein für die korrekte Einordnung eines Organismus herangezogen werden. Die errechnete Position im Stammbaum muss stets mit anderen Methoden bestätigt werden. Hierzu zählen nach wie vor auch morphologische und physiologische Merkmale. So ist es beispielsweise nicht möglich, allein auf Basis einer rRNA-Analyse eine neue Art zu definieren.
Große Bedeutung hat die rRNA-basierte Phylogenetik bei Mikroorganismen, denn Einzeller sind anhand morphologischer und physiologischer Merkmale allein schwer einzuordnen. Hier bietet die Analyse der ribosomalen RNA eine schnelle und zuverlässige Ergänzung. Anhand empirischer Daten geht man heute davon aus, dass Bakterien, deren 16S-rRNA-Sequenzen zu 97–98 % übereinstimmen, einer Art zugerechnet werden können.
Man hat aus verschiedenen Umweltproben (zum Beispiel Wasser, Boden oder Klärschlamm) DNA isoliert und hieraus rRNA-Sequenzen bestimmt. In einem Gramm Waldboden fand man so zum Beispiel rRNA-Gene von etwa 13.000 (!) verschiedenen „Arten“. Vergleicht man diese Sequenzen mit denen von kultivierbaren und daher bekannten Mikroorganismen, kann man abschätzen, dass wir heute nur 1–5 % aller Mikroorganismen überhaupt kennen. Von der Existenz der übergroßen Mehrheit aller Bakterien und Archaeen weiß man nur durch ihre rRNA-Sequenzen, ohne eine Vorstellung davon zu haben, wovon sie leben und welche Rolle sie in der Natur spielen.
Alle bisher gültig beschriebenen (das heißt kultivierbaren) Bakterien werden – je nach Autor – derzeit in 26 Phyla oder Stämme eingeordnet. Jedoch verteilt sich die übergroße Mehrheit aller Bakterien auf nur wenige Stämme, zum Beispiel auf die Proteobacteria, Firmicutes und Actinobacteria. Die meisten Phyla werden hingegen nur durch einen oder wenige kultivierbare Vertreter repräsentiert (zum Beispiel Acidobacteria), obwohl man weiß, dass diese Gruppen viel mehr Vertreter umfassen müssen.
26 weitere Phyla werden nur mit Hilfe von aus Umweltproben isolierten rRNA-Sequenzen postuliert, ohne bisher einen Vertreter kultiviert und charakterisiert zu haben.
Die wichtigste Konsequenz aus der Anwendung der rRNA-basierten Phylogenetik war bisher aber die Einteilung aller Organismen in die drei Domänen der Bakterien, Archaeen und Eukaryoten. Aber auch die derzeit aktuelle Einteilung der Urmünder (Protostomia), der artenreichsten Tiergruppe, in Häutungstiere (Ecdysozoa, u. a. Insekten, Fadenwürmer) und Lophotrochozoen (Lophotrochozoa, u. a. Weichtiere, Ringelwürmer) ist vor allem anhand von Untersuchungen der 18-rRNA der Ribosomen entwickelt worden.
Weblinks
Anmerkungen
- Spricht man von eukaryotischer rRNA, so ist stets die der Mitochondrien und Plastiden ausgenommen. Diese besitzen nämlich eigene Ribosomen (wie die mitochondrialen Ribosomen), die aber eher prokaryotischem Muster entsprechen. Dabei gibt es allerdings vielerlei Variationen. So enthalten z. B. die mitochondrialen Ribosomen vieler Spezies, so auch die des Menschen, nur zwei rRNA-Moleküle.
Einzelnachweise
- Eintrag RF00177 in der Rfam-Datenbank, abgerufen am 31. Mai 2017.
- S. J. Goodfellow, J. C. Zomerdijk: Basic mechanisms in RNA polymerase I transcription of the ribosomal RNA genes. In: Sub-cellular biochemistry. Band 61, 2013, S. 211–236, doi:10.1007/978-94-007-4525-4_10, PMID 23150253, PMC 3855190 (freier Volltext).
- M. Penzo, A. Galbiati, D. Treré, L. Montanaro: The importance of being (slightly) modified: The role of rRNA editing on gene expression control and its connections with cancer. In: Biochimica et Biophysica Acta. Band 1866, Nummer 2, Dezember 2016, S. 330–338, doi:10.1016/j.bbcan.2016.10.007, PMID 27815156.
- Mary Campbell: Biochemistry. Cengage Learning, 2007, ISBN 978-0-495-39041-1, S. 254.
- W. S. Yip, N. G. Vincent, S. J. Baserga: Ribonucleoproteins in archaeal pre-rRNA processing and modification. In: Archaea. Band 2013, 2013, S. 614735, doi:10.1155/2013/614735, PMID 23554567, PMC 3608112 (freier Volltext).
- Klaus Urich: Comparative Animal Biochemistry. Springer Science & Business Media, 1994, ISBN 978-3-540-57420-0, S. 45–46.
- P. I. Penev, S. Fakhretaha-Aval, V. J. Patel, J. J. Cannone, R. R. Gutell, A. S. Petrov, L. D. Williams, J. B. Glass: Supersized ribosomal RNA expansion segments in Asgard archaea. In: Genome Biology and Evolution. 12, Nr. 10, August 2020, S. 1694–1710. doi:10.1093/gbe/evaa170. PMID 32785681. PMC 7594248 (freier Volltext).
- Petar I. Penev, Sara Fakhretaha-Aval, Vaishnavi J. Patel, Jamie J. Cannone, Robin R. Gutell, Anton S. Petrov, Loren Dean Williams, Jennifer B. Glass: Supersized Ribosomal RNA Expansion Segments in Asgard Archaea. In: Genome Biology and Evolution. 12, Nr. 10, 1. Oktober 2020, S. 1694–1710. doi:10.1093/gbe/evaa170.
- K. R. Luehrsen, D. E. Nicholson, D. C. Eubanks, G. E. Fox: An archaebacterial 5S-rRNA contains a long insertion sequence. In: Nature. 293, Nr. 5835, Mai 1981, S. 755–756. bibcode:1981Natur.293..755L. doi:10.1038/293755a0. PMID 6169998.
- M. R. Tirumalai, J. T. Kaelber, D. R. Park, Q. Tran, G. E. Fox: Cryo‐electron microscopy visualization of a large insertion in the 5S ribosomal RNA of the extremely halophilic archaeon Halococcus morrhuae. In: FEBS Open Bio. 10, Nr. 10, 31. August 2020, S. 1938–1946. doi:10.1002/2211-5463.12962. PMID 32865340. PMC 7530397 (freier Volltext).
- Victor G. Stepanov, George E. Fox: Expansion segments in bacterial and archaeal 5S ribosomal RNAs, in: RNA Band 27, Nr. 2, Februar 2021, S. 133–150, doi:10.1261/rna.077123.120, PMID 33184227, PMC 7812874 (freier Volltext), Epub 12. November 2020
- A. W. Coleman: Nuclear rRNA transcript processing versus internal transcribed spacer secondary structure. In: Trends in genetics : TIG. Band 31, Nummer 3, März 2015, S. 157–163, doi:10.1016/j.tig.2015.01.002, PMID 25648500.
- K. Fukuda, M. Ogawa, H. Taniguchi, M. Saito: Molecular Approaches to Studying Microbial Communities: Targeting the 16S Ribosomal RNA Gene. In: Journal of UOEH. Band 38, Nummer 3, September 2016, S. 223–232, doi:10.7888/juoeh.38.223, PMID 27627970.
- A. L. Torres-Machorro, R. Hernández, A. M. Cevallos, I. López-Villaseñor: Ribosomal RNA genes in eukaryotic microorganisms: witnesses of phylogeny? In: FEMS microbiology reviews. Band 34, Nummer 1, Januar 2010, S. 59–86, doi:10.1111/j.1574-6976.2009.00196.x, PMID 19930463.
- C. U. Chukwudi: rRNA Binding Sites and the Molecular Mechanism of Action of the Tetracyclines. In: Antimicrobial agents and chemotherapy. Band 60, Nummer 8, August 2016, S. 4433–4441, doi:10.1128/AAC.00594-16, PMID 27246781, PMC 4958212 (freier Volltext).
- J. Trylska, M. Kulik: Interactions of aminoglycoside antibiotics with rRNA. In: Biochemical Society transactions. Band 44, Nummer 4, August 2016, S. 987–993, doi:10.1042/BST20160087, PMID 27528743.