Folding@home
Folding@home (oft auch kurz F@H oder FAH) ist ein Volunteer-Computing-Projekt für die Krankheitsforschung, das die Proteinfaltung und andere Arten von Molekulardynamik simuliert. Statt die Rechenleistung eines einzelnen Rechners zu nutzen, wird dabei eine komplexe Aufgabe in Teilaufgaben aufgeteilt, diese auf mehrere Rechner verteilt und deren Rechenleistungen zur Aufgabenbewältigung genutzt. Das Projekt nutzt durch verteiltes Rechnen die ungenutzten Verarbeitungsressourcen von Personalcomputern und Servern, auf denen die Software installiert ist und die so zur Erforschung von Krankheiten beitragen.
| Folding@home | |
|---|---|
 | |
| Bereich: | Medizin und Biologie |
| Ziel: | Simulation der Proteinfaltung |
| Betreiber: | Stanford University |
| Land: | |
| Plattform: | Windows, Linux, macOS, FreeBSD ehemals: BOINC, Android |
| Website: | foldingathome.org |
| Projektstatus | |
| Status: | aktiv |
| Beginn: | 19. September 2000 |
| Ende: | aktiv |
Das Projekt verwendet hierbei eine statistische Simulationsmethode, die gegenüber traditionellen Berechnungsmethoden einen Paradigmenwechsel darstellt. Als Teil des Client-Server-Modells erhalten die Teilnehmer (Clienten) nach Anforderung eines Dienstes beim Server jeweils Teile einer Simulation (Arbeitseinheiten/Work-Units), berechnen und vervollständigen sie und geben sie an die Datenbankserver des Projekts zurück, wo die Einheiten sodann zu einer Gesamtsimulation zusammengestellt werden.[1]
Der Hauptzweck des Projekts ist die Bestimmung der Mechanismen der Proteinfaltung, d. h. des Prozesses, durch den Proteine ihre endgültige dreidimensionale Struktur erreichen, und die Untersuchung der Ursachen von Proteinfehlfaltungen. Dies ist von Interesse für die medizinische Forschung über Alzheimer, Huntington und viele Formen von Krebs, neben weiteren, anderen Krankheiten. In geringerem Umfang versucht Folding@home auch, die endgültige Struktur eines Proteins vorherzusagen und zu bestimmen, wie andere Moleküle mit ihm interagieren können, was sich auf die Entwicklung von Medikamenten auswirkt.[2]
Folding@Home erreichte am 13. April 2020 während der COVID-19-Pandemie eine kombinierte Rechenleistung, die schneller als die 500 schnellsten Supercomputer der Welt zusammengenommen war, und übertraf damit den zu diesem Zeitpunkt schnellsten Supercomputer um das 15-Fache.[3]
Am 17. April 2020 wurde eine neue Client-Software veröffentlicht, die die Liste der priorisierbaren Projekte um COVID-19-Projekte ergänzt.[4]
Geschichte
Die ersten Ideen zur Nutzung zahlreicher Computer entstanden laut Vijay Pande im Sommer 2000, damals wurde dann auch die erste Version der Client-Software programmiert – seinerzeit noch ein Bildschirmschoner.[5] Am 19. September 2000 wurde die erste Software für Folding@home seitens des Pande Laboratory der Stanford University offiziell veröffentlicht[6] und wurde seitdem gemeinnützig unter der Leitung von Vijay Pande entwickelt. Es steht seit 2019 unter der Leitung von Gregory Bowman, Professor für Biochemie und molekulare Biophysik an der Washington University School of Medicine in St. Louis[7] – es wird von verschiedenen wissenschaftlichen Einrichtungen und Forschungslabors weltweit kollektiv genutzt. Die von Folding@home generierten Ergebnisse werden nicht verkauft.[8] Die generierten Datensätze können Forscher weltweit auf Anfrage abrufen[9] und direkt von einer Website aus beziehen.[10]
Folding@home-Forscher Gregory Bowman wurde 2010 mit dem Thomas Kuhn Paradigm Shift Award der American Chemical Society für die Entwicklung der Open-Source-Software MSMBuilder[11] und für das Erreichen einer quantitativen Übereinstimmung zwischen Theorie und Experiment ausgezeichnet.[12] Für seine Arbeit wurde Vijay Pande 2012 wiederum mit dem Michael und Kate Bárány-Preis für junge Forscher für die „Entwicklung von Berechnungsmethoden zur Erstellung führender theoretischer Modelle für die Protein- und RNA-Faltung“[13] und 2006 mit dem Irving Sigal Young Investigator Award[14] für seine Simulationsergebnisse ausgezeichnet.
Hintergrund
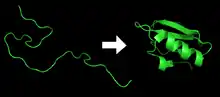
Proteine sind ein wesentlicher Bestandteil vieler biologischer Funktionen und sind an allen Prozessen, die in Zellen ablaufen, beteiligt. Oftmals sind solche Proteine Enzyme, die biochemische Reaktionen ausführen, einschließlich Signaltransduktion, molekularer Transport und Zellregulation. Einige Proteine fungieren als Strukturproteine, die als eine Art Gerüst für Zellen dienen, während andere Proteine wie Antikörper am Immunsystem beteiligt sind. Damit ein Protein diese Funktionen ausführen kann, muss es sich in eine funktionelle dreidimensionale Struktur falten, ein Prozess, der häufig spontan abläuft und von Wechselwirkungen innerhalb seiner Aminosäuresequenz und Wechselwirkungen der Aminosäuren mit seiner Umgebung abhängt.
Die Proteinfaltung wird hauptsächlich durch hydrophobe Wechselwirkungen, die Bildung intramolekularer Wasserstoffbrückenbindungen und Van-der-Waals-Kräfte bestimmt, die der Konformationsentropie entgegenwirken.[15][16] Der Faltungsprozess beginnt häufig co-translational, sodass sich der N-Terminus des Proteins zu falten beginnt, während der C-terminale Teil des Proteins noch vom Ribosom synthetisiert wird. Ein Proteinmolekül kann sich jedoch während oder nach der Biosynthese spontan falten.[17] Der Faltungsprozess hängt auch vom Lösungsmittel (Wasser oder Doppellipidschicht)[18] der Salzkonzentration, dem pH-Wert, der Temperatur, dem möglichen Vorhandensein von Cofaktoren und molekularen Chaperonen ab. Proteine haben Einschränkungen hinsichtlich ihrer Faltungsmöglichkeiten aufgrund von sterischen Hinderungen zwischen einzelnen Atomen, sodass nur bestimmte Kombinationen von Diederwinkeln erlaubt sind. Diese zulässigen Winkel der Proteinfaltung werden in einem zweidimensionalen Diagramm beschrieben, das als Ramachandran-Diagramm bekannt ist und mit φ (Phi)- und ψ (Psi)-Winkeln beschrieben werden.[19] Für die Bioinformatik ist das Verständnis der Proteinfaltung wichtig, um bestimmte Funktionen und Mechanismen des Proteins vorherzusagen.[20][21] Obwohl die Faltung in einer zellulären Umgebung stattfindet, die eine hohe Konzentration an Proteinen aufweist (im Englischen als macromolecular crowding bekannt), verläuft sie normalerweise reibungslos. Aufgrund der chemischen Eigenschaften eines Proteins oder anderer Faktoren kann es zu Fehlfaltungen des Proteins kommen. Auch wenn zelluläre Mechanismen dazu beitragen, dass fehlgefaltete Proteine entfernt oder neu gefaltet werden, kann es zur Aggregation der fehlgefalteten Proteine kommen und eine Vielzahl von Krankheiten verursacht werden.[22] Laborexperimente zur Untersuchung von Proteinfaltungsprozessen sind hinsichtlich ihres Anwendungsbereichs und ihrer Genauigkeit eingeschränkt, weshalb physikalische Rechenmodelle hinzugezogen werden, die ein tieferes Verständnis zur Proteinfaltung, -fehlfaltung und -aggregation liefern.[23][24]
Aufgrund der Komplexität der Proteinkonformation oder des Konfigurationsraums des Proteins (die Menge aller möglichen Faltungszustände, die ein Protein annehmen kann) und der begrenzten Rechenleistung sind Molekulardynamik-Simulationen hinsichtlich der Untersuchung von Proteinfaltungen stark eingeschränkt. Während sich die meisten Proteine typischerweise in der Größenordnung von Millisekunden falten,[23][25] konnten Simulationen vor 2010 nur Zeitskalen von Nanosekunden bis Mikrosekunden erreichen.[26] Supercomputer wurden verwendet, um die Proteinfaltung zu simulieren, aber solche Systeme sind kostspielig und werden größtenteils von vielen Forschungsgruppen gemeinsam genutzt. Da die Berechnungen in kinetischen Modellen zeitlich nacheinander erfolgen, ist zudem eine Skalierung traditioneller Molekulardynamik-Simulationen auf solche Systeme außerordentlich schwierig.[27][28] Da die Proteinfaltung ein stochastischer Prozess ist und im Laufe der Zeit statistisch variieren kann, ist es außerdem rechnerisch schwierig, lange Simulationen für umfassende Ansichten des Faltungsprozesses zu verwenden.[29][30]
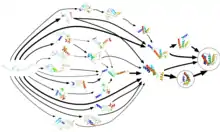
Die Proteinfaltung erfolgt nicht in einem Schritt.[22] Stattdessen verbringen Proteine den größten Teil der gesamten Faltungszeit, in einigen Fällen mit bis zu 96 %[31], in verschiedenen Konformationszwischenzuständen, die jeweils ein lokales thermodynamisches Minimum an freier Energie in der Energielandschaft des Proteins darstellen (siehe Faltungstrichter). Durch einen Prozess namens Adaptive Sampling werden diese Konformationen von Folding@home als Ausgangspunkte für eine Reihe von Simulationsverläufen für Faltungsprozesse verwendet. Mit der Zeit werden neue Konformationen entdeckt, die als neue Ausgangspunkte für Simulationsverläufe dienen (zyklischer Prozess). Sobald man den zugrundeliegenden verborgenen Zuständen eine Übertragungswahrscheinlichkeit und den von außen beobachtbaren Ausgabesymbolen (sog. Emissionen), die aus den verborgenen Zuständen resultieren, eine Emissionswahrscheinlichkeit zuordnen kann, spricht man vom Hidden Markov Model (HMM). HMM sind zeitdiskrete Mastergleichungsmodelle, welche die Konformations- und Energielandschaft eines Biomoleküls als eine Menge an unterschiedlichen Strukturen und kurzen Übergangszuständen zwischen den Strukturen beschreiben. Das Hidden Markov Model, kombiniert mit Adaptive Sampling, erhöht die Effizienz der Simulation erheblich. Da dadurch die Berechnung innerhalb des lokalen Energieminimums vermieden wird, ist diese Kombination für verteilte Systeme (einschließlich GPUGRID) geeignet, da sie die statistische Anhäufung kurzer, unabhängiger Simulationsverläufe für Faltungsprozesse ermöglicht.[32] Die Zeit, die zum Erstellen eines Hidden Markov Model benötigt wird, ist umgekehrt proportional zur Anzahl der parallelen Simulationen, d. h. zur Anzahl der verfügbaren Prozessoren. Mit anderen Worten: Es wird eine parallele Verarbeitung geschaffen (siehe Parallelrechner), was zu einer Verringerung der gesamten Berechnungszeit um ungefähr vier Größenordnungen führt. Ein abgeschlossenes Hidden Markov Model kann bis zu zehntausend Zustände aus dem Phasenraum des Proteins (alle Konformationen, die ein Protein annehmen kann) und die Übergänge zwischen ihnen enthalten. Das Modell veranschaulicht Faltungsprozesse und -pfade und Forscher können später kinetische Cluster verwenden, um eine sogenannte coarse-grained-Darstellung (zu deutsch „grobkörnig“) des detaillierten Modells zu erstellen. Diese Hidden Markov Modelle können verwendet werden, um Fehlfaltungsprozesse zu bestimmen sowie Simulationen quantitativ mit Experimenten zu vergleichen.[33][29][34]
Zwischen 2000 und 2010 hat sich die Länge einer Aminosäuresequenz der von Folding@home untersuchten Proteine um den Faktor vier erhöht, während sich die Zeitskalen für Proteinfaltungssimulationen um sechs Größenordnungen erhöht haben.[35] Im Jahr 2002 verwendete Folding@home Markov-Zustandsmodelle[36] um eine Prozessorzeit von ungefähr eine Million Tagen mit Simulationen über einen Zeitraum von mehreren Monaten durchzuführen,[37] und im Jahr 2011 erfolgte eine parallele Verarbeitung einer weiteren Simulation, die insgesamt eine Prozessorzeit von 10 Millionen Rechenstunden erforderte.[38] Im Januar 2010 simulierte Folding@home mithilfe von HMM die Dynamik des langsam faltenden NTL9-Proteins mit 32 Aminosäureresten mit einer Simulationszeit von 1,52 Millisekunden, eine Zeitskala, die mit experimentellen Vorhersagen der Faltungsrate übereinstimmt, aber tausendmal länger als die früher erreichte Zeit ist.[39] Das Modell bestand aus vielen einzelnen Trajektorien, die jeweils um zwei Größenordnungen kürzer waren, und lieferte eine genaue Darstellung der Energielandschaft des Proteins.[33][26][40]
Anwendungen in der Biomedizin
Viruserkrankungen
Einige Folding@home-Projekte haben als Forschungsziel, Viren – zum Beispiel das Influenzavirus oder HIV – daran zu hindern, biologische Zellen zu erkennen und in diese einzudringen.[41] 2011 begann Folding@home mit Simulationen der Dynamik des Enzyms RNase H, einer Schlüsselkomponente von HIV, um zu versuchen, Medikamente zu entwickeln, die dieses Enzym deaktivieren.[42] Folding@home wurde auch zur Untersuchung der Membranfusion, eines wesentlichen Ereignisses für die Virusinfektion und eine Vielzahl biologischer Funktionen, eingesetzt. Diese Fusion beinhaltet Konformationsänderungen der viralen Fusionsproteine und das Andocken der Proteine, aber die genauen molekularen Mechanismen hinter der Fusion sind weitgehend unbekannt.[43] Fusionsereignisse können aus über einer halben Million Atomen bestehen, die für Hunderte von Mikrosekunden interagieren.[44] Die Entwicklung von Modellen zur Vorhersage der Mechanismen der Membranfusion trägt zum wissenschaftlichen Verständnis des Prozesses mit antiviralen Medikamenten bei. Im Jahr 2006 haben die Wissenschaftler Markov-Zustandsmodelle und das Folding@home-Netzwerk angewandt, um zwei Wege für die Fusion zu entdecken und weitere Erkenntnisse zu gewinnen.
Nach detaillierten Simulationen von Folding@home von kleinen Zellen, die als Vesikel bekannt sind, führte das Pande-Labor 2007 eine neue Berechnungsmethode ein, um die Topologie der strukturellen Veränderungen während der Fusion zu messen.[45] 2009 verwendeten die Forscher Folding@home zur Untersuchung von Mutationen des Influenza-Hämagglutinins, eines Proteins, das ein Virus an seine Wirtszelle bindet und den Eintritt des Virus unterstützt. Mutationen des Hämagglutinins beeinflussen, wie gut das Protein an die Rezeptormoleküle der Zelloberfläche eines Wirts bindet, was bestimmt, wie infektiös der Virusstamm für den Wirtsorganismus ist. Die Kenntnis der Auswirkungen von Hämagglutinin-Mutationen hilft bei der Entwicklung antiviraler Medikamente.[46] Seit 2012 simuliert Folding@home weiterhin die Faltung und die Wechselwirkungen von Hämagglutinin und ergänzt damit experimentelle Studien an der Universität von Virginia.[47]
SARS-CoV-2-Virus

Im März 2020 startete Folding@home ein Programm zur Unterstützung von Forschern auf der ganzen Welt, die daran arbeiten, ein Heilmittel zu finden und mehr über den Ausbruch von COVID-19 – auch bekannt als die Atemwegserkrankung, die durch das neuartige Coronavirus ausgelöst wird – zu erfahren.[49][50] Die erste Welle von Projekten simuliert potenziell medikamentös behandelbare Protein-Targets des SARS-CoV-2-Virus und des verwandten SARS-CoV-Virus, von denen es wesentlich mehr Daten gibt.[51][52][53][54][55]
Alzheimer-Krankheit
Die Alzheimer-Krankheit ist eine unheilbare neurodegenerative Erkrankung, die vor allem ältere Menschen betrifft und für mehr als die Hälfte aller Demenzfälle verantwortlich ist. Die genaue Ursache bleibt unbekannt, aber die Krankheit wird als eine Protein-Fehlfaltungskrankheit identifiziert. Alzheimer ist mit toxischen Aggregationen des Peptids Beta-Amyloid (Aβ) assoziiert, die durch Fehlfaltung und Verklumpung von Aβ zusammen mit anderen Aβ-Peptiden verursacht werden. Diese Aβ-Aggregate wachsen dann zu signifikant größeren senilen Plaques, einem pathologischen Marker der Alzheimer-Krankheit.[56][57][58] Aufgrund der heterogenen Natur dieser Aggregate hatten experimentelle Methoden wie die Röntgenkristallographie und die kernmagnetische Resonanz (NMR) Schwierigkeiten, ihre Strukturen zu charakterisieren. Darüber hinaus sind die atomaren Simulationen der Aβ-Aggregation aufgrund ihrer Größe und Komplexität rechnerisch sehr anspruchsvoll.[59]
Die Verhinderung der Aggregation von Aβ ist eine vielversprechende Methode zur Entwicklung von Therapeutika für die Alzheimer-Krankheit, so die Doktoren Naeem und Fazili in einem Übersichtsartikel.[60] 2008 simulierte Folding@home die Dynamik der Aggregation von Aβ in atomaren Details über Zeitskalen in der Größenordnung von zehn Sekunden. Frühere Studien konnten nur etwa 10 Mikrosekunden simulieren. Folding@home konnte die Faltung von Aβ um sechs Größenordnungen länger als bisher möglich simulieren. Die Forscher nutzten die Ergebnisse dieser Studie, um eine Beta-Haarnadel (beta-hairpin) zu identifizieren, die eine Hauptquelle für molekulare Interaktionen innerhalb der Struktur war.[61] Die Studie half, das Pande Laboratory für zukünftige Aggregationsstudien und für weitere Forschungen vorzubereiten, um ein kleines Peptid zu finden, das den Aggregationsprozess stabilisieren könnte.[59]
Im Dezember 2008 fand Folding@home mehrere kleine Arzneimittelkandidaten, die die Toxizität der Aβ-Aggregate zu hemmen scheinen.[62] Im Jahr 2010 wurde in enger Zusammenarbeit mit dem Center for Protein Folding Machinery damit begonnen, diese Arzneimittelkandidaten an biologischem Gewebe zu testen. 2011 schloss Folding@home die Simulationen mehrerer Mutationen von Aβ ab, die die Aggregatbildung zu stabilisieren scheinen, was die Entwicklung therapeutischer Arzneimitteltherapien für die Krankheit unterstützen und bei experimentellen Kernspinresonanzspektroskopie-Studien an Aβ-Oligomeren sehr hilfreich sein könnte.[63] Später im selben Jahr begann Folding@home mit Simulationen verschiedener Aβ-Fragmente, um zu bestimmen, wie verschiedene natürliche Enzyme die Struktur und Faltung von Aβ beeinflussen.[64]
Chorea Huntington
Die Huntington-Krankheit ist eine neurodegenerative genetische Erkrankung, die mit einer Fehlfaltung und Aggregation von Proteinen einhergeht. Übermäßige Wiederholungen der Glutaminsäure am N-Terminus des Huntingtin-Proteins verursachen eine Aggregation, und obwohl das Verhalten der Wiederholungen nicht vollständig verstanden wird, führt es doch zu dem mit der Krankheit verbundenen kognitiven Rückgang.[65] Wie bei anderen Aggregaten gibt es Schwierigkeiten bei der experimentellen Bestimmung ihrer Struktur.[66] Wissenschaftler verwenden Folding@home, um die Struktur des Huntingtin-Proteinaggregats zu untersuchen und vorherzusagen, wie es sich bildet, und unterstützen dabei rationales Wirkstoffdesign, um die Aggregatbildung zu stoppen. Das N17-Fragment des Huntington-Proteins beschleunigt diese Aggregation, und obwohl mehrere Mechanismen vorgeschlagen wurden, ist seine genaue Rolle in diesem Prozess noch weitgehend unbekannt.[67] Folding@home hat dieses und andere Fragmente simuliert, um ihre Rolle bei der Krankheit zu klären. Seit 2008 werden seine Methoden zum Medikamentenentwurf für die Alzheimer-Krankheit auf Huntington angewendet.[64]
Krebs
Bei mehr als der Hälfte aller bekannten Krebsarten handelt es sich um Mutationen von p53, einem in jeder Zelle vorhandenen Tumorsuppressorprotein, das den Zellzyklus reguliert und bei einer Schädigung der DNA das Signal zum Zelltod gibt. Spezifische Mutationen in p53 können diese Funktionen stören, so dass eine abnorme Zelle unkontrolliert weiter wachsen kann, was zur Entstehung von Tumoren führt. Die Analyse dieser Mutationen trägt dazu bei, die Grundursachen von p53-verwandten Krebsarten zu erklären.[68] Im Jahr 2004 wurde mit Folding@home die erste molekulardynamische Studie zur Rückfaltung des p53-Proteindimers in einer rein atomaren Simulation von Wasser durchgeführt. Die Ergebnisse der Simulation stimmten mit experimentellen Beobachtungen überein und gaben Einblicke in die Rückfaltung des Dimers, die zuvor nicht möglich waren.[69] Dies war die erste von Fachleuten überprüfte Publikation über Krebs aus einem verteilten Computerprojekt. Im folgenden Jahr wurde mit Folding@home eine neue Methode zur Identifizierung der Aminosäuren, die für die Stabilität eines bestimmten Proteins entscheidend sind, angewandt, die dann zur Untersuchung von Mutationen von p53 verwendet wurde. Die Methode war bei der Identifizierung krebsfördernder Mutationen einigermaßen erfolgreich und bestimmte die Auswirkungen spezifischer Mutationen, die sonst nicht experimentell gemessen werden konnten.[70]
Folding@home wird auch zur Untersuchung von Chaperonen verwendet, Hitzeschockproteinen, die eine wesentliche Rolle für das Überleben der Zelle spielen, indem sie die Faltung anderer Proteine in der überfüllen und chemisch belastenden Umgebung innerhalb einer Zelle unterstützen. Rasch wachsende Krebszellen sind auf spezifische Chaperone angewiesen, und einige Chaperone spielen eine Schlüsselrolle bei der Chemotherapieresistenz. Die Hemmung dieser spezifischen Chaperone wird als potentielle Wirkungsweise für effiziente Chemotherapeutika oder zur Verringerung der Krebsausbreitung angesehen.[71] Mit Folding@home und in enger Zusammenarbeit mit dem Center for Protein Folding Machinery hofft das Pande Laboratory ein Medikament zu finden, das die in Krebszellen beteiligten Chaperone hemmt. Die Forscher verwenden Folding@home auch zur Untersuchung anderer Moleküle, die mit Krebs in Verbindung stehen, wie das Enzym Src-Kinase und einige Formen der gravierten Homöodomäne: ein großes Protein, das an vielen Krankheiten, darunter auch Krebs, beteiligt sein könnte. 2011 begann Folding@home mit Simulationen der Dynamik des kleinen Proteins EETI, das durch Bindung an Oberflächenrezeptoren von Krebszellen Karzinome in bildgebenden Verfahren identifizieren kann.
Interleukin 2 (IL-2) ist ein Protein, das den T-Zellen des Immunsystems hilft, Krankheitserreger und Tumore anzugreifen. Seine Verwendung als Krebsbehandlung ist jedoch wegen schwerer Nebenwirkungen, wie zum Beispiel einem Lungenödem, eingeschränkt. IL-2 bindet an diese Lungenzellen anders als an T-Zellen, so dass die IL-2-Forschung die Unterschiede zwischen diesen Bindungsmechanismen verstehen muss. Im Jahr 2012 unterstützte Folding@home die Entdeckung einer mutierten Form von IL-2, die dreihundertmal wirksamer in ihrer Rolle als Immunsystem ist, aber weniger Nebenwirkungen hat. In Experimenten hat diese veränderte Form das natürliche IL-2 bei der Behinderung des Tumorwachstums deutlich übertroffen.[72] Pharmazeutische Unternehmen haben Interesse an dem mutierten Molekül bekundet, und die National Institutes of Health testen es gegen eine Vielzahl von Tumormodellen, um seine Entwicklung als Therapeutikum zu beschleunigen.[73]
Osteogenesis imperfecta
Osteogenesis imperfecta, bekannt als die sogenannte Glasknochenkrankheit, ist eine unheilbare genetische Knochenerkrankung, die tödlich sein kann. Die Erkrankten sind nicht in der Lage, funktionsfähiges Bindegewebe zu bilden. Dies ist meist auf eine Mutation im Typ-I-Kollagen zurückzuführen, das eine Vielzahl struktureller Aufgaben erfüllt und das am häufigsten vorkommende Protein bei Säugetieren ist. Die Mutation verursacht eine Verformung der Dreifachhelixstruktur des Kollagens, die, wenn sie nicht auf natürliche Weise zerstört wird, zu abnormalem und geschwächtem Knochengewebe führt. 2005 testete Folding@home eine neue quantenmechanische Methode, die frühere Simulationsmethoden verbesserte und die für künftige Computerstudien über Kollagen nützlich sein könnte.[74] Obwohl Forscher Folding@home zur Untersuchung der Kollagenfaltung und -fehlfaltung verwendet haben, ist das Interesse an diesem Projekt im Vergleich zur Alzheimer- und Huntington-Forschung als Pilotprojekt zu sehen.[64]
Wirkstoffdesign
Medikamente funktionieren, indem sie an bestimmte Stellen auf den Zielmolekülen binden und eine gewünschte Veränderung hervorrufen, wie z. B. die Deaktivierung eines Ziels oder die Herbeiführung einer Konformationsänderung. Im Idealfall sollte ein Medikament sehr spezifisch wirken und nur an sein Zielmolekül binden, ohne andere biologische Funktionen zu beeinträchtigen. Es ist jedoch schwierig, genau zu bestimmen, wo und wie fest zwei Moleküle binden werden. Aufgrund der begrenzten Rechenleistung müssen die heutigen Methoden in silico in der Regel Geschwindigkeit gegen Genauigkeit eintauschen – z. B. müssen schnelle Protein-Docking-Methoden statt rechenintensiver freier Energieberechnungen verwendet werden. Die Rechenleistung von Folding@home ermöglicht es den Forschern, beide Methoden zu verwenden und ihre Effizienz und Zuverlässigkeit zu bewerten.[75][76] Computergestütztes Wirkstoffdesign hat das Potenzial, die Medikamentenentwicklung zu beschleunigen und die Kosten zu senken.[77] 2010 verwendete Folding@home MSMs und Berechnungen der freien Energie, um den nativen Zustand des Villin-Proteins mit einer Abweichung von bis zu 1,8 Angström (Å) via RMSD (root mean square deviation) von der experimentell durch Röntgenkristallographie bestimmten Kristallstruktur vorherzusagen. Diese Genauigkeit hat Auswirkungen auf künftige Methoden zur Vorhersage von Proteinstrukturen, auch für inhärent unstrukturierte Proteine. Wissenschaftler haben Folding@home zur Erforschung von Medikamentenresistenzen verwendet, indem sie Vancomycin, ein Antibiotikum letzter Instanz, und Beta-Laktamase, ein Protein, das Antibiotika wie Penicillin abbauen kann, untersucht haben.[78][79]
Die chemische Aktivität findet entlang der aktiven Stelle eines Proteins statt. Traditionelle Methoden des Arzneimitteldesigns beinhalten eine enge Bindung an diese Stelle und die Blockierung ihrer Aktivität, unter der Annahme, dass das Zielprotein in einer starren Struktur existiert. Dieser Ansatz funktioniert jedoch nur bei etwa 15 % aller Proteine. Proteine enthalten allosterische Stellen, die, wenn sie durch kleine Moleküle gebunden sind, die Konformation eines Proteins verändern und letztlich die Aktivität des Proteins beeinflussen können. Diese Stellen sind attraktive Zielorte für Medikamente, aber ihre Lokalisierung ist sehr rechenaufwändig. Im Jahr 2012 wurden Folding@home und MSMs verwendet, um allosterische Stellen in drei medizinisch relevanten Proteinen zu identifizieren: Beta-Laktamase, Interleukin-2 und RNase H.[79][80]
Ungefähr die Hälfte aller bekannten Antibiotika greifen in die Funktionsweise des Ribosoms eines Bakteriums ein, einer großen und komplexen biochemischen Maschine, die die Proteinbiosynthese durch die Übersetzung von Boten-RNA in Proteine durchführt. Makrolid-Antibiotika verstopfen den Ausgangstunnel des Ribosoms und verhindern so die Synthese essentieller bakterieller Proteine. Im Jahr 2007 erhielt das Pande Laboratory ein Stipendium zur Untersuchung und Entwicklung neuer Antibiotika.[64] Im Jahr 2008 untersuchten sie mit Folding@home das Innere dieses Tunnels und wie bestimmte Moleküle ihn beeinflussen können.[81] Die vollständige Struktur des Ribosoms wurde erst ab 2011 bestimmt, und Folding@home hat auch ribosomale Proteine simuliert, da viele ihrer Funktionen weitgehend unbekannt sind.[82]
Rechenleistung
| Datum | Rechenleistung |
|---|---|
| 16.09.2007 | 1 PetaFLOPS |
| 07.05.2008 | 2 PetaFLOPS |
| 20.08.2008 | 3 PetaFLOPS |
| 28.09.2008 | 4 PetaFLOPS |
| 19.07.2016 | 100 PetaFLOPS |
| 20.03.2020 | 470 PetaFLOPS |
| 13.04.2020 | 2,4 ExaFLOPS |
| 16.04.2020 | 2,6 ExaFLOPS |
| 21.05.2020 | 2,2 ExaFLOPS |
| 03.07.2020 | 1,5 ExaFLOPS |
| 25.07.2020 | 1,3 ExaFLOPS |
| 02.08.2020 | 1,2 ExaFLOPS |
| 02.09.2020 | 2,5 ExaFLOPS |
| 10.09.2020 | 3,5 ExaFLOPS |
| 14.09.2020 | 4,0 ExaFLOPS |
| 24.09.2020 | 4,3 ExaFLOPS |
| 07.10.2020 | 307 PetaFLOPS |
| 24.02.2021 | 195 PetaFLOPS |
| 15.05.2021 | 369 PetaFLOPS |
2007 bis 2016
Zwischen Juni 2007 und Juni 2011 übertraf die Rechenleistung aller am Folding@home Projekt beteiligten Computer die Leistung des schnellsten Supercomputers der TOP500. Er wurde jedoch im November 2011 vom K computer und im Juni 2012 vom Computer Blue Gene/Q in den Schatten gestellt. Am 16. September 2007 erreichte das Folding@home-Projekt, vor allem dank der Beteiligung von PlayStation-3-Konsolen, offiziell ein Leistungsniveau, das höher als ein natives[83] PetaFLOPS war, und wurde damit zum ersten Computersystem überhaupt, das dies erreicht hat. Am gleichen Tage erfolgte die Eintragung des Rekords in das Guinness-Buch der Rekorde.[84][85]
Am 7. Mai 2008 erreichte das Projekt ein nachhaltiges Leistungsniveau, das höher als zwei native PetaFLOPS war, gefolgt von den drei und vier PetaFLOPS-Meilensteinen am 20. August 2008 beziehungsweise am 28. September 2008.[86] Am 19. Juli 2016 wurde bekannt gegeben, dass man die Rechenleistung von 100 ×86-PetaFLOPS überschritten habe.[87]
COVID-19-Pandemie (2020)
Vor Ausbruch der COVID-19-Pandemie nahmen circa 30.000 Nutzer an dem Projekt teil.[88] In einem Video der Financial Times auf YouTube sprach Gregory Bowman am 7. April 2020 davon, dass man im Rahmen der Pandemie über 700.000 neue Nutzer hinzugewonnen habe.[89] Unter anderen rief Nvidia Computerspieler dazu auf, ihre GPU-Rechenleistung beizutragen.[90] Am 20. März 2020 verkündete Folding@Home, über die Rechenleistung von mehr als 470 x86-PetaFLOPS zu verfügen,[91] womit man den bisher schnellsten Supercomputer – den IBM Summit mit 148 PetaFLOPS – deutlich übertroffen hat. Am 13. April konnte das Projekt eine Rechenleistung von über 2,4 ×86-ExaFLOPS und über 1,4 Millionen Nutzer aufweisen und ist damit schneller als alle TOP500 Supercomputer der Welt zusammengenommen beziehungsweise 15-mal schneller als IBM Summit.[3] Bereits am 16. April überschritt die Rechenleistung die Marke von 2,6 ×86-ExaFLOPS.[92] Am 7. Oktober 2020 betrug die gesamte Rechenleistung nach einer Korrektur der Statistiken, die aufgrund eines Fehlers im System in die Höhe getrieben wurden, nur noch 0,3 ×86-ExaFLOPS.[93] Am 24. Februar 2021 war sie auf unter 0,19 ×86-ExaFLOPS abgesunken.[94] Seitdem erfolgte ein Anstieg auf 369 PetaFLOPS.[95]
Software
Jeder Benutzer eines PCs mit Windows, macOS oder Linux kann ein Client-Programm herunterladen, das als Dienst im Hintergrund arbeitet. Die Version 7.6.21 (im Oktober 2020) unterstützt Einkern- und Mehrkernprozessoren von AMD, ARM, Intel sowie auch Grafikkarten von Nvidia und AMD[96]. Der erste Client war im Jahr 2000 ein Bildschirmschoner, der lief, während der Computer sonst nicht in Gebrauch war.[97]
Professionelle Softwareentwickler sind für den größten Teil des Codes der Folding@home-Software verantwortlich, sowohl für die Client- als auch für die Serverseite. Zum Entwicklungsteam gehören Programmierer von Nvidia, ATI, Sony und Cauldron Development.[98]
Clients können nur von der offiziellen Folding@home-Website oder deren kommerziellen Partnern heruntergeladen werden und interagieren nur mit Folding@home-Computerdateien. Sie tauschen Daten ausschließlich mit den Webservern von Folding@home aus (über Port 8080, alternativ 80). Die Kommunikation wird hierbei mithilfe von 2048-Bit-Digitalsignaturen verifiziert.[99]
Der Client, GROMACS, diverse Cores und die grafische Benutzeroberfläche (GUI) des Clients sind quelloffen.[100]
Arbeitseinheit/Work-Unit
Eine Arbeitseinheit sind die Proteindaten, die der Client zu verarbeiten hat. Arbeitseinheiten sind ein Bruchteil der Simulation zwischen den Zuständen in einem Markov-Zustandsmodell. Nachdem die Arbeitseinheit heruntergeladen und vollständig vom Computer verarbeitet wurde, wird sie an die Folding@home-Server zurückgegeben, die dem Benutzer dann Kreditpunkte erteilen.[101] Dieser Zyklus wiederholt sich automatisch. Alle Arbeitseinheiten haben zugehörige Fristen, und wenn diese Fristen überschritten werden, werden diese Arbeitseinheiten automatisch an einen anderen Benutzer neu verteilt.[102] Da die Proteinfaltung seriell erfolgt und viele Arbeitseinheiten aus ihren jeweiligen Vorgängern generiert werden, kann der Simulationsprozess auch dann normal ablaufen, wenn eine Arbeitseinheit nicht nach einer angemessenen Zeitspanne zurück an den Server gesendet wurde.
Vor der öffentlichen Freigabe durchlaufen die Arbeitseinheiten mehrere Qualitätssicherungsschritte, um zu verhindern, dass problematische Einheiten vollständig verfügbar werden. Diese Testphasen umfassen interne, Beta- und fortgeschrittene Phasen, bevor eine endgültige vollständige Freigabe über Folding@home erfolgt.[103]
Die Arbeitseinheiten von Folding@home werden normalerweise nur einmal verarbeitet, außer in dem seltenen Fall, dass bei der Verarbeitung Fehler auftreten. Tritt dieser Fall bei drei verschiedenen Anwendern auf, wird die Einheit automatisch aus der Verteilung gezogen.[104]
Cores
Spezielle Molekulardynamik-Programme, die als FahCores bezeichnet werden und häufig mit Core (zu deutsch Kern) abgekürzt werden, führen die Berechnungen auf der Arbeitseinheit als Hintergrundprozess durch. Cores sind hierbei wissenschaftliche Computerprogramme, die speziell zur Ausführung von Berechnungen aus der Modifizierung und Optimierung von Molekulardynamik-Simulationen hervorgehen. Eine große Mehrheit der Cores, die von Folding@home genutzt werden, basiert auf GROMACS.[105] Weniger aktiv verwendete Cores sind ProtoMol und SHARPEN. Folding@home verwendete außerdem AMBER, CPMD, Desmond und TINKER, aber diese wurden inzwischen eingestellt.[106][107][108] Einige dieser Cores können die in der Computerchemie bekannten Wassermodelle darstellen, bei denen das umgebende Lösungsmittel (normalerweise Wasser) Atom für Atom modelliert wird (Explicit-Solvation-Methode). Andere Cores führen Implicit-Solvation-Methoden durch, bei denen das Lösungsmittel als mathematisches Kontinuum behandelt wird.[109][110] Der Core ist vom Client getrennt, damit die wissenschaftlichen Methoden automatisch aktualisiert werden können, ohne dass ein Client-Update erforderlich ist. Die Cores erstellen regelmäßig Berechnungs-Checkpoints, damit bei Unterbrechung der Berechnung diese wieder an dem Punkt fortgesetzt werden kann.[105]
GPU-Unterstützung
Der Folding@home-Client kann für die Berechnung, je nach Einstellung, neben der CPU auch die GPU heranziehen. Unterstützt werden Grafikkarten von Nvidia und AMD. Voraussetzung für Nvidia-Grafikkarten ist die CUDA-Technik (ab G80 mit GeForce-Treiber ab 174.55).[111] AMD-Grafikkarten werden ab der HD5000er-Serie unterstützt. Die Grafikeinheiten sämtlicher APUs von AMD, egal ob Desktop oder Notebook, können mittlerweile ebenso verwendet werden. Dabei greift der V7-Client auf den Standard OpenCL zurück.
PlayStation 3
Von März 2007 bis November 2012 nutzte Folding@home die Rechenleistung der PlayStation 3. Zum Zeitpunkt seiner Einführung lieferte der Haupt-Cell-Streamprozessor für einige Berechnungen eine zwanzigfache Geschwindigkeitssteigerung gegenüber PCs.[64][112] Die hohe Geschwindigkeit und Effizienz der PS3 eröffneten weitere Möglichkeiten für Optimierungen gemäß dem amdahlschen Gesetz (eine mathematische Gleichung, mit der die Gesamtbeschleunigung eines Programms bei paralleler Ausführung anhand , der parallele Anteil eines Programms und , der Anzahl der Prozessoren, berechnet werden kann). Sie veränderten das Verhältnis zwischen Recheneffizienz und Gesamtgenauigkeit erheblich, sodass komplexere molekulare Modelle bei geringen zusätzlichen Rechenkosten verwendet werden konnten.[113] Dies ermöglichte Folding@home die Durchführung biomedizinischer Berechnungen, die sonst rechnerisch nicht möglich gewesen wären.[114]
Der PS3-Client wurde in Zusammenarbeit mit Sony und Pande Lab entwickelt und am 23. März 2007 erstmals als eigenständiger Client veröffentlicht.[64][115] Durch die Veröffentlichung wurde Folding@home zum ersten verteilten Computerprojekt, das die PS3 verwendete.[116] Am 18. September des darauffolgenden Jahres wurde der PS3-Client bei seiner Markteinführung zu einem Kanal von Life mit PlayStation (eine ehemalige Multimedia-Anwendungssoftware des PlayStation Network).[117][118] Im Gegensatz zu Clients, die auf PCs ausgeführt werden, konnten Benutzer während der Ausführung von Folding@home keine anderen Aktivitäten auf ihrer PS3 ausführen.[114] Die einheitliche Konsolenumgebung der PS3 erleichterte den technischen Support und machte Folding@home benutzerfreundlicher.[64] Die PS3 hatte auch die Möglichkeit, Daten schnell auf ihren Grafikprozessor zu streamen, die zur Echtzeit-Visualisierung der aktuellen Proteindynamik auf atomarer Ebene verwendet wurde.[113]
Am 6. November 2012 beendete Sony die Unterstützung von Folding@home für den PS3-Client und andere unter für Life with PlayStation verfügbare Dienste. Während seiner fünfjährigen und siebenmonatigen Laufzeit haben mehr als 15 Millionen Benutzer mehr als 100 Millionen Stunden Rechenzeit für Folding@home bereitgestellt und das Projekt bei der Erforschung von Krankheiten maßgeblich unterstützt. Nach Gesprächen mit Pande Lab beschloss Sony, die Anwendung zu beenden. Vijay Pande betrachtete den PlayStation-3-Client als wichtigen Entwicklungsschritt für das Projekt.[119][120]
Client V7 für Mac, Windows & Linux
Der V7-Client ist die siebte und neueste Generation der Folding@home-Client-Software und stellt eine vollständige Neufassung und Vereinheitlichung der früheren Clients für die Betriebssysteme Windows, MacOS und Linux dar.[121][122]
Die Mindestsystemvoraussetzung für den Folding@home Client ist Stand März 2020 ein Pentium 4 1,4 GHz CPU.[123]
Er wurde am 22. März 2012 veröffentlicht. Wie seine Vorgänger kann V7 Folding@home im Hintergrund mit einer sehr niedrigen Priorität laufen, sodass andere Anwendungen die CPU-Ressourcen nach Bedarf nutzen können. Es ist so konzipiert, dass die Installation, die Inbetriebnahme und der Betrieb für Anfänger benutzerfreundlicher sind und den Forschern eine größere wissenschaftliche Flexibilität bieten als frühere Clients. V7 verwendet Trac für die Verwaltung seiner Bug-Tickets, so dass die Benutzer den Entwicklungsprozess sehen und Feedback geben können.
V7 besteht aus vier integrierten Elementen. Der Benutzer interagiert typischerweise mit der Open-Source-GUI von V7, genannt FAHControl. Diese verfügt über die Benutzeroberflächenmodi „Novice“, „Advanced“ und „Expert“ und bietet die Möglichkeit, viele entfernte Faltclients von einem Computer aus zu überwachen, zu konfigurieren und zu steuern.
FAHControl steuert den FAHClient, eine Back-End-Anwendung, die wiederum jeden FAHSlot (oder Slot) verwaltet.[124] Jeder Slot fungiert als Ersatz für die ehemals unterschiedlichen Folding@home v6 Uniprozessor-, SMP- oder GPU-Computer-Clients, da er Arbeitseinheiten unabhängig voneinander herunterladen, verarbeiten und hochladen kann. Die FAHViewer-Funktion, die dem Viewer der PS3 nachempfunden ist, zeigt, falls verfügbar, eine Echtzeit-3D-Darstellung des gerade bearbeiteten Proteins an.[125]
Chrome
Im Jahr 2014 wurde ein Client für die Webbrowser Google Chrome und Chromium veröffentlicht, mit dem Benutzer Folding@home in ihrem Webbrowser ausführen können. Der Client verwendete Googles Native-Client-Funktion (NaCl) auf Chromium-basierten Webbrowsern, um den Folding@Home-Code mit nahezu nativer Geschwindigkeit in einer Sandbox auf dem Rechner des Benutzers auszuführen. Aufgrund des Auslaufens von NaCl und Änderungen bei Folding@Home wurde der Web-Client im Juni 2019 endgültig abgeschaltet.[126]
Android
Im Juli 2015 wurde ein Client für Android-Handys auf Google Play für Geräte mit Android 4.4 KitKat oder neuer veröffentlicht.
Am 16. Februar 2018 wurde der Android-Client, der in Zusammenarbeit mit Sony angeboten wurde, aus dem Google Play Store entfernt. Es wurden Pläne angekündigt, in Zukunft eine Open-Source-Alternative anzubieten,[127] Stand August 2020 existierte jedoch kein neuer Android-Client.[128]
Motivationsanreize
Wie bei vielen anderen Projekten, die verteiltes Rechnen anwenden, werden auch bei Folding@home Statistiken in Form eines Punktesystems über die beigetragene Rechenleistung erstellt.[129][130] Durchschnittlich laufen zwischen 500 und 550 Projekte unter Folding@home,[131] wobei es für jedes Projekt individuelle Basispunkte gibt, die von einem Referenz-PC festgelegt werden. Für jede fertig gerechnete Arbeitseinheit (Work Unit) eines Projekts erhält der Benutzer die dafür vorgesehenen Basispunkte[132] Diese Punkte lassen sich jedoch vervielfachen (Bonuspunkte), je schneller eine Work Unit fertiggestellt wird. Voraussetzung für den Erhalt von Punkten ist eine Registrierung beim Projekt mit einem Benutzernamen. Um weitere Bonuspunkte zu erhalten, ist zusätzlich die Verwendung eines sogenannten „Passkey“ erforderlich.[133]
Jeder Benutzer kann wählen, ob seine Rechenleistung anonym (keine Punkte), nur unter seinem Benutzernamen oder auch für ein Team gezählt wird.
Wenn ein Benutzer kein neues Team bildet oder sich nicht einem bestehenden Team anschließt, wird dieser Benutzer automatisch Teil des „Standard“-Teams. Dieses „Standard“-Team hat die Teamnummer „0“. Die Statistiken werden sowohl für dieses „Standard“-Team als auch für speziell benannte Teams gesammelt.
Ergebnisse
Insgesamt wurden 224 wissenschaftliche Publikationen (Stand 15. Mai 2020) als direktes Ergebnis von Folding@home veröffentlicht.[134][135]
Verwandte Projekte
Rosetta@home, Predictor@home und POEM@home waren bzw. sind Projekte, die das gleiche Ziel haben, aber andere Methoden anwenden. Foldit etwa ist ein experimentelles Computerspiel, das Wissenschaftlern bei der Optimierung von Proteinen helfen soll.[136] Das Projekt DreamLab ermöglicht es Smartphones während des Ladevorganges Prozessorkapazitäten für die medizinische Forschung des Imperial College London bereitzustellen.
Rosetta@home
Rosetta@home ist ein Projekt für verteiltes Rechnen zur Vorhersage von Proteinstrukturen und ist einer der genauesten Tertiärstruktur-Prädiktoren.[137][138] Die Konformationszustände aus der Software von Rosetta können zur Initialisierung eines Markov-Zustandsmodells als Ausgangspunkt für Folding@home-Simulationen verwendet werden. Umgekehrt können die Algorithmen zur Strukturvorhersage aus thermodynamischen und kinetischen Modellen sowie die Sampling-Aspekte von Proteinfaltungssimulationen verbessert werden. Da Rosetta nur versucht, den endgültigen Faltungszustand vorherzusagen, nicht aber, wie die Faltung abläuft, sind Rosetta@home und Folding@home komplementär und adressieren sehr unterschiedliche molekulare Fragestellungen.[139]
Foldit
Ziel von Foldit ist es, ein möglichst gut „gefaltetes“ Protein zu erhalten, d. h. ein Modell des Proteins im Zustand des Energieminimums. Das ist die Form, in der es in der Natur vorkommt. Dazu sind allerdings keinerlei Vorkenntnisse nötig, die Bewertung erledigt das Programm. Die Möglichkeiten, die der Spieler zur Proteinmanipulation hat, werden in einer Serie von Tutorialpuzzles erklärt. Für das Spiel wird dabei eine grafische Entsprechung der Proteinstruktur angezeigt, die der Spieler mit verschiedenen Werkzeugen verändern kann. Wenn die Struktur verändert wird, berechnet das Programm einen Punktwert basierend darauf, wie gut das Protein gefaltet ist. Für jedes Puzzle wird ein Highscore sowohl für Einzel- als auch für Gruppenlösungen errechnet, der sich in Echtzeit ändert. Foldit ist der Versuch, die natürlichen menschlichen 3-D-Mustererkennungsfähigkeiten auf dieses Problem anzusetzen. Gegenwärtige Puzzles basieren auf gut verstandenen Proteinen. Indem untersucht wird, auf welche Art die Spieler intuitiv an diese Puzzles herangehen, versuchen die Wissenschaftler, existierende Proteinfaltungssoftware zu verbessern.
Anton
Anton ist ein Supercomputer, der für Molekulardynamik-Simulationen gebaut wurde. Im Oktober 2011 waren Anton und Folding@home die beiden leistungsstärksten Molekulardynamiksysteme.[140] Anton ist einzigartig in seiner Fähigkeit, einzelne ultralange, rechenintensive Molekültrajektorien zu erzeugen[141] wie zum Beispiel eine im Jahr 2010, die den Millisekundenbereich erreichte.[142][143] Diese langen Trajektorien können besonders hilfreich für einige Arten von biochemischen Problemen sein.[144][145] Anton verwendet jedoch keine Markov-Zustandsmodelle (MSM) für die Analyse. Im Jahr 2011 konstruierte das Pande-Labor ein MSM aus zwei 100-Mikrosekunden Anton-Simulationen und fand alternative Faltungswege, die durch Antons traditionelle Analyse nicht sichtbar waren. Sie kamen zu dem Schluss, dass es kaum einen Unterschied zwischen MSMs gab, die aus einer begrenzten Anzahl langer Bahnen konstruiert wurden, und solchen, die aus vielen kürzeren Bahnen zusammengesetzt wurden. Im Juni 2011 fügte Folding@home die Probenahme einer Anton-Simulation hinzu, um besser zu bestimmen, wie ihre Methoden im Vergleich zu Antons Methoden aussehen. Im Gegensatz zu den kürzeren Bahnen von Folding@home, die für verteilte Berechnungen und andere Parallelisierungsmethoden besser geeignet sind, erfordern längere Bahnen keine adaptive Probenahme, um den Phasenraum des Proteins ausreichend zu beproben. Aus diesem Grund ist es möglich, dass eine Kombination der Simulationsmethoden von Anton und Folding@home eine gründlichere Abtastung dieses Raumes ermöglichen würde.[141]
Weblinks
Einzelnachweise
- Vijay S. Pande, Kyle Beauchamp, Gregory R. Bowman: Everything you wanted to know about Markov State Models but were afraid to ask. In: Methods (San Diego CA). Band 52, Nr. 1, September 2010, ISSN 1046-2023, S. 99–105, doi:10.1016/j.ymeth.2010.06.002, PMID 20570730, PMC 2933958 (freier Volltext).
- Maria Temming: You can help fight the coronavirus. All you need is a computer. In: Science News. 25. März 2020, abgerufen am 26. März 2020 (amerikanisches Englisch).
- Folding@Home mit 2,4 Exaflops schneller als die Top 500 Supercomputer. 14. April 2020, abgerufen am 14. April 2020.
- New Folding@home software with the option to prioritize COVID-19 projects
- Vijay Pande: History of Folding@Home. Stanford University, 14. April 2007, abgerufen am 23. März 2020 (englisch).
- Public Launch. Stanford University, 19. September 2000, abgerufen am 22. März 2020 (englisch): „We released our software to the public and very soon after we had thousands of computers donating otherwise unused computer power.“
- Julia Evangelou Strait: Bowman leading international supercomputing project. Washington University School of Medicine in St. Louis, 26. Februar 2019, abgerufen am 19. März 2020 (englisch): „With this networked computing power, Folding@home is, essentially, one of the world’s largest supercomputers.“
- Folding@Home FAQ. In: foldingathome.org. Abgerufen am 6. März 2020 (englisch).
- Vijay Pande: Folding@home and Simbios. In: typepad.com. 23. April 2008, abgerufen am 23. März 2020 (englisch): „Second, we’re also starting to make large data sets from Folding@home available to others. You can see some of the first data sets on this project page, and we expect to put more data up as time goes on. Folding@home donors have generated wonderful data sets that aren’t possible to generate by other means, and our hope is to publish them so that other scientists can data mine them for other purposes as well.“
- Vijay Pande: Changes to F@h Website. In: foldingforum.org. 25. Oktober 2011, abgerufen am 23. März 2020 (englisch). “We have been making these available in general on request and in cases where people ask for data sets repeatedly (eg simtk.org we make them available on a website linked from folding.stanford.edu.”
- Matthew P. Harrigan, Mohammad M. Sultan, Carlos X. Hernández, Brooke E. Husic, Peter Eastman: MSMBuilder: Statistical Models for Biomolecular Dynamics. In: Biophysical Journal. Band 112, Nr. 1, 10. Januar 2017, ISSN 1542-0086, S. 10–15, doi:10.1016/j.bpj.2016.10.042, PMID 28076801, PMC 5232355 (freier Volltext).
- SimTK: MSMBuilder: Neuigkeiten. In: simtk.org. Abgerufen am 18. März 2020.
- Biophysical Society Names Five 2012 Award Recipients. (Nicht mehr online verfügbar.) 29. August 2011, archiviert vom Original am 21. September 2012; abgerufen am 18. März 2020 (englisch).
- Award. (Memento vom 21. September 2012 auf WebCite) In: folding.stanford.edu, abgerufen am 18. März 2020.
- C. Pratt, K. Cornely: Thermodynamics. Essential Biochemistry. Wiley-VCH, 2004, ISBN 978-0-471-39387-0.
- Vijay Pande [Stanford University]: When and where do Proteins fold? auf YouTube, 16. August 2007, abgerufen am 23. März 2020 (englisch).
- G. Zhang, Z. Ignatova: Folding at the birth of the nascent chain: coordinating translation with co-translational folding. In: Current opinion in structural biology. Band 21, Nummer 1, Februar 2011, S. 25–31, doi:10.1016/j.sbi.2010.10.008, PMID 21111607 (Review).
- B. van den Berg, R. Wain, C. M. Dobson, R. J. Ellis: Macromolecular crowding perturbs protein refolding kinetics: implications for folding inside the cell. In: The EMBO Journal. Band 19, Nummer 15, August 2000, S. 3870–3875, doi:10.1093/emboj/19.15.3870, PMID 10921869, PMC 306593 (freier Volltext).
- S. Al-Karadaghi: Torsion Angles and the Ramachandran Plot. In: proteinstructures.com. Abgerufen am 17. März 2020.
- F. Marinelli, F. Pietrucci, A. Laio, S. Piana: A kinetic model of trp-cage folding from multiple biased molecular dynamics simulations. In: PLoS Computational Biology. Band 5, Nummer 8, August 2009, S. e1000452, doi:10.1371/journal.pcbi.1000452, PMID 19662155, PMC 2711228 (freier Volltext).
- So much more to know … In: Science. Band 309, Nummer 5731, Juli 2005, S. 78–102, doi:10.1126/science.309.5731.78b, PMID 15994524.
- H. Ecroyd, J. A. Carver: Unraveling the mysteries of protein folding and misfolding. In: IUBMB life. Band 60, Nummer 12, Dezember 2008, S. 769–774, doi:10.1002/iub.117, PMID 18767168 (Review).
- Y. Chen, F. Ding, H. Nie, A. W. Serohijos, S. Sharma, K. C. Wilcox, S. Yin, N. V. Dokholyan: Protein folding: then and now. In: Archives of biochemistry and biophysics. Band 469, Nummer 1, Januar 2008, S. 4–19, doi:10.1016/j.abb.2007.05.014, PMID 17585870, PMC 2173875 (freier Volltext).
- L. M. Luheshi, D. C. Crowther, C. M. Dobson: Protein misfolding and disease: from the test tube to the organism. In: Current opinion in chemical biology. Band 12, Nummer 1, Februar 2008, S. 25–31, doi:10.1016/j.cbpa.2008.02.011, PMID 18295611 (Review).
- C. D. Snow, E. J. Sorin, Y. M. Rhee, V. S. Pande: How well can simulation predict protein folding kinetics and thermodynamics? In: Annual review of biophysics and biomolecular structure. Band 34, 2005, S. 43–69, doi:10.1146/annurev.biophys.34.040204.144447, PMID 15869383 (Review).
- V. A. Voelz, G. R. Bowman, K. Beauchamp, V. S. Pande: Molecular simulation of ab initio protein folding for a millisecond folder NTL9(1–39). In: Journal of the American Chemical Society. Band 132, Nummer 5, Februar 2010, S. 1526–1528, doi:10.1021/ja9090353, PMID 20070076, PMC 2835335 (freier Volltext).
- A. Verma, S .M. Gopal, A. Schug, J. S. Oh, K. V. Klenin K. H. Lee, W. Wenzel: Massively Parallel All Atom Protein Folding in a Single Day In: Advances in Parallel Computing. Band 15, 2008, S. 527–534, ISBN 978-1-58603-796-3.
- V. S. Pande, I. Baker, J. Chapman, S. P. Elmer, S. Khaliq, S. M. Larson, Y. M. Rhee, M. R. Shirts, C. D. Snow, E. J. Sorin, B. Zagrovic: Atomistic protein folding simulations on the submillisecond time scale using worldwide distributed computing. In: Biopolymers. Band 68, Nummer 1, Januar 2003, S. 91–109, doi:10.1002/bip.10219, PMID 12579582.
- G. R. Bowman, V. A. Voelz, V. S. Pande: Taming the complexity of protein folding. In: Current opinion in structural biology. Band 21, Nummer 1, Februar 2011, S. 4–11, doi:10.1016/j.sbi.2010.10.006, PMID 21081274, PMC 3042729 (freier Volltext) (Review).
- John D. Chodera, William C. Swope, Jed W. Pitera, Ken A. Dill: Long-Time Protein Folding Dynamics from Short-Time Molecular Dynamics Simulations. In: Multiscale Modeling & Simulation. Band 5, 2006, S. 1214, doi:10.1137/06065146X.
- R. B. Best: Atomistic molecular simulations of protein folding. In: Current opinion in structural biology. Band 22, Nummer 1, Februar 2012, S. 52–61, doi:10.1016/j.sbi.2011.12.001, PMID 22257762 (Review).
- T. J. Lane, Gregory Bowman, Robert McGibbon, Christian Schwantes, Vijay Pande, Bruce Borden: Folding@home Simulation FAQ. (Nicht mehr online verfügbar.) Folding@home, 21. September 2012, archiviert vom Original am 21. September 2012; abgerufen am 17. März 2020.
- V. S. Pande, K. Beauchamp, G. R. Bowman: Everything you wanted to know about Markov State Models but were afraid to ask. In: Methods. Band 52, Nummer 1, September 2010, S. 99–105, doi:10.1016/j.ymeth.2010.06.002, PMID 20570730, PMC 2933958 (freier Volltext) (Review).
- G. R. Bowman, D. L. Ensign, V. S. Pande: Enhanced modeling via network theory: Adaptive sampling of Markov state models. In: Journal of chemical theory and computation. Band 6, Nummer 3, 2010, S. 787–794, doi:10.1021/ct900620b, PMID 23626502, PMC 3637129 (freier Volltext).
- Vijay Pande: FAHcon 2012: Thinking about how far FAH has come. In: typepad.com. Folding@home, 8. Juni 2012, abgerufen am 17. März 2020.
- Brooke E. Husic, Vijay S. Pande: Markov State Models: From an Art to a Science. In: Journal of the American Chemical Society. Band 140, Nr. 7, 21. Februar 2018, ISSN 1520-5126, S. 2386–2396, doi:10.1021/jacs.7b12191, PMID 29323881.
- C. D. Snow, H. Nguyen, V. S. Pande, M. Gruebele: Absolute comparison of simulated and experimental protein-folding dynamics. In: Nature. Band 420, Nummer 6911, November 2002, S. 102–106, doi:10.1038/nature01160, PMID 12422224.
- K. A. Beauchamp, D. L. Ensign, R. Das, V. S. Pande: Quantitative comparison of villin headpiece subdomain simulations and triplet-triplet energy transfer experiments. In: Proceedings of the National Academy of Sciences. Band 108, Nummer 31, August 2011, S. 12734–12739, doi:10.1073/pnas.1010880108, PMID 21768345, PMC 3150881 (freier Volltext).
- Pande Lab Science [Stanford University]: Simulation of millisecond protein folding: NTL9 (from Folding@home) auf YouTube, 18. Januar 2010, abgerufen am 23. März 2020 (englisch. Simulating protein folding on the millisecond timescale has been a major challenge for many years. In a recent paper (doi:10.1021/ja9090353), Folding@home researchers Vincent Voelz, Greg Bowman, Kyle Beauchamp, and Vijay Pande have broken this barrier. This is a movie of one of the trajectories that folded (i.e. started unfolded and ended up in the folded state)).
- T. H. Click, D. Ganguly, J. Chen: Intrinsically disordered proteins in a physics-based world. In: International Journal of Molecular Sciences. Band 11, Nummer 12, 2010, S. 5292–5309, doi:10.3390/ijms11125292, PMID 21614208, PMC 3100817 (freier Volltext) (Review).
- Which diseases or biomedical problems are you currently studying? (Nicht mehr online verfügbar.) Stanford University, 12. September 2012, archiviert vom Original am 21. September 2012; abgerufen am 18. März 2020 (englisch).
- Greg Bowman: Folding Forum • Login. 22. August 2011, abgerufen am 18. März 2020 (englisch): „This A3 project for windows and linux clients aims to characterize the dynamics of RNase H, a key component of HIV. By understanding the role of dynamics in its mechanism, we hope to be better able to design drugs to deactivate this enzyme.“
- Hana Robson Marsden, Itsuro Tomatsu, Alexander Kros: Model systems for membrane fusion. In: Chemical Society Reviews. Band 40, Nr. 3, 22. Februar 2011, ISSN 1460-4744, S. 1572–1585, doi:10.1039/C0CS00115E.
- Gregory R Bowman, Xuhui Huang, Vijay S Pande: Network models for molecular kinetics and their initial applications to human health. In: Cell research. Band 20, Nr. 6, Juni 2010, ISSN 1001-0602, S. 622–630, doi:10.1038/cr.2010.57, PMID 20421891, PMC 4441225 (freier Volltext).
- Peter M. Kasson, Afra Zomorodian, Sanghyun Park, Nina Singhal, Leonidas J. Guibas: Persistent voids: a new structural metric for membrane fusion. In: Bioinformatics. Band 23, Nr. 14, 15. Juli 2007, ISSN 1367-4803, S. 1753–1759, doi:10.1093/bioinformatics/btm250 (academic.oup.com [abgerufen am 18. März 2020]).
- Peter M. Kasson, Daniel L. Ensign, Vijay S. Pande: Combining molecular dynamics with Bayesian analysis to predict and evaluate ligand-binding mutations in influenza hemagglutinin. In: Journal of the American Chemical Society. Band 131, Nr. 32, 19. August 2009, ISSN 0002-7863, S. 11338–11340, doi:10.1021/ja904557w, PMID 19637916, PMC 2737089 (freier Volltext).
- Protein folding and viral infection. Abgerufen am 18. März 2020.
- Christian Gruber, Georg Steinkellner: Wuhan coronavirus 2019-nCoV – what we can find out on a structural bioinformatics level. In: Innophore. 29. Januar 2020. doi:10.6084/m9.figshare.11752749.
- Sean Fleming: These researchers want to use your computer in the search for a COVID-19 vaccine. World Economic Forum, 17. März 2020, abgerufen am 22. März 2020 (englisch).
- Kiona Smith: SETI@Home Is Over; The Fight Against COVID-19 Coronavirus Is Just Beginning. Forbes Magazine, 15. März 2020, abgerufen am 22. März 2020 (englisch): „Folding@Home (a program similar to SETI@Home that focuses on disease research – specifically how proteins fold) just rolled out “an initial wave of projects” that simulate how proteins from SARS-CoV-2 (the virus that causes COVID-19) work and how they interact with human cells.“
- Greg Bowman: FOLDING@HOME TAKES UP THE FIGHT AGAINST COVID-19 / 2019-NCOV. 27. Februar 2020, abgerufen am 18. März 2020 (englisch).
- Folding@home Turns Its Massive Crowdsourced Computer Network Against COVID-19. 16. März 2020, abgerufen am 18. März 2020 (amerikanisches Englisch).
- John Chodera: FOLDING@HOME UPDATE ON SARS-COV-2 (10 MAR 2020). 10. März 2020, abgerufen am 18. März 2020 (englisch).
- Julia Evangelou Strait: Crowdsourced supercomputing project sets sights on coronavirus. Washington University School of Medicine in St. Louis, 19. März 2020, abgerufen am 19. März 2020 (englisch).
- Greg Bowman: CORONAVIRUS – WHAT WE’RE DOING AND HOW YOU CAN HELP IN SIMPLE TERMS. Washington University, 15. März 2020, abgerufen am 22. März 2020 (englisch): „We’re simulating the dynamics of COVID-19 proteins to hunt for new therapeutic opportunities.“
- G Brent Irvine; Omar M El-Agnaf; Ganesh M Shankar; Dominic M Walsh: Protein Aggregation in the Brain: The Molecular Basis for Alzheimer’s and Parkinson’s Diseases. Molecular Medicine (review). 14 (7–8): 451–464, 2008.
- Claudio Soto; Lisbell D. Estrada: Protein Misfolding and Neurodegeneration. Archives of Neurology (review). 65 (2): 184–189, 2008.
- Robin Roychaudhuri; Mingfeng Yang; Minako M. Hoshi; David B. Teplow: Amyloid β-Protein Assembly and Alzheimer Disease. Journal of Biological Chemistry. 284 (8): 4749–53, 2008.
- Nicholas W. Kelley; V. Vishal; Grant A. Krafft; Vijay S. Pande: Simulating oligomerization at experimental concentrations and long timescales: A Markov state model approach. Journal of Chemical Physics. 129 (21): 214707, 2008.
- Aabgeena Naeem; Naveed Ahmad Fazili: Defective Protein Folding and Aggregation as the Basis of Neurodegenerative Diseases: The Darker Aspect of Proteins. Cell Biochemistry and Biophysics (review). 61 (2): 237–50, 2011.
- Gregory R Bowman; Xuhui Huang; Vijay S Pande: Network models for molecular kinetics and their initial applications to human health. Cell Research (review). 20 (6): 622–630, 2010.
- Vijay Pande: New FAH results on possible new Alzheimer’s drug presented. Stanford University, 18. Dezember 2008, abgerufen am 23. März 2020 (englisch).
- J. Rajadas, C. W. Liu, P. Novick, N. W. Kelley, M. Inayathullah, M. C. Lemieux, V. S. Pande: Rationally designed turn promoting mutation in the amyloid-β peptide sequence stabilizes oligomers in solution. In: PLOS ONE. Band 6, Nummer 7, 2011, S. e21776, doi:10.1371/journal.pone.0021776, PMID 21799748, PMC 3142112 (freier Volltext).
- Pande lab: Folding@home Diseases Studied FAQ. (Memento vom 21. September 2012 auf WebCite) In: folding.stanford.edu, 2012, Abgerufen am 18. März 2020.
- Walker FO: Huntington’s disease. In: Lancet. 369 (9557): 218–28, 2007, S. 220
- Nicholas W. Kelley; Xuhui Huang; Stephen Tam; Christoph Spiess; Judith Frydman; Vijay S. Pande: The predicted structure of the headpiece of the Huntingtin protein and its implications on Huntingtin aggregation. In: Journal of Molecular Biology, 2009, 388 (5), S. 919–927. doi:10.1016/j.jmb.2009.01.032
- Susan W Liebman: Protein folding: Sticky N17 speeds huntingtin pile-up. Springer Nature Limited, Januar 2010, abgerufen am 23. März 2020 (englisch): „Aggregation of huntingtin protein with an expanded polyglutamine region is enhanced by its 17-residue N-terminal domain, which binds to itself and to the polyglutamine region. This enhancement is inhibited when the N-terminal domain binds to the chaperonin TRiC.“
- M Hollstein; D Sidransky; B Vogelstein; CC Harris (1991). p53 mutations in human cancers. Science. 253 (5015): 49–53.
- Lillian T Chong: Dimerization of the p53 Oligomerization Domain: Identification of a Folding Nucleus by Molecular Dynamics Simulations. ScienceDirect, 28. Januar 2005, abgerufen am 23. März 2020 (englisch).
- Lillian T. Chong; William C. Swope; Jed W. Pitera; Vijay S. Pande: Kinetic Computational Alanine Scanning: Application to p53 Oligomerization. In: Journal of Molecular Biology. 2005. 357 (3): 1039–1049.
- Almeida MB, do Nascimento JL, Herculano AM, Crespo-López ME: Molecular chaperones: toward new therapeutic tools. Journal of Molecular Biology, 65 (4): 239–43, 2011.
- Stanford University Medical Center: Scientists boost potency, reduce side effects of IL-2 protein used to treat cancer. MedicalXPress, 18. März 2012, abgerufen am 23. März 2020 (englisch).
- Aron M. Levin; Darren L. Bates; Aaron M. Ring; Carsten Krieg; Jack T. Lin; Leon Su; Ignacio Moraga; Miro E. Raeber; Gregory R. Bowman; Paul Novick; Vijay S. Pande; C. Garrison Fathman; Onur Boyman; K. Christopher Garcia: Exploiting a natural conformational switch to engineer an interleukin-2 ‘superkine’. In: Nature. 484 (7395): 529–33, 2012.
- Sanghyun Park, Randall J. Radmer, Teri E. Klein: A new set of molecular mechanics parameters for hydroxyproline and its use in molecular dynamics simulations of collagen-like peptides. 16. September 2005, abgerufen am 23. März 2020 (englisch).
- Vijay Pande: New methods for computational drug design. Folding@home, typepad.com; Archivierung des Originals am 21. September 2012; abgerufen am 20. März 2020.
- Guha Jayachandran, M. R. Shirts, S. Park; V. S. Pande: Parallelized-Over-Parts Computation of Absolute Binding Free Energy with Docking and Molecular Dynamics. In: Journal of Chemical Physics, 2006, 125 (8), S. 084901.
- Chun Song, Shen Lim, Joo Tong: Recent advances in computer-aided drug design. In: Briefings in Bioinformatics, 2009, 10 (5), S. 579–591 (review).
- Pande Laboratory: Project 10721 Description. (Memento vom 21. September 2012 auf WebCite) Folding@home. foldingathome.org; abgerufen am 20. März 2020.
- Gregory Bowman: Searching for new drug targets. Folding@home. foldingathome.org. Archivierung des Originals am 21. September 2012. Abgerufen am 20. März 2020.
- Gregory R. Bowman; Phillip L. Geissler: Equilibrium fluctuations of a single folded protein reveal a multitude of potential cryptic allosteric sites. PNAS. 109 (29): 11681–6, 2012.
- Paula M. Petrone; Christopher D. Snow; Del Lucent; Vijay S. Pande: Side-chain recognition and gating in the ribosome exit tunnel. In: Proceedings of the National Academy of Sciences. 105 (43): 16549–54, 2008.
- Prof. Stefan Voelz: Folding@home Project Description. Abgerufen am 23. März 2020 (englisch).
- What are native FLOPS? In: FAQs. Folding@Home, abgerufen am 23. März 2020 (englisch): „We refer to the FLOP count on a given hardware as the native FLOP count. For example, an exponential on a GPU is one native GPU FLOP but many native ×86 FLOPS.“
- Crossing the petaflop barrier. In: Folding@home A blog all about Folding@home, from its Director, Prof. Vijay Pande. 16. September 2007, abgerufen am 6. März 2020 (englisch).
- Most powerful distributed computing network. Guinness World Records Limited, 16. September 2007, abgerufen am 22. März 2020 (englisch): „On 16 September 2007 Folding@home, a distributed computing network operating from Stanford University (USA) achieved a computing power of 1 petaflop – or 1 quadrillion floating point operations per second.“
- Guest Recorder: 3 PetaFLOP barrier – PetScience. LongeCity – Advocacy & Research for Unlimited Lifespans, 19. August 2008, abgerufen am 6. März 2020 (englisch).
- Folding@Home: A significant milestone: 100 petaFLOPS. 19. Juli 2016, abgerufen am 6. März 2020 (englisch).
- Michael Günsch: Rosetta@home: Aktive Nutzer durch Kampf gegen Coronavirus verdoppelt. In: ComputerBase.
- Financial Times: Coronavirus: how your computer could help find a vaccine | FT auf YouTube, 7. April 2020, abgerufen am 7. April 2021 (englisch. „In the past three week, though, we had over 700.000 people join the project.“).
- Connor Sheridan: Nvidia’s calling on gaming PC owners to put their systems to work fighting COVID-19. In: gamesradar.com. 13. März 2020, abgerufen am 26. April 2020 (englisch).
- Greg Bowman: Amazing! @foldingathome now has over 470 petaFLOPS of compute power. To put that in perspective, that’s more than 2x the peak performance of the Summit super computer! In: @drGregBowman. Twitter, 20. März 2020, abgerufen am 20. März 2020 (englisch).
- Folding@home stats report. Abgerufen am 16. April 2020.
- Folding@home stats report. Abgerufen am 7. Oktober 2020.
- Folding@home stats report. Abgerufen am 24. Februar 2021.
- Folding@home stats report. Abgerufen am 15. Mai 2021.
- Emma: new client with arm support. 24. November 2020, abgerufen am 26. November 2020 (englisch).
- Michael Shirts: Screen Savers of the World Unite. 2000, abgerufen am 23. März 2020 (englisch).
- How does FAH code development and sysadmin get done? Abgerufen am 18. März 2020.
- FAQ Folding at Home. Pande Lab, 21. September 2012, abgerufen am 18. März 2020 (englisch).
- OpenSource – Folding@home.
- WHAT IS A WORK UNIT? Abgerufen am 19. März 2020 (englisch).
- Running Folding@Home. In: foldingathome.org. Abgerufen am 23. März 2020 (englisch): „These unfinished work units ‘expire’ and are reassigned to new machines. You will still receive credit for all WUs completed and uploaded prior to the Timeout (formerly preferred deadline).“
- More transparency in testing. Abgerufen am 19. März 2020.
- Folding Forum • View topic – Gromacs cannot continue further. Abgerufen am 19. März 2020.
- Adam Beberg, Daniel Ensign, Guha Jayachandran, Siraj Khaliq, Vijay Pande: Folding@home: Lessons from eight years of volunteer distributed computing. (PDF; 304 kB) In: hicomb.org. 2009 IEEE International Symposium on Parallel & Distributed Processing. Proceedings, 2009, abgerufen am 18. März 2020.
- Pande lab: Opensource. (Nicht mehr online verfügbar.) In: webcitation.org. 3. August 2012, archiviert vom Original am 3. März 2020; abgerufen am 18. März 2020.
- Pande lab: Folding@home Frequently Asked Questions (FAQ) Index. (Nicht mehr online verfügbar.) In: webcitation.org. 21. September 2012, archiviert vom Original am 21. August 2012; abgerufen am 18. März 2020.
- Vijay Pande: Update on new FAH cores and clients. In: folding.typepad.com. Folding@home, 25. September 2009, abgerufen am 18. März 2020.
- M. S. Friedrichs, P. Eastman, V. Vaidyanathan, M. Houston, S. Legrand, A. L. Beberg, D. L. Ensign, C. M. Bruns, V. S. Pande: Accelerating molecular dynamic simulation on graphics processing units. In: Journal of computational chemistry. Band 30, Nummer 6, April 2009, S. 864–872, doi:10.1002/jcc.21209, PMID 19191337, PMC 2724265 (freier Volltext).
- Pande lab: Folding@home Petaflop Initiative (FPI). (Nicht mehr online verfügbar.) In: webcitation.org. 19. August 2012, archiviert vom Original am 21. September 2012; abgerufen am 18. März 2020.
- ComputerBase: Folding@Home Client für Nvidia-GPUs ist raus
- Futures in Biotech 27: Folding@home at 1.3 Petaflops. (Nicht mehr online verfügbar.) In: Castroller.com. CastRoller, 28. Dezember 2007, archiviert vom Original am 21. September 2012; abgerufen am 18. März 2020.
- E. Luttmann, D. L. Ensign, V. Vaidyanathan, M. Houston, N. Rimon, J. Øland, G. Jayachandran, M. Friedrichs, V. S. Pande: Accelerating molecular dynamic simulation on the cell processor and Playstation 3. In: Journal of computational chemistry. Band 30, Nummer 2, Januar 2009, S. 268–274, doi:10.1002/jcc.21054, PMID 18615421.
- David E. Williams: PlayStation’s serious side: Fighting disease. In: CNN. 20. Oktober 2006, abgerufen am 18. März 2020.
- Jerry Liao: The Home Cure: PlayStation 3 to Help Study Causes of Cance. (Nicht mehr online verfügbar.) In: mb.com. Manila Bulletin Publishing Corporation, 23. März 2007, archiviert vom Original am 21. September 2012; abgerufen am 18. März 2020.
- Lou Kesten: Week in video-game news: 'God of War II' storms the PS2; a PS3 research project. In: Post-Gazette.com. Pittsburgh Post-Gazette, 26. März 2007, abgerufen am 18. März 2020.
- Elaine Chow: PS3 News Service, Life With Playstation, Now Up For Download. In: Gizmodo.com. Gizmodo, 18. September 2008, abgerufen am 18. März 2020.
- Vijay Pande: Life with Playstation -- a new update to the FAH/PS3 client. In: typepad.com. Folding@home, 18. September 2008, abgerufen am 18. März 2020.
- Eric Lempel: PS3 System Software Update (v4.30). In: PlayStation blog. Sony, 21. Oktober 2012, abgerufen am 18. März 2020.
- Termination of Life with PlayStation. (Nicht mehr online verfügbar.) In: Life with PlayStation. Sony, 6. November 2012, archiviert vom Original am 21. September 2012; abgerufen am 18. März 2020.
- Windows V7 Client. (Nicht mehr online verfügbar.) Stanford University, 18. August 2012, archiviert vom Original am 21. September 2012; abgerufen am 6. März 2020 (englisch): „We are pleased to say that everything is new in this version, using completely new software coding from the ground up.“
- Client version 7 now in open beta. 9. März 2011, abgerufen am 6. März 2020 (englisch): „I am happy to announce that after many months of development and testing the new version 7 Folding@home client software is now available for open-beta testing. The V7 client is a complete rewrite of the previous client for Windows, OS-X and Linux with the following goals.“
- Requirements. In: foldingathome.org. Abgerufen am 23. März 2020 (englisch): „CPU Slot Requirements Windows XP SP3 or newer, 32 or 64 bit Intel P4 1.4 GHz processor or newer, or AMD equivalent (modern multi-core processors recommended)“
- Link to GPL in FAHControl. (Nicht mehr online verfügbar.) 3. Oktober 2011, archiviert vom Original am 12. Oktober 2012; abgerufen am 6. März 2020 (englisch).
- Folding@home 3D Viewer. Github, abgerufen am 23. März 2020 (englisch): „The Folding@home viewer allows you to visualize protein folding simulations and monitor the status of the simulation work units as they run on your computer. Installing and running the viewer is not necessary to run Folding@home.“
- Folding@home Chrome Client. Abgerufen am 6. März 2020 (englisch): „Our decision to retire the NaCl client was due to a combination of Google deprecating NaCl and infrastructure upgrades at Folding@home which would have required extra effort to continue to support the NaCl folding client.“
- Anto Thynell: android-client-overhaul. 2. Februar 2018, abgerufen am 6. März 2020 (englisch): „From the 16th of February 2018, it will no longer be possible to use the Folding@Home Android client from Sony“
- Alternative Downloads / Clients. Abgerufen am 19. März 2020 (englisch).
- FAQ-Points (Memento vom 19. Juli 2013 im Internet Archive) Folding@home
- Vickie Curtis: Patterns of Participation and Motivation in Folding@home: The Contribution of Hardware Enthusiasts and Overclockers. In: Citizen Science: Theory and Practice. Band 3, Nr. 1, 27. April 2018, ISSN 2057-4991, S. 5, doi:10.5334/cstp.109 (citizenscienceassociation.org [abgerufen am 20. März 2020]).
- Projects Summary. Folding@Home
- Folding@home Active Projects
- FAQ-Passkey FAQ Points Passkey. Folding@home (englisch) abgerufen am 27. Februar 2021
- Papers & Results. Auf FoldingAtHome, abgerufen am 15. Mai 2020.
- Research from Stanford University. Stanford University, abgerufen am 23. März 2020 (englisch).
- The Science Behind Foldit. In: Foldit. Vanderbilt University, abgerufen am 19. März 2020 (englisch).
- Marc F. Lensink, Raúl Méndez, Shoshana J. Wodak: Docking and scoring protein complexes: CAPRI 3rd Edition. In: Proteins: Structure, Function, and Bioinformatics. Band 69, Nr. 4, 2007, ISSN 1097-0134, S. 704–718, doi:10.1002/prot.21804.
- What is Rosetta at Home? Abgerufen am 19. März 2020 (englisch).
- Vijay Grande: Folding@home vs. Rosetta@home. 5. März 2006, abgerufen am 19. März 2020 (englisch): „However, Rosetta and Folding@Home are addressing very different problems. Rosetta only predicts the final folded state, not how do proteins fold (and Rosetta has nothing to do with protein misfolding). Thus, those methods are not useful for the questions we’re interested in and the diseases we’re tackling (Alzheimer’s Disease and other aggregation related diseases).“
- Vijay Pande: Comparison between FAH and Anton’s approaches. Folding@home. typepad.com. Archivierung des Originals am 21. September 2012. Abgerufen am 18. März 2020
- Thomas J. Lane, Gregory R. Bowman, Kyle A Beauchamp, Vincent Alvin Voelz, Vijay S. Pande: Markov State Model Reveals Folding and Functional Dynamics in Ultra-Long MD Trajectories. In: Journal of the American Chemical Society, 2011, 133 (45), S. 18413–18419.
- David E. Shaw et al.: Millisecond-scale molecular dynamics simulations on Anton. Proceedings of the Conference on High Performance Computing Networking, Storage and Analysis. S. 1–11, 2009.
- David E. Shaw et al.: Atomic-Level Characterization of the Structural Dynamics of Proteins. In: Science, 2010, 330 (6002), S. 341–346.
- Ron O. Dror; Robert M. Dirks; J.P. Grossman; Huafeng Xu; David E. Shaw: Biomolecular Simulation: A Computational Microscope for Molecular Biology. In: Annual Review of Biophysics, 2012 41, S. 429–452.
- David E. Shaw, Martin M. Deneroff, Ron O. Dror, Jeffrey S. Kuskin, Richard H. Larson, John K. Salmon, Cliff Young, Brannon Batson, Kevin J. Bowers, Jack C. Chao;, Michael P. Eastwood, Joseph Gagliardo, J. P. Grossman, C. Richard Ho, Douglas J. Ierardi et al.: Anton, A Special-Purpose Machine for Molecular Dynamics Simulation. In: Communications of the ACM, 2008, 51 (7), S. 91–97,.