Ribosom
Ribosomen sind die makromolekularen Komplexe in Zellen, an denen Proteine hergestellt werden. Hierbei wird die Nukleotidsequenz (Basensequenz) eines Messenger-Ribonukleinsäure-Einzelstrangs (mRNA) in die Aminosäurensequenz der Polypeptidkette eines Proteins übersetzt. Diese Umwandlung der in der RNA gespeicherten Information in eine Abfolge von verknüpften Aminosäuren heißt Translation (lateinisch für Übersetzung) und ist in allen Lebewesen ein zentraler Bestandteil der Proteinbiosynthese.[1] Die dabei wirksame Übersetzungsregel wird als Genetischer Code bezeichnet. In der Zelle geschieht die Translation, nachdem die in der Abfolge von Basenpaaren des DNA-Doppelstrangs niedergelegte Erbinformation eines Gens in die Sequenz des mRNA-Einzelstrangs umgeschrieben wurde.
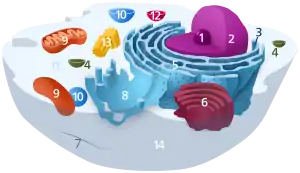
1. Nucleolus (Kernkörperchen)
2. Zellkern (Nukleus)
3. Ribosomen
4. Vesikel
5. Raues (Granuläres) ER (Ergastoplasma)
6. Golgi-Apparat
7. Cytoskelett
8. Glattes (Agranuläres) ER
9. Mitochondrien
10. Lysosom
11. Cytoplasma (mit Cytosol und Cytoskelett)
12. Peroxisomen
13. Zentriolen
14. Zellmembran
Ribosomen sind aus Ribosomaler RNA, englisch Ribonucleic acid (rRNA) und Proteinen (rProtein, auch r-Protein[2][3][4]) aufgebaut und finden sich im Cytoplasma, sowie in Zellorganellen, die aufgrund ihres endosymbiotischen Ursprungs eine eigene Maschinerie zur Proteinbiosynthese besitzen, wie den Mitochondrien und Chloroplasten. Auch bei einigen Viren sind in den Viruspartikeln Ribosomen enthalten, die von den Wirtszellen stammen, ohne dass sie in diesen eine Funktion erfüllen; beispielsweise sorgen diese bei den Arenaviren für das „sandfarbige“ Aussehen (lateinisch arena ‚Sand‘).
Aufbau und Arten
Ribosomen sind granuläre Partikel mit einem Durchmesser von etwa 20–25 nm. Sie bestehen zu etwa zwei Dritteln aus RNA (rRNA) und einem Drittel aus ribosomalen Proteinen. Sie setzen sich in allen Organismen aus zwei unterschiedlich großen und funktionell verschiedenen Untereinheiten zusammen. Die Masse der Ribosomen wird durch ihr Sedimentationsverhalten charakterisiert, das in Svedberg-Einheiten (S) angegeben wird. Während der Translation assemblieren sie zu einem funktionalen Komplex, wobei die große Untereinheit in der Proteinbiosynthese die Aminosäuren zur Kette verknüpft (Peptidyltransferaseaktivität), und die kleine Untereinheit für die mRNA-Erkennung verantwortlich ist. Beide Untereinheiten bestehen aus Proteinen und rRNA, wobei die Proteine für den Zusammenhalt und die richtige Positionierung zuständig sind, die eigentlichen Reaktionen hingegen werden durch die rRNAs vorgenommen. Beide Untereinheiten werden bei Eukaryonten in den Nucleoli innerhalb der Zellkerne gebildet und werden dann durch die Kernporen ins Cytoplasma geleitet.
Prokaryotische Ribosomen
| Ribosom | Untereinheit | rRNAs | r-Proteine |
|---|---|---|---|
| 70S | 50S | 23S (2904 nt) | 31 |
| 5S (120 nt) | |||
| 30S | 16S (1542 nt) | 21 |
Die Anzahl von Ribosomen je Zelle liegt bei Prokaryoten in der Größenordnung von 10.000, beispielsweise besitzt ein einzelnes E.-coli-Bakterium etwa 20.000 Ribosomen.[5] Die Ribosomen haben einen Sedimentationskoeffizienten von 70S und eine molare Masse von etwa 2,5 MDa. Bei Magnesiumkonzentrationen unter 1 mmol/l zerfällt das 70S-Ribosom zu einer 50S- und einer kleineren 30S-Untereinheit. Die 30S-Untereinheit (0,9 MDa) ist aus 21 verschiedenen ribosomalen Proteinen und einer 16S ribosomalen RNA (16S rRNA) zusammengesetzt. In der 50S-Untereinheit (1,6 MDa) finden sich 31 verschiedene Proteine und zwei rRNAs (23S und 5S rRNA).
Die Proteine der kleinen Untereinheit werden mit „S“ (englisch small ‚klein‘), die der großen Untereinheit mit „L“ (engl. large ‚groß‘) gekennzeichnet. Ihre Aminosäuresequenzen besitzen keine besonderen Gemeinsamkeiten, sind aber reich an positiv geladenen Aminosäuren wie L-Lysin oder L-Arginin. Dies erlaubt eine bessere Interaktion mit den negativ geladenen rRNAs. Das größte bakterielle, ribosomale Protein ist S1 mit 61,2 kDa und 557 Aminosäuren, das kleinste ist L34 mit 5,4 kDa und 34 Aminosäuren.
Eukaryotische Ribosomen
In eukaryotischen Zellen befinden sich die Ribosomen im Cytoplasma (nicht im Karyoplasma des Zellkerns). Daneben kommen spezielle Ribosomen auch in einigen Organellen vor, wenn diese über eigene DNA verfügen:
- in den Mitochondrien (oder ersatzweise in Hydrogenosomen, wenn mit DNA), sowie
- in (fast allen) Chloroplasten und anderen Plastiden, falls – wie bei den Pflanzen – vorhanden.
Cytosolische Ribosomen
| Ribosom | Untereinheit | rRNAs | r-Proteine |
|---|---|---|---|
| 80S | 60S | 28S (4718 nt) | 49 |
| 5,8S (160 nt) | |||
| 5S (120 nt) | |||
| 40S | 18S (1874 nt) | 33 |
Man schätzt die Zahl cytosolischer Ribosomen je Zelle auf zwischen 105 und über 107, womit eukaryotische Zellen weitaus mehr Ribosomen besitzen als prokaryotische. Die Anzahl ist vom Zelltyp abhängig und zwar von der Proteinsyntheserate der Zelle. So ist die Ribosomenanzahl in Leberzellen besonders hoch. Außerdem sind die eukaryotischen Ribosomen des Cytosols auch größer, sie haben einen Durchmesser von etwa 25 nm.[7] Diese haben eine molare Masse von etwa 4,2 MDa, der Sedimentationskoeffizient beträgt 80S. Bei der großen Untereinheit liegt er bei 60S (2,8 MDa) und bei seiner kleinen Untereinheit bei 40S (1,4 MDa).[8] Die kleine Untereinheit besteht in Säugern aus 33 Proteinen und einer rRNA (18S rRNA), die große Untereinheit aus 49 Proteinen und drei rRNAs (28S, 5,8S und 5S). Cytosolische Ribosomen höherer Eukaryoten sind komplexer als die niederer Eukaryoten. So ist die 28S rRNA in Backhefe 3.392 Nukleotide lang, in Säugern wie der Ratte dagegen 4.718 Nukleotide. Auch die 18S rRNA ist in Backhefe kleiner als in der Ratte (1799 gegenüber 1.874 Nukleotide).
Die eigentliche katalytische Funktion besitzt die rRNA, wohingegen die Proteine eher am Rand des Ribosoms sitzen. In Eukaryoten gibt es außer den freien cytoplasmatischen Ribosomen auch membrangebundene Ribosomen, die an die Membran des rauen Endoplasmatischen Retikulums (ER) gebunden sind (s. u.). Die Bildung der ribosomalen Untereinheiten findet im Nucleolus statt. Zellen mit hoher Proteinsyntheserate haben deshalb besonders gut ausgeprägte Nucleoli. Freie und membrangebundene Ribosomen haben die gleiche Struktur und können zwischen den Funktionen wechseln.
Die Schreibweise eukaryotischer ribosomaler Proteine ist nicht ganz einheitlich. In Backhefe werden Proteine der großen Untereinheit mit „Rpl“, die der kleinen mit „Rps“ bezeichnet. Bei den entsprechenden Proteinen der Säuger verwendet man auch die Großschreibung RPL bzw. RPS.
Mitoribosomen und Plastoribosomen
Die Ribosomen aus Mitochondrien und Chloroplasten sind den prokaryotischen Ribosomen ähnlich, was die Endosymbiontenhypothese stützt. Die mitochondrialen Ribosomen des Menschen und anderen Säugern bestehen aus vielen Proteinen, von denen 21 nur in Mitochondrien vorkommen, und erzeugen nur mitochondriale Membranproteine.[9][10]
80S-Ribosomen in komplexen Plastiden
Im Gegensatz dazu können die komplexen Plastiden etwa der Chlorarachniophyten mit einem zusätzlichen Zellkern (Nucleomorph) eigene eukaryotische 80S-Ribosomen enthalten.[11] Die komplexen Plastiden werden als Resultat einer sekundären Endosymbiose gedeutet (sekundäre Plastiden).
Freie und membrangebundene Ribosomen
Ribosomen können in eukaryotischen Zellen nach dem Ort ihrer Synthesetätigkeit unterschieden werden. Freie Ribosomen liegen im Cytoplasma verstreut und erzeugen Proteine, die ihre Aufgabe meistens ebenfalls im Zellplasma wahrnehmen. Membrangebundene Ribosomen sind mit der Membran des Endoplasmatischen Retikulums verbunden. Die dort synthetisierten Proteine werden mittels des cotranslationalen Proteintransportes in das Lumen des Endoplasmatischen Reticulums geleitet. Membrangebundene Ribosomen findet man gehäuft in sekretbildenden Zellen wie z. B. in der Bauchspeicheldrüse.
Funktionsweise
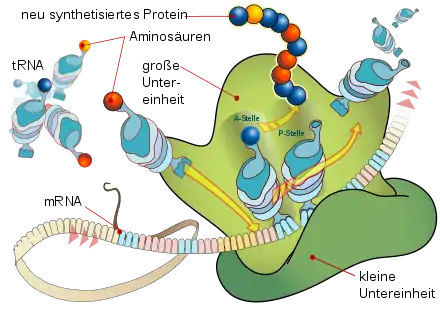
Die Funktionsweise des Ribosoms während der Translation kann durch das Dreistellenmodell charakterisiert werden. Demnach besitzt das Ribosom drei tRNA-Bindungsstellen, die A-(Aminoacyl-), P-(Peptidyl-) und E-(Exit-)Stelle. Während des Elongationszyklus oszilliert das Ribosom zwischen zwei Zuständen, dem prä- und dem post-translationalen Zustand, wobei zwei der drei tRNA-Bindungsstellen mit einer tRNA besetzt sind. Im prätranslationalen Zustand sind die A- und P-Stelle besetzt, wobei die P-Stelle die tRNA mit der Polypeptidkette trägt und die A-Stelle von der neu hinzugekommenen Aminoacyl-tRNA besetzt ist. Im Ribosom wird nun die Polypeptidkette mittels Peptidyltransferase von der P-Stellen-tRNA auf die A-Stellen-tRNA übertragen. Danach wechselt das Ribosom in den posttranslationalen Zustand und wandert um drei Basen auf der mRNA weiter, wodurch die vorherige A-Stellen-tRNA zur P-Stellen-tRNA wird und die nun leere ehemalige P-Stellen-tRNA über die E-Stelle (Exit) aus dem Ribosom geschleust wird. Dabei ist eine Translokase (EF-G) beteiligt.
Die beiden Hauptzustände des Ribosoms (prä- und posttranslational) sind durch eine hohe Aktivierungsenergie-Barriere voneinander getrennt. Die zentrale Rolle der beiden Elongationsfaktoren besteht darin, diese Energiebarriere zu erniedrigen und so das Ribosom in den jeweils anderen Zustand zu versetzen.
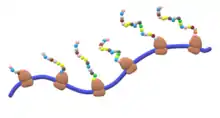
Manchmal formieren sich mehrere prokaryotische Ribosomen an demselben mRNA-Molekül perlschnurartig zu einem Polysom.
Nachdem ein Peptid im Ribosom verknüpft wurde, durchwandert es einen ribosomalen Tunnel. Dieser besteht größtenteils aus rRNA und tritt aus der großen ribosomalen Untereinheit aus. Er hat eine Länge von ca. 100 Å (10 nm) und einen durchschnittlichen Durchmesser von 15 Å (1,5 nm). An dessen engster Stelle wird der Kanal durch zwei konservierte ribosomale Proteine begrenzt, L4e und L22.
Ribophagie
Der Abbau von Ribosomen ist noch nicht vollständig verstanden. Er wird in der Regel unter Nährstoffmangel eingeleitet. Für Bakterien wie E. coli wurde vorgeschlagen, dass intakte 70S-Ribosomen zunächst in beide Untereinheiten zerfallen.[12] Unter Mangelbedingungen wird die Translation in der Zelle heruntergefahren, so dass viele Ribosomen inaktiv sind. Die beiden Untereinheiten sind wesentlich empfindlicher gegenüber Ribonukleasen (RNasen) als ein intaktes Ribosom, da sie eine größere Angriffsfläche bieten. Danach könnten auch Exonukleasen die ribosomale RNA weiter abbauen.
Für Backhefe, einen Eukaryot, wurde ein mit „Ribophagie“ bezeichneter Autophagieweg vorgeschlagen.[13] Dieser lehnt an die Begriffe Mitophagie (Abbau von Mitochondrien)[14], Pexophagie (Abbau von Peroxisomen)[15] und Reticulophagie (Abbau des Endoplasmatischen Retikulums)[16] an. Unter Nährstoffmangel baut Hefe Ribosomen auf einem Weg ab, der ähnlich wie bei Prokaryoten beginnt. Zunächst werden die beiden Untereinheiten getrennt. Eine Ubiquitinligase entfernt dann Ubiquitin an der 60S-Untereinheit, welche dann in einem Vesikel zur Vakuole transportiert wird. Dies erscheint zunächst paradox, da Ubiquitin ein allgemeines Abbausignal für die meisten Proteine ist. Von den Autoren wurde vorgeschlagen, dass eine Ubiquitinligase die 60S-Untereinheit zunächst für den Abbauweg markiert, der Prozess aber erst durch die Ubiquitinprotease endgültig ablaufen kann.
Strukturaufklärung
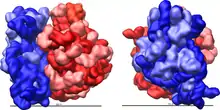
Ribosomen wurden durch den Forscher Albert Claude Mitte des 20. Jahrhunderts entdeckt.[17][18] 1940 hatte er mit Hilfe der Dunkelfeldmikroskopie RNA-enthaltende Granula aus dem Cytosol tierischer Zellen identifiziert, die kleiner als Mitochondrien waren.[19] Er bezeichnete diese als „Mikrosomen“, spätere Analysen zeigten, dass sie Komplexe aus Phospholipiden und Ribonukleinproteinen waren. Heutzutage werden Fragmente des ERs als Mikrosomen bezeichnet. Durch Fortschritte in der Elektronenmikroskopie gelang es 1955 George Emil Palade, jene „Mikrosomen“ eindeutig als Bestandteile einer Zelle und nicht bloß als Artefakte von Zelltrümmern zu identifizieren.[20] Es gab immer mehr Hinweise darauf, dass diese Ribonukleinproteinpartikel etwas mit der Translation zu tun hatten. 1959, wurde auch der Beweis in E. coli erbracht, dass Ribosomen für die Biosynthese von Polypeptiden notwendig sind.[21]
1958 griff Richard B. Roberts in einem Symposium den Vorschlag auf, den Namen „Mikrosom“ bzw. „mikrosome Partikel“ auf den besserklingenden und einfachen Namen – so Roberts – „Ribosom“ zu ändern.[22] Diese Abkürzung verweist auf die Art der Partikel, Komplexe aus RNA und Proteinen (Ribonukleopartikel). Die Bezeichnung „Ribosom“ konnte sich durchsetzen und wird im heutigen Sprachgebrauch ausschließlich verwendet.
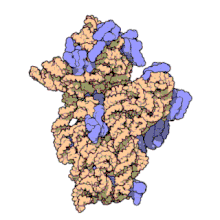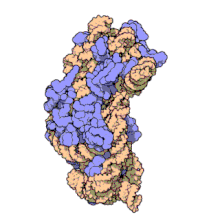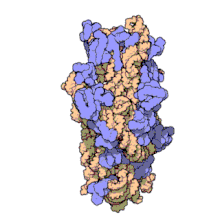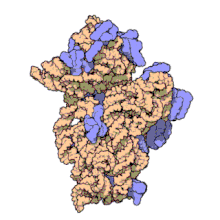
Wegen ihrer Kleinheit konnten erst in jüngerer Zeit hochauflösende Strukturen von Ribosomen gewonnen werden, wenngleich der grobe molekulare Aufbau seit den 1970er-Jahren bekannt ist. Einige Details ribosomaler Proteine konnten mittels Affinitätsmarkierung und chemisches Quervernetzen (crosslinking) aufgeklärt werden.[23] Ende 2000 wurde zum ersten Mal die 50S-Untereinheit des Archaeon Haloarcula marismortui in einer Auflösung von 2,4 Å aufgeklärt.[24] In dieser Auflösung kann man einzelne Moleküle auflösen. Zeitgleich wurde auch die Strukturen der kleinen ribosomalen Untereinheit aus Thermus thermophilus in einer atomaren Auflösung von 3 Å publiziert.[25][26] Da zu diesem Zeitpunkt keine Strukturdaten des kompletten Ribosoms vorlagen, wurden die vorhandenen Daten genutzt, um das prokaryotische Ribosom zu rekonstruieren.[27]
Während die A und P-Stelle schon länger bekannt waren, wurde die E-Stelle erst 1981 entdeckt (Knud Nierhaus und Kollegen, Alpha-Epsilon-Theorie der Bindung der tRNA im Ribosom).
2005 wurden zum ersten Mal die kristallographischen Strukturdaten eines intakten Ribosom aus E. coli in einer Auflösung von 3,5 Å vorgestellt.[28] Nahezu zeitgleich konnte eine andere Forschergruppe eine Struktur präsentieren, die mit Hilfe der Cryoelektronenmikroskopie gewonnen wurde.[29] Die Auflösung war mit über 10 Å vergleichsweise gering, zeigte aber eine Momentaufnahme der Translation am Translokon.
Später wurden immer mehr Strukturdaten von (prokaryotischen) Ribosomen veröffentlicht, die gerade mRNAs oder tRNAs gebunden hatten und damit einen besseren Einblick auf die Prozesse der Translation gewährten.[30][31]
Für das eukaryotische Ribosom (80S) gibt es noch keine vergleichbaren Strukturdaten. Eine dreidimensionale Rekonstruktion ist indes aus den gesammelten Daten der Kryoelektronenmikroskopie, der Röntgenkristallographie einzelner ribosomalen Komponenten sowie Homologievergleiche mit prokaryontischen Ribosomen möglich.[32][33][34][35]
Die kleine Untereinheit (SSU) zeigt beim Vergleich von Bakterien, Archaaen und Eukaryoten (Cytosol) charakteristische Unterschiede: die SSU der Bakterien besteht aus einem sog. Körper (englisch body), einer Platte (en. platform) und einem Kopf (en. head), bei den Archaeen kommt noch ein Schnabel (en. bill) genannte Ausstülpung am Kopf hinzu, bei Eukaryoten (Cytosol) darüber hinaus noch zweibenachbarte Lappen (en. lobes) am Körper. Insgesamt wird die Struktur von Bakterien über Archaeen bis hin zu Eukaryoten immer komplexer.[36][37]
Thomas A. Steitz, Ada Yonath und Venkatraman Ramakrishnan erhielten für ihre Arbeit an der Strukturaufklärung 2009 den Nobelpreis für Chemie.[38]
Ursprung
Der Ursprung der Ribosomen wird in der RNA-Welt vermutet, in der ein selbstreplizierender Komplex erst später die Fähigkeit zur Proteinsynthese entwickelte, als dafür ausreichend Aminosäuren zur Verfügung standen.[39] Die katalytischen Fähigkeiten der RNA (Ribozym) sind ein zentraler Bestandteil der RNA-Welt-Hypothese. Untersuchungen legen nahe, dass diese Ribosomen-Vorläufer, die ausschließlich aus rRNA aufgebaut waren, die Fähigkeit zur Bildung von Peptidbindungen entwickelt haben könnten.[40][41][42] Darüber hinaus gibt es eine starke Evidenz, dass ursprüngliche Ribosomen selbstreplizierende Komplexe waren, in denen die rRNA informationelle, strukturelle und katalytische Zwecke hatte, da sie tRNA und Proteine für die ribosomale Selbstreplikation codiert haben könnte.[43] Die hypothetischen DNA-freien zellulären Organismen, die mit solcher selbstreplizierender RNA ausgestattet waren, nennt man Ribozyten.[44]
Als sich in der RNA-Welt unter noch präbiotischen Bedingungen allmählich Aminosäuren anreicherten,[45][46] könnte ihre Wechselwirkung mit der katalytischen RNA sowohl deren Wirkungsbereich als auch ihre Effizienz erhöht haben.[39] Der selektive Druck, Proteine in die selbstreplizierenden Mechanismen der Ribosomen einzubauen, könnte die treibende Kraft für die Evolution der Ribosomen von einer ursprünglich selbstreplizierenden Maschine in ihre heutige Form als Translationsmaschine gewesen sein, da dies die Kapazität der Selbstreplikation erhöht hätte.[43]
Entstehung der DNA
Die Speicherung des Genoms in Form der DNA-Doppelhelix erscheint als eine erst spätere Zutat. DNA-Replikation und Transkription sind bei Bakterien auf der einen, und Archaeen und Eukaryoten (Neomura) auf der anderen Seite so stark verschieden, dass die Annahme eines gemeinsamen Ursprungs (Homologie) unwahrscheinlich erscheint. Stattdessen könnten diese beiden Gruppen – ausgehend von primitiven zellulären Organismen mit Ribosomen – die Fähigkeit zur Speicherung der Erbinformation in DNA jeweils für sich erworben haben, mutmaßlich mit Hilfe von DNA-Viren.[47] Nach dieser Annahme hatten sich zuvor die DNA-Viren aus den ursprünglicheren RNA-Viren entwickelt um ihr Genom besser vor Attacken durch die Wirtszellen zu schützen, was das Ende der reinen RNA-Welt bedeutete.[48][49]
Literatur
- Donald Voet und Judith G. Voet: Biochemie. Wiley-VCH 1994; ISBN 3-527-29249-7; S. 917ff.
- Alexander S. Spirin: Ribosomes (Cellular Organelles). Springer, Berlin 1999; ISBN 0-306-46145-5
- Reginald Garrett und Charles M. Grisham: Biochemistry. (International Student Edition). Cengage Learning Services; 4. Auflage 2009; ISBN 978-0-495-11464-2
- S. Klinge et al. (2012): Atomic structures of the eukaryotic ribosome. In: Trends Biochem Sci. 37(5); 189–198; PMID 22436288; doi:10.1016/j.tibs.2012.02.007
- D. N. Wilson und J. H. Doudna Cate (2012): The structure and function of the eukaryotic ribosome. In: Cold Spring Harb Perspect Biol. 4(5); 1–17; PMID 22550233; PDF (freier Volltextzugriff, engl.)
- S. Melnikov et al. (2012): One core, two shells: bacterial and eukaryotic ribosomes. In: Nat Struct Mol Biol. 19(6); 560–567; PMID 22664983; doi:10.1038/nsmb.2313
Weblinks
- Proteopedia: Ribosome
- Ribosomal Database (Datenbank über ribosomale Proteine)
- Video: Erklärung der Ribosomstruktur und ihre Entdeckung
- Video: Proteinbiosynthese am Ribosom (Kenyon College, englisch) (MP4; 39,8 MB)
Einzelnachweise
- Hans G. Kloepfer: Struktur und Funktion von Ribosomen. In: Chemie in unserer Zeit. Band 7, Nr. 2, 1973, S. 49–58, doi:10.1002/ciuz.19730070204.
- Salini Konikkat: Dynamic Remodeling Events Drive the Removal of the ITS2 Spacer Sequence During Assembly of 60S Ribosomal Subunits in S. cerevisiae. Carnegie Mellon University Dissertations, Feb. 2016.
- Elmar W. Weiler, Lutz Nover: Allgemeine und molekulare Botanik. Georg Thieme Verlag, Stuttgart 2008, ISBN 978-3-13-152791-2, S. 532 (eingeschränkte Vorschau in der Google-Buchsuche).
- Jesus de la Cruz, Katrin Karbstein, John L. Woolford, Jr.: Functions of Ribosomal Proteins in Assembly of Eukaryotic Ribosomes In Vivo. In: Annual review of biochemistry. Band 84, 2015, S. 93–129, doi:10.1146/annurev-biochem-060614-033917, PMID 25706898, PMC 4772166 (freier Volltext).
- Reginald Garrett und Charles M. Grisham: Biochemistry. (International Student Edition). Cengage Learning Services; 4. Auflage 2009; ISBN 978-0-495-11464-2; S. 962.
- Reginald Garrett und Charles M. Grisham: Biochemistry. (International Student Edition). Cengage Learning Services; 4. Auflage 2009; ISBN 978-0-495-11464-2; S. 965.
- Helmut Plattner und Joachim Hentschel: Zellbiologie. Thieme, Stuttgart; 3., neu bearb. Auflage 2006; ISBN 3-13-106513-3; S. 181.
- Reginald Garrett und Charles M. Grisham: Biochemistry. (International Student Edition). Cengage Learning Services; 4. Auflage 2009; ISBN 978-0-495-11464-2; S. 964.
- A. Brown, A. Amunts, X. C. Bai, Y. Sugimoto, P. C. Edwards, G. Murshudov, S. H. Scheres, V. Ramakrishnan: Structure of the large ribosomal subunit from human mitochondria. In: Science. Band 346, Nummer 6210, November 2014, S. 718–722, doi:10.1126/science.1258026. PMID 25278503.
- B.J. Greber, D. Boehringer, M. Leibundgut, P. Bieri, A. Leitner, N. Schmitz, R. Aebersold, N. Ban: The complete structure of the large subunit of the mammalian mitochondrial ribosome. In: Nature. Band 515, Nummer 7526, November 2014, S. 283–286, doi:10.1038/nature13895. PMID 25271403.
- Shigekatsu Suzuki, Shu Shirato, Yoshihisa Hirakawa, Ken-Ichiro Ishida: Nucleomorph Genome Sequences of Two Chlorarachniophytes, Amorphochlora amoebiformis and Lotharella vacuolata. In: Genome Biology and Evolution. Band 7, Nr. 6, 2015, ISSN 1759-6653, S. 1533–1545, doi:10.1093/gbe/evv096, PMID 26002880, PMC 4494063 (freier Volltext).
- Zundel, MA. et al. (2009): Initiation of ribosome degradation during starvation in Escherichia coli. In: RNA 15(5); 977–783; PMID 19324965; PDF (freier Volltextzugriff, englisch).
- Kraft, C. et al. (2008): Mature ribosomes are selectively degraded upon starvation by an autophagy pathway requiring the Ubp3p/Bre5p ubiquitin protease. In: Nat Cell Biol. 10(5); 602–610; PMID 18391941; doi:10.1038/ncb1723
- Kim, I. et al. (2007): Selective degradation of mitochondria by mitophagy. In: Arch Biochem Biophys. 462(2); 245–253; PMID 17475204; PMC 2756107 (freier Volltext)
- Dunn, WA. Jr. et al. (2005): Pexophagy: the selective autophagy of peroxisomes. In: Autophagy 1(2); 75–83; PMID 16874024; PDF (freier Volltextzugriff, englisch).
- Klionsky, DJ. et al. (2007): How shall I eat thee? In: Autophagy 3(5); 413–416; PMID 17568180; PDF (freier Volltextzugriff, englisch).
- Donald Voet und Judith G. Voet: Biochemie. Wiley-VCH 1994; ISBN 3-527-29249-7; S. 917
- Alexander S. Spirin: Ribosomes (Cellular Organelles). Springer, Berlin 1999; ISBN 0-306-46145-5, S. 47.
- A. Claude: Particulate components of normal and tumor cells. In: Science, Band 91, Nr. 2351, 1940, S. 77–78: PMID 17783332; doi:10.1126/science.91.2351.77
- G. E. Palade: A small particulate component of the cytoplasm. In: J Biophys Biochem Cytol. Band 1, Nr. 1, 1955, S. 59–68; PMID 14381428; PMC 2223592 (freier Volltext)
- McQuillen, K., Roberts, RB. und Britten, RJ. (1959): SYNTHESIS OF NASCENT PROTEIN BY RIBOSOMES IN ESCHERICHIA COLI. In: Proc Natl Acad Sci USA, Band 45, Nr. 9, S. 1437–1447; PMID 16590524; PMC 222733 (freier Volltext)
- Richard B. Roberts: Microsomal Particles and Protein Synthesis. London, Pergamon Press 1958; Volltextzugriff (englisch).
- Czernilofsky, AP., Kurland, CG. und Stöffler, G. (1975): 30S ribosomal proteins associated with the 3'-terminus of 16S RNA. In: FEBS Lett. 58(1); 281–284; PMID 1225593.
- Ban, N. et al. (2000). The complete atomic structure of the large ribosomal subunit at 2.4 ångström resolution. In: Science 289 (5481); 905–920; PMID 10937989; doi:10.1126/science.289.5481.905
- Schluenzen, F. et al. (2000): Structure of functionally activated small ribosomal subunit at 3.3 angstroms resolution. In: Cell 102(5); 615–623; PMID 11007480
- Wimberly, BT., et al. (2000): Structure of the 30S ribosomal subunit. In: Nature. 407(6802); 327–339; PMID 11014182; doi:10.1038/35030006
- Yusupov, MM. et al. (2001): Crystal structure of the ribosome at 5.5 ångström resolution. In: Science. 292(5518); 883–896. PMID 11283358; doi:10.1126/science.1060089
- Schuwirth, B. S. et al. (2005): Structures of the bacterial ribosome at 3.5 A resolution. In: Science 310(5749): 827–834; PMID 16272117; doi:10.1126/science.1117230.
- Mitra, K. et al. (2005): Structure of the E. coli protein-conducting channel bound to a translating ribosome. In: Nature. 438(7066); 318–324; PMID 16292303; PMC 1351281 (freier Volltext).
- Selmer, M. et al (2006): Structure of the 70S ribosome complexed with mRNA and tRNA. In: Science 313(5795): 1935–1942; PMID 16959973.
- Korostelev, A., Trakhanov, S., Laurberg, M. und Noller, H. F. (2006): Crystal structure of a 70S ribosome-tRNA complex reveals functional interactions and rearrangements. In: Cell 126(6); 1065–1077; PMID 16962654.
- Gilbert, R. J. et al. (2004): Three-dimensional structures of translating ribosomes by Cryo-EM. In: Mol Cell 14(1): 57–66; PMID 15068803.
- Stark, H. (2002): Three-dimensional electron cryomicroscopy of ribosomes. In: Curr Protein Pept Sci 3(1): 79–91; PMID 12370013.
- Spahn, C. M., et al. (2001): Structure of the 80S ribosome from Saccharomyces cerevisiae--tRNA-ribosome and subunit-subunit interactions. In: Cell 107(3): 373–386; PMID 11701127.
- Heena Khatter, Alexander G. Myasnikov, S. Kundhavai Natchiar, Bruno P. Klaholz: Structure of the human 80S ribosome. In: Nature. 520, 2015, S. 640, doi:10.1038/nature14427.
- Lynn Margulis, Karlene V. Schwartz: Die fünf Reiche der Organismen — ein Leitfaden. Spektrum der Wissenschaft Verlagsgesellschaft, Heidelberg, 1989, ISBN 3-89330-694-3. Hier Abb. S. 37. Originaltitel der en. Ausgabe: Five Kingdoms. Freeman & Co., New York/Oxford 182/1988. Hier Fig. B-3: Ribosome Morphology.
- James A. Lake: Ribosomal Evolution: The Structural Basis for Protein Synthesis in Archaebacteria, Eubacteria and Eukaryotes. In: Prog Nucleic Acid Res Mol Biol, Band 30, 1983, S. 163–194; doi:10.1016/S0079-6603(08)60686-8, PMID 6420842. Hier S. 318 f.
- Nobelpreis für Chemie 2009.
- H. F. Noller: Evolution of protein synthesis from an RNA world. In: Cold Spring Harbor Perspectives in Biology. 4, 2012, S. 1–U20. doi:10.1101/cshperspect.a003681. PMC 3312679 (freier Volltext).
- E. R. Dabbs: Structure, Function, and Genetics of Ribosomes. Hrsg.: B. Hardesty, G. Kramer. Springer, New York 1986, Kapitel 43: Mutant Studies on the Prokaryotic Ribosome, S. 733–748, doi:10.1007/978-1-4612-4884-2_43.
- H. F. Noller, V. Hoffarth, L. Zimniak: Unusual resistance of peptidyl transferase to protein extraction procedures. In: Science. Band 256, Nr. 5062, Juni 1992, S. 1416–1419, doi:10.1126/science.1604315, PMID 1604315.
- M. Nomura, S. Mizushima, M. Ozaki, P. Trau, C. V. Lowry: Structure and function of ribosomes and their molecular components. In: Cold Spring Harbor Symposium of Quantitative Biology. 34, 1969, S. 49–61. doi:10.1101/sqb.1969.034.01.009.
- M. Root-Bernstein, R. Root-Bernstein: The ribosome as a missing link in the evolution of life. In: Journal of Theoretical Biology. 367, 2015, S. 130–158. doi:10.1016/j.jtbi.2014.11.025. PMID 25500179.
- M. Yarus: Primordial genetics: phenotype of the ribocyte. In: Annu. Rev. Genet.. 36, 2002, S. 125–51. doi:10.1146/annurev.genet.36.031902.105056. PMID 12429689.
- G. Caetano-Anolles, M. J. Seufferheld: The coevolutionary roots of biochemistry and cellular organization challenge the RNA world paradigm. In: Journal of Molecular Microbiology and Biotechnology. 23, 2013, S. 152–177. doi:10.1159/000346551.
- R. Saladino, G. Botta, S. Pino, G. Costanzo, E. Di Mauro: Genetics first or metabolism first? The formamide clue. In: Chemical Society Reviews. 41, 2012, S. 5526–5565. doi:10.1039/c2cs35066a.
- P. Forterre: Evolution – Die wahre Natur der Viren. In: Spektrum. August 2017, S. 37 (Online-Artikel vom 19. Juli 2017).
- P. Forterre: Three RNA cells for ribosomal lineages and three DNA viruses to replicate their genomes: a hypothesis for the origin of cellular domain. In: Proc. Natl. Acad. Sci. U.S.A.. 103, März 2006, S. 3669–74. doi:10.1073/pnas.0510333103. PMID 16505372. PMC 1450140 (freier Volltext).
- C. Zimmer: Did DNA come from viruses?. In: Science. 312, Nr. 5775, Mai 2006, S. 870–2. doi:10.1126/science.312.5775.870. PMID 16690855.