Ribonukleasen H
Ribonuklease H (von Ribonuklease Hybrid, synonym RNase H) ist eine Gruppe von Enzymen (Ribonukleasen), die RNA in DNA-RNA-Hybriden abbauen.
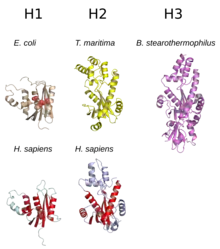
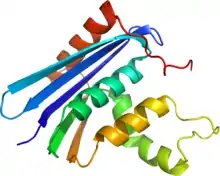
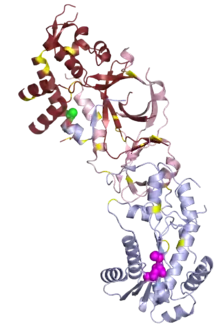
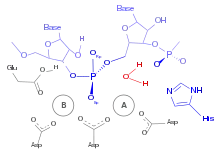
Eigenschaften
Sie werden in zwei Gruppen unterteilt, RNase HI (in Bakterien) bzw. RNase H1 (in Eukaryoten) und RNase HII (in Bakterien) bzw. RNase H2 (in Eukaryoten).[4] Während die RNase HI von Escherichia coli (155 AS, 17.600 Da) und die RNase H1 von Menschen (286 AS, 32.200 Da, und ohne mitochondriale Signalsequenz 260 AS, 29.400 Da) jeweils Monomere sind, ist die RNase HII von E. coli (198 AS, 21.500 Da) monomer und die RNase H2 von Menschen heterotrimer (RNase H2A mit 299 AS und 33.400 Da; RNase H2B mit 308 AS und 34.800 Da; RNase H2C mit 164 AS und 17.800 Da).[4] In wenigen Prokaryoten wird noch eine RNase HIII gebildet.[5] Eine RNase H wird auch vom HIV als Proteindomäne der reversen Transkriptase codiert, die der RNase H1 von Menschen in der Proteinstruktur und im Reaktionsmechanismus ähnelt[4] und notwendig für die Replikation ist.[6][7]
Mutationen in der menschlichen RNase H2 können zum Aicardi-Goutières-Syndrom führen.[4][8]
Funktion
Ribonukleasen des Typs H kommen in fast allen Lebewesen vor und sind sequenzunspezifische Endonukleasen, welche die Phosphorsäureester-Bindung von RNA in Doppelsträngen von DNA und RNA hydrolysieren, wodurch eine 3'-Hydroxygruppe und eine 5'-Phosphatgruppe entsteht. Die RNase H1 ist an der Replikation der mitochondrialen DNA beteiligt. RNase H1 und H2 sind am Abbau des R-Loop beteiligt.[4][9][10][11] In Prokaryoten und niederen Eukaryoten sind RNasen H nicht essentiell, in höheren Eukaryoten sind sie essentiell.[4] RNase H1 und H2 haben zwar unterschiedliche Substratvorlieben, aber überlappende Funktionen im Mitochondrium.
RNase H1
RNase H1 benötigt als Substrat mindestens vier Ribonukleotide und wird durch Desoxyribonukleotide im gleichen Strang gehemmt. Daher ist es unwahrscheinlich, dass RNase H1 am Abbau der RNA-Primer an den Okazaki-Fragmenten während der DNA-Replikation beteiligt ist.[4] Ein Gen-Knockout der RNase HI in E. coli erzeugt einen temperaturempfindlichen Phänotyp, ist aber nicht letal.[9] In Saccharomyces cerevisiae wird durch einen Gen-Knockout von RNase H1 die Reaktion auf Zellstress gestört und ist ebenfalls nicht letal.[12] In vielen Eukaryoten, einschließlich Säugetiere, besitzt RNase H1 eine mitochondriale Signalsequenz, wodurch es in der Zelle außerhalb des Mitochondriums mit der Signalsequenz und innerhalb des Mitochondriums ohne Signalsequenz vorkommt. Ein Gen-Knockout der RNase H1 in Mäusen ist letal während der Embryogenese aufgrund von gestörter Replikation der mitochondrialen DNA,[4][13][14] vermutlich aufgrund des gestörten Abbaus des R-loop.[11]
RNase H2
Die RNase H2 Untereinheit A ist homolog zur RNase HII, während die Untereinheiten B und C kein prokaryotisches Homolog haben und innerhalb der Eukaryoten vergleichsweise stark variieren.[15][16] Die Untereinheit B vermittelt Protein-Protein-Interaktionen zwischen RNase H2 und PCNA, wodurch RNase H2 an den Ort der DNA-Replikation lokalisiert wird.[17] Prokaryotische RNase HII besitzen im Vergleich zur RNase H2 eine niedrigere Reaktionsrate und werden durch Desoxyribonukleotide am 5'-Ende gehemmt.[4][18] Die RNase 2 ist zudem an der DNA-Reparatur (nucleotide excision repair) beteiligt, in dem sie falsch eingebaute Ribonukleotide aus DNA entfernt.[19][20][17] Im Zellkern von Säugetieren ist die RNase H2 die aktivere RNase H.[17]
RNase HIII
In wenigen Prokaryoten wird noch eine RNase III gebildet, die strukturell der RNase II ähnelt, aber im Reaktionsmechanismus der RNase I ähnelt.[5][9][21] Während RNase III in Prokaryoten eher vereinzelt vorkommt, ist sie in Archaeen etwas häufiger zu finden und meistens anstelle von RNase HI.[22]
Einzelnachweise
- E. coli PDB 2RN2, T. maritima PDB 303F, B. stearothermophilus PDB 2D0B, H. sapiens H1 PDB 2QK9, H. sapiens PDB 3P56.
- M. Figiel, H. Chon, S. M. Cerritelli, M. Cybulska, R. J. Crouch, M. Nowotny: The structural and biochemical characterization of human RNase H2 complex reveals the molecular basis for substrate recognition and Aicardi-Goutières syndrome defects. In: Journal of Biological Chemistry. Band 286, Nummer 12, März 2011, S. 10540–10550, doi:10.1074/jbc.M110.181974, PMID 21177858, PMC 3060507 (freier Volltext).
- Simon L. Dürr, Olga Bohuszewicz, Dénes Berta, Reynier Suardiaz, Pablo G. Jambrina: The Role of Conserved Residues in the DEDDh Motif: the Proton-Transfer Mechanism of HIV-1 RNase H. In: ACS Catalysis. 16. Juni 2021, S. 7915–7927, doi:10.1021/acscatal.1c01493.
- S. M. Cerritelli, R. J. Crouch: Ribonuclease H: the enzymes in eukaryotes. In: The FEBS journal. Band 276, Nummer 6, März 2009, S. 1494–1505, doi:10.1111/j.1742-4658.2009.06908.x, PMID 19228196, PMC 2746905 (freier Volltext).
- M. Figiel, M. Nowotny: Crystal structure of RNase H3-substrate complex reveals parallel evolution of RNA/DNA hybrid recognition. In: Nucleic acids research. Band 42, Nummer 14, August 2014, S. 9285–9294, doi:10.1093/nar/gku615, PMID 25016521, PMC 4132731 (freier Volltext).
- J. F. Davies, Z. Hostomska, Z. Hostomsky, S. R. Jordan, D. A. Matthews: Crystal structure of the ribonuclease H domain of HIV-1 reverse transcriptase. In: Science. Band 252, Nummer 5002, April 1991, S. 88–95, doi:10.1126/science.1707186, PMID 1707186.
- J. Hansen, T. Schulze, W. Mellert, K. Moelling: Identification and characterization of HIV-specific RNase H by monoclonal antibody. In: The EMBO Journal. Band 7, Nummer 1, Januar 1988, S. 239–243, PMID 2452083, PMC 454263 (freier Volltext).
- Y. J. Crow, A. Leitch, B. E. Hayward, A. Garner, R. Parmar, E. Griffith, M. Ali, C. Semple, J. Aicardi, R. Babul-Hirji, C. Baumann, P. Baxter, E. Bertini, K. E. Chandler, D. Chitayat, D. Cau, C. Déry, E. Fazzi, C. Goizet, M. D. King, J. Klepper, D. Lacombe, G. Lanzi, H. Lyall, M. L. Martínez-Frías, M. Mathieu, C. McKeown, A. Monier, Y. Oade, O. W. Quarrell, C. D. Rittey, R. C. Rogers, A. Sanchis, J. B. Stephenson, U. Tacke, M. Till, J. L. Tolmie, P. Tomlin, T. Voit, B. Weschke, C. G. Woods, P. Lebon, D. T. Bonthron, C. P. Ponting, A. P. Jackson: Mutations in genes encoding ribonuclease H2 subunits cause Aicardi-Goutières syndrome and mimic congenital viral brain infection. In: Nature genetics. Band 38, Nummer 8, August 2006, S. 910–916, doi:10.1038/ng1842, PMID 16845400.
- T. Tadokoro, S. Kanaya: Ribonuclease H: molecular diversities, substrate binding domains, and catalytic mechanism of the prokaryotic enzymes. In: The FEBS journal. Band 276, Nummer 6, März 2009, S. 1482–1493, doi:10.1111/j.1742-4658.2009.06907.x, PMID 19228197.
- Amon JD, Koshland D: RNase H enables efficient repair of R-loop induced DNA damage. In: eLife. 5, Dezember 2016, S. e20533. doi:10.7554/eLife.20533. PMID 27938663. PMC 5215079 (freier Volltext).
- Lima WF, Murray HM, Damle SS, Hart CE, Hung G, De Hoyos CL, Liang XH, Crooke ST: Viable RNaseH1 knockout mice show RNaseH1 is essential for R loop processing, mitochondrial and liver function. In: Nucleic Acids Research. 44, Nr. 11, Juni 2016, S. 5299–312. doi:10.1093/nar/gkw350. PMID 27131367. PMC 4914116 (freier Volltext).
- Arudchandran A, Cerritelli S, Narimatsu S, Itaya M, Shin DY, Shimada Y, Crouch RJ: The absence of ribonuclease H1 or H2 alters the sensitivity of Saccharomyces cerevisiae to hydroxyurea, caffeine and ethyl methanesulphonate: implications for roles of RNases H in DNA replication and repair. In: Genes to Cells. 5, Nr. 10, 2000, S. 789–802. doi:10.1046/j.1365-2443.2000.00373.x. PMID 11029655.
- Cerritelli SM, Frolova EG, Feng C, Grinberg A, Love PE, Crouch RJ: Failure to produce mitochondrial DNA results in embryonic lethality in Rnaseh1 null mice. In: Molecular Cell. 11, Nr. 3, 2003, S. 807–15. doi:10.1016/s1097-2765(03)00088-1. PMID 12667461.
- Reyes A, Melchionda L, Nasca A, Carrara F, Lamantea E, Zanolini A, Lamperti C, Fang M, Zhang J, Ronchi D, Bonato S, Fagiolari G, Moggio M, Ghezzi D, Zeviani M: RNASEH1 Mutations Impair mtDNA Replication and Cause Adult-Onset Mitochondrial Encephalomyopathy. In: American Journal of Human Genetics. 97, Nr. 1, Juli 2015, S. 186–93. doi:10.1016/j.ajhg.2015.05.013. PMID 26094573. PMC 4572567 (freier Volltext).
- Thomas Hollis, Nadine M. Shaban: Allen W. Nicholson (Hrsg.): Ribonucleases (= Nucleic Acids and Molecular Biology). Springer Berlin Heidelberg, 1. Januar 2011, ISBN 9783642210778, S. 299–317, doi:10.1007/978-3-642-21078-5_12.
- Chon H, Vassilev A, DePamphilis ML, Zhao Y, Zhang J, Burgers PM, Crouch RJ, Cerritelli SM: Contributions of the two accessory subunits, RNASEH2B and RNASEH2C, to the activity and properties of the human RNase H2 complex. In: Nucleic Acids Research. 37, Nr. 1, Januar 2009, S. 96–110. doi:10.1093/nar/gkn913. PMID 19015152. PMC 2615623 (freier Volltext).
- Reijns MA, Jackson AP: Ribonuclease H2 in health and disease. In: Biochemical Society Transactions. 42, Nr. 4, August 2014, S. 717–25. doi:10.1042/BST20140079. PMID 25109948.
- Chon H, Vassilev A, DePamphilis ML, Zhao Y, Zhang J, Burgers PM, Crouch RJ, Cerritelli SM: Contributions of the two accessory subunits, RNASEH2B and RNASEH2C, to the activity and properties of the human RNase H2 complex. In: Nucleic Acids Research. 37, Nr. 1, Januar 2009, S. 96–110. doi:10.1093/nar/gkn913. PMID 19015152. PMC 2615623 (freier Volltext).
- Wahba L, Amon JD, Koshland D, Vuica-Ross M: RNase H and multiple RNA biogenesis factors cooperate to prevent RNA:DNA hybrids from generating genome instability. In: Molecular Cell. 44, Nr. 6, Dezember 2011, S. 978–88. doi:10.1016/j.molcel.2011.10.017. PMID 22195970. PMC 3271842 (freier Volltext).
- Kim N, Huang SN, Williams JS, Li YC, Clark AB, Cho JE, Kunkel TA, Pommier Y, Jinks-Robertson S: Mutagenic processing of ribonucleotides in DNA by yeast topoisomerase I. In: Science. 332, Nr. 6037, Juni 2011, S. 1561–4. doi:10.1126/science.1205016. PMID 21700875. PMC 3380281 (freier Volltext).
- Ohtani N, Haruki M, Morikawa M, Crouch RJ, Itaya M, Kanaya S: Identification of the genes encoding Mn2+-dependent RNase HII and Mg2+-dependent RNase HIII from Bacillus subtilis: classification of RNases H into three families. In: Biochemistry. 38, Nr. 2, Januar 1999, S. 605–18. doi:10.1021/bi982207z. PMID 9888800.
- Kochiwa H, Tomita M, Kanai A: Evolution of ribonuclease H genes in prokaryotes to avoid inheritance of redundant genes. In: BMC Evolutionary Biology. 7, Juli 2007, S. 128. doi:10.1186/1471-2148-7-128. PMID 17663799. PMC 1950709 (freier Volltext).