Selenocystein
Selenocystein ist eine Aminosäure. L-Selenocystein (Abk. Sec oder U) ist die 21. proteinogene L-Aminosäure und ein reaktives Analogon des natürlichen L-Cysteins. Selenocystein enthält statt des Schwefelatoms ein Selenatom.
| Strukturformel | ||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
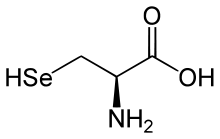 | ||||||||||||||||||||||
| Struktur des natürlich vorkommenden L-Selenocysteins | ||||||||||||||||||||||
| Allgemeines | ||||||||||||||||||||||
| Name | Selenocystein | |||||||||||||||||||||
| Andere Namen |
| |||||||||||||||||||||
| Summenformel | C3H7NO2Se | |||||||||||||||||||||
| Externe Identifikatoren/Datenbanken | ||||||||||||||||||||||
| ||||||||||||||||||||||
| Eigenschaften | ||||||||||||||||||||||
| Molare Masse | 168,0 g·mol−1 | |||||||||||||||||||||
| Sicherheitshinweise | ||||||||||||||||||||||
| ||||||||||||||||||||||
| Soweit möglich und gebräuchlich, werden SI-Einheiten verwendet. Wenn nicht anders vermerkt, gelten die angegebenen Daten bei Standardbedingungen. | ||||||||||||||||||||||
Isomerie
Selenocystein kann in den enantiomeren Formen D und L vorliegen, wobei in Proteinen nur die L-Form, synonym auch (R)-Selenocystein, vorkommt. D-Selenocystein ist enantiomer zu L-Selenocystein und besitzt nur geringe Bedeutung; in der wissenschaftlichen Literatur steht „Selenocystein“ (ohne Präfix) stets für L-Selenocystein.
| Isomere von Selenocystein | ||
| Name | L-Selenocystein | D-Selenocystein |
| Andere Namen | (R)-Selenocystein | (S)-Selenocystein |
| Strukturformel |  | 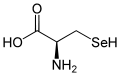 |
| CAS-Nummer | 10236-58-5 | 176300-66-6 |
| 3614-08-2 (DL) | ||
| EG-Nummer | 808-428-7 | – |
| – (DL) | ||
| ECHA-Infocard | 100.236.386 | – |
| – (DL) | ||
| PubChem | 25076 | 5460539 |
| 6326972 (DL) | ||
| Wikidata | Q408663 | Q27110363 |
| Q65017088 (DL) | ||
Eigenschaften
L-Selenocystein ist mit der Aminosäure L-Cystein chemisch nahe verwandt, besitzt jedoch eine niedrigere Säurekonstante von pKs = 5,3 für die Selenolgruppe im Vergleich zu pKs = 8–10 für die Thiolgruppe des L-Cysteins. Auch ist Selenocystein redoxaktiver als Cystein. Diese Eigenschaften dürften ein wesentlicher Grund für den Einbau von L-Selenocystein in Enzyme sein.
.png.webp)
Selenocystein liegt überwiegend als inneres Salz bzw. Zwitterion vor, dessen Bildung dadurch zu erklären ist, dass das Proton von der Carboxygruppe abgespalten wird und vom freien Elektronenpaar des Stickstoffatoms der Aminogruppe aufgenommen wird.
Im elektrischen Feld wandert das Zwitterion nicht, da es als Ganzes ungeladen ist. Genaugenommen ist dies am isoelektrischen Punkt (bei einem bestimmten pH-Wert) der Fall, bei dem das Selenocystein auch seine geringste Löslichkeit in Wasser besitzt.
Biochemie
Der genetische Code gilt für alle Formen des Lebens, jedoch gibt es Besonderheiten. Während der Standardcode es Zellen ermöglicht, Proteine aus den 20 kanonischen α-Aminosäuren herzustellen, können sowohl Vertreter der Archaeen, Bakterien wie Eukaryoten während der Translation Selenocystein über einen als Recodierung bezeichneten Mechanismus einbauen. Der Einbau von L-Selenocystein als zusätzlicher proteinogener Aminosäure ermöglicht oft erst die Funktionsfähigkeit einiger essentieller Enzyme.
Vorkommen
Da Menschen, vielen Tieren, einigen Algen und Einzellern das Codon für die lebensnotwendige 21. Aminosäure Selenocystein fehlt, wird das Stoppcodon UGA aufwändig über ein Umkodieren als Sec ausgelesen. Selbst in Insekten oder Nematoden bilden einige Vertreter Selenocystein, andere nicht. Pilze galten lange als Organismen, denen Selenocystein völlig fehlt, bis man auch hier unter rund 1000 Arten neun Arten fand, die Selenocystein bilden.[2]
Man kennt über 30 eukaryotische und mehr als 15 bakterielle Selenocystein-haltige Proteine. So fand man bei Säugern u. a. verschiedene Glutathion-Peroxidasen, Tetraiodthyronin-Deiodinasen oder große Thioredoxin-Reduktasen und bei Bakterien und Archaeen Formiat-Dehydrogenasen, Hydrogenasen, Protein-Komponenten der Glycin-Reduktase- und D-Prolin-Reduktase-Systeme und mehrere Enzyme des Stoffwechselwegs der Methanbildung als selenocysteinhaltige Enzyme.
Viele der Enzyme vermitteln Redox-Reaktionen. Bei ihnen befindet sich das reaktive Selenocystein im aktiven Zentrum. Bedeutung für Eukaryonten hat die Glutathion-Peroxidase als Teil der zellulären Abwehr von Schäden durch oxidativen Stress. Die Funktion der Selenoproteine ist bei Selenmangel gestört. So tritt die Keshan-Krankheit – eine Kardiomyopathie in Zusammenhang mit Coxsackie-Virusinfektionen – gehäuft auf, wenn es am Spurenelement Selen in der Nahrung mangelt;[3] auch die Kaschin-Beck-Krankheit kommt als Mangelsyndrom in Gegenden mit selenarmen Böden vor. Die Annahme jedoch, dass eine Nahrungsergänzung mit Selen generell Krebs vorbeuge, bestätigte die (SELECT-)Studie nicht.[4]
Biosynthese
Biosynthetisch entsteht L-Selenocystein durch Selenylierung eines L-Serins, das an eine spezifische tRNA gebunden vorliegt:
- Bindung der α-Aminosäure L-Serin (Ser) an eine besondere tRNA, die tRNASec mit dem Anticodon UCA (5′→3′ notiert).
- Diese Ser-tRNASec wird selenyliert, d. h. das L-Serin wird zu L-Selenocystein (Sec) umgesetzt, indem die Hydroxygruppe der Seitenkette durch Selenol (SeH) ersetzt wird. Damit entsteht die Sec-tRNASec.
Der Biosyntheseweg unterscheidet sich also deutlich von anderen Aminosäuren, welche zunächst als freie Aminosäuren gebildet und erst danach an eine tRNA gebunden werden.
Recodierung
Die tRNASec hat das Anticodon UCA und dieses Triplett, gegenläufig 3′-ACU-5′ notiert, kann mit dem Basentriplett des Codons UGA der mRNA paaren. Normalerweise bewirkt UGA als Stopcodon die Termination der Translation. Bilden jedoch besondere Sequenzen der mRNA eine Haarnadelstruktur aus, so wird es möglich, die beladene Sec-tRNASec mit dem Codon UGA zu paaren. Damit wird das Stopsignal ignoriert und Selenocystein an dieser Stelle in das Protein eingebaut. Dieser Vorgang wird auch als Recodierung bezeichnet.
Bei Bakterien findet sich eine solche Secis (selenocysteine insertion sequence) genannte Sequenz der mRNA in unmittelbarer Nachbarschaft zum Codon UGA und nur dieses benachbarte wird dann recodiert. Die Secis-Sequenz wird durch einen spezifischen GTP-abhängigen Translationsfaktor, den Elongationsfaktor SelB, erkannt, welcher zugleich die Sec-tRNASec bindet. Nach dem Einbau des Selenocysteins wird auch die Secis-Sequenz vom Ribosom abgelesen und übersetzt in entsprechende Aminosäuren des Proteins.
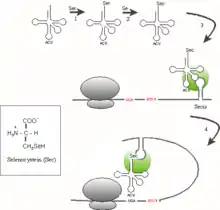
Bei Eukaryoten und Archaeen ist eine Secis-Sequenz hingegen am 3'-Ende der mRNA angebracht, wird nicht vom Ribosom abgelesen, und erlaubt es, alle Codons UGA dieser mRNA zu recodieren.[5] So enthält beispielsweise das menschliche Selenoprotein P an zehn Positionen Selenocysteine.[6]
Der Einbau von L-Selenocystein während der Proteinbiosynthese bei Eukaryoten bedarf weiterer Faktoren (siehe Abbildung):
- Die Sec-tRNASec wird durch den spezifischen GTP-abhängigen Translationsfaktor mSelB gebunden.[7]
- mSelB bildet einen Komplex mit einem weiteren Protein SBP2, welches die Secis-Sequenz erkennt und an sie bindet.
- Dieser Komplex (in der Abbildung als grüne Kugel dargestellt) ermöglicht jeweils die Neuinterpretation: Wird ein Codon
UGAam Ribosom abgelesen und mit dem Anticodon der bereit gehaltenen Sec-tRNASec gepaart, so wird an dieser Position nun Selenocystein eingebaut. - Die Translation muss bei dieser Art des Recodierens dann durch ein anderes Stopcodon, entweder
UAAoderUAG, beendet werden (in der Abbildung als Stop gekennzeichnet).
Die Verhältnisse bei der Selenoproteinsynthese der Archaea sind noch nicht aufgeklärt.
Literatur
- Joseph W. Lengeler, G. Drews, Hans Günter Schlegel: Biology of the prokaryotes. Thieme, Stuttgart 1999, ISBN 3-13-108411-1, S. 185 ff.
Weblinks
- Translationsprozess – Einbau von Sec
- Selenocystein – einige weitere Details (pdf, auf Englisch; 324 kB, 20 Seiten) – Errata: Auf Seite 19 trägt jedes SeCys ein Se (Selen) statt wie dargestellt ein S (Schwefel), bei Cys selbst ist S korrekt.
Einzelnachweise
- Für diesen Stoff liegt noch keine harmonisierte Einstufung vor. Wiedergegeben ist eine von einer Selbsteinstufung durch Inverkehrbringer abgeleitete Kennzeichnung von [No public or meaningful name is available] im Classification and Labelling Inventory der Europäischen Chemikalienagentur (ECHA), abgerufen am 10. Juli 2019.
- Marco Mariotti, Gustavo Salinas, Toni Gabaldón, Vadim N. Gladyshev: Utilization of selenocysteine in early-branching fungal phyla. In: Nature Microbiology. 11. Februar 2019. doi:10.1038/s41564-018-0354-9.
- Jun Lu, Arne Holmgren: Selenoproteins. In: Journal of Biological Chemistry. Band 284, Nr. 2, Januar 2009, S. 723–727. doi:10.1074/jbc.R800045200. PMID 18757362.
- M. Reeves, P. Hoffmann: The human selenoproteome: recent insights into functions and regulation. In: Cellular and Molecular Life Sciences. Band 66, Nr. 15, August 2009, S. 2457–2478. PMID 19399585. PMC 2866081 (freier Volltext).
- Berry, M. J., Banu, L., Harney, J. W., Larsen, P. R.: Functional Characterization of the Eukaryotic SECIS Elements which Direct Selenocysteine Insertion at UGA Codons. In: The EMBO Journal. 12, Nr. 8, 1993, S. 3315–3322. PMID 8344267. PMC 413599 (freier Volltext).
- Burk RF, Hill KE: Selenoprotein P: an extracellular protein with unique physical characteristics and a role in selenium homeostasis. In: Annu Rev Nutr. 25, 2005, S. 215–235. doi:10.1146/annurev.nutr.24.012003.132120. PMID 16011466.
- Fagegaltier D, Hubert N, Yamada K, Mizutani T, Carbon P, Krol A: Characterization of mSelB, a novel mammalian elongation factor for selenoprotein translation. In: The EMBO Journal. 1, 2000, S. 4796–4805. doi:10.1093/emboj/19.17.4796. PMID 10970870.
