Stopcodon
Als Stopcodon oder Terminationscodon, auch Nonsense-Codon, wird in der Genetik ein Codon der Ribonukleinsäure (RNA) bezeichnet, für das keine zugehörige tRNA (transfer-RNA) vorliegt und das daher das Ende einer Sequenz von Nukleotiden darstellt, die an Ribosomen in die Sequenz von Aminosäuren eines Polypeptids übersetzt werden können.
Ein Stopcodon bestimmt damit das Ende eines Leserahmens, der die Biosynthese eines Proteins erlaubt, und ist somit notwendige Bedingung für einen codierenden Nukleinsäureabschnitt. Das Basentriplett eines Stopcodons auf einer mRNA (messenger-RNA) führt bei der Proteinbiosynthese in einer Zelle zum Abbruch der Translation und markiert so – ähnlich dem Punkt am Ende einer Wortfolge bei einem Satz – das Ende der für ein Protein codierenden Nukleotidsequenz und damit dessen Satz an zu verbindenden Aminosäuren. Das Gegenstück zum Stopcodon ist das Startcodon zu Anfang dieser Nukleotidsequenz, wo die Translation beginnt.
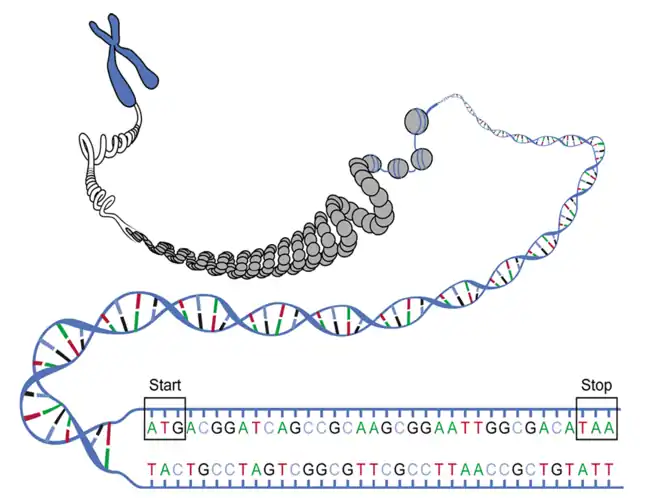
AUGACGGAUCAGCCGCAAGCGGAAUUGGCGACAUAA. Das letzte ist das Stopcodon.Drei aufeinander folgende Nukleinbasen, ein Triplett, bilden die kleinste bedeutungstragende Einheit des genetischen Codes, ein Codon genannt. Jedes Basentriplett innerhalb eines offenen Leserahmens codiert für eine der proteinogenen Aminosäuren, aus denen eine Polypeptidkette aufgebaut wird, ein Triplett der Stopcodons nicht. Diese codieren für keine Aminosäure, da keine zugehörige tRNA zu diesen Codons vorliegt, sondern sie definieren das Ende eines Leserahmens und somit bei der Translation das Ende der Synthese einer Polypeptidkette für ein Protein.
Neben den 61 für Aminosäuren codierenden Basentripletts des genetischen Standard-Codes gibt es drei Kombinationen von Nukleinbasen, mit denen die Translation terminiert werden kann; diese Stopcodons sind auf der mRNA
UAGmit UAG = Uracil – Adenin – GuaninUGAmit UGA = Uracil – Guanin – AdeninUAAmit UAA = Uracil – Adenin – Adenin
Die beiden ersteren Codons können in manchen Organismen unter besonderen Bedingungen auch so interpretiert werden, dass sie jeweils für eine Aminosäure codieren. Voraussetzung dafür ist, dass eine mit der jeweiligen Aminosäure beladene tRNA vorliegt, deren Anticodon-Region an das Codon auf der mRNA bindet. Für die Unterscheidung zum üblichen Stopcodon sind dann zusätzlich noch weitere Umstände nötig, etwa bestimmte Nukleotidsequenzen in der Nachbarschaft bzw. besondere RNA-Strukturen wie Haarnadelbildungen. Bei einigen Lebewesen sind derartige Bedingungen so gegeben, dass das Codon UAG auch in die Aminosäure Pyrrolysin translatiert werden kann, oder das Codon UGA in die Aminosäure Selenocystein. Diese beiden zählen daher – neben den kanonischen zwanzig – ebenfalls zu den natürlich auftretenden genetisch codierten proteinogenen Aminosäuren.
Die Rolle von nonsense-Mutationen, bei denen ein Stopcodon entsteht, konnte in den 1960er Jahren aufgeklärt werden. In diesem Zusammenhang wurde das Basentriplett UAG nach einem Mitglied der Forschergruppe, Harris Bernstein, amber (bernsteinfarben) genannt.[1] In der Folge wurde das Triplett UAA als ochre (ockerfarben) und das Triplett UGA als opal (opalfarben) bezeichnet.[2][3] Diese Namen sind eine Allusion, da die Farben physikalisch und chemisch nichts mit den Basentripletts zu tun haben.
Mutanten
Bakterienmutanten, deren mRNA das durch Punktmutation entstandene Codon UAG enthält, werden auch amber-Mutanten genannt.[4] Eine kompensatorische Mutation in einem tRNA-Gen kann das proteinsynthetisierende System hier dazu befähigen, dieses Codon als sense-Codon zu interpretieren. Mit dem entsprechenden tRNA-Gen im Genom eines Organismus kann das Codon UAG dann in eine (proteinogene) Aminosäure translatiert werden.
Führen Punktmutationen zu einem Stopcodon (sogenannte nonsense-Mutation), resultiert daraus in der Regel ein verkürztes Protein (trunkierende Mutation), sofern die Mutation nicht innerhalb eines Introns liegt, das beim Spleißen wegfällt. Für die Umwandlung des Startcodons AUG in ein Stopcodon wären im Fall von UAG zwei treffende Punktmutationen nötig, bei UGA und UAA drei.
Weblinks
Einzelnachweise
- Bob Edgar: The genome of bacteriophage T4. An archeological dig. In: Genetics, Bd. 168 (2004), S. 575–582, ISSN 0016-6731, PMID 15514035; PDF.
- S. Brenner, L. Barnett, E. Katz, F. Crick: UGA: a third nonsense triplet in the genetic code. In: Nature. Band 213, Nr. 5075, 4. Februar 1967, PMID 6032223, doi:10.1038/213449a0, S. 449–450.
- D. Kasbekar: A cross-eyed geneticist’s view (V.) – How Sydney Brenner, Leslie Barnett, Eugene Katz and Francis Crick inferred that UGA is a nonsense codon. In: Journal of Bioscience. Band 44, Nr. 134, Oktober 2019, doi:10.1007/s12038-019-9955-6; PDF.
- F. Stahl: The Amber Mutants of Phage T4. In. Genetics. Band 141, Nr. 2, Oktober 1995, PMID 8647382, PMC1206745, S. 439–442.