Pyrrolysin
Pyrrolysin (Abk. Pyl oder O) ist eine natürlich auftretende genetisch codierte proteinogene α-Aminosäure und ein Derivat des L-Lysins. Pyrrolysin hat drei stereogene Zentren (Kohlenstoffatome mit vier verschiedenen Substituenten). Damit gibt es acht mögliche Stereoisomere. Das biologisch aktive Isomer (vgl. Bild rechts) hat die vollständige Bezeichnung nach IUPAC: N6-[(2R,3R)-3-Methyl-3,4-dihydro- 2H-pyrrol-2-ylcarbonyl]-(S)-lysin. Pyrrolysin wird auch als 22. proteinogene Aminosäure bezeichnet, ihr Buchstabensymbol „O“ ist seit 2006 Bestandteil des Protein-Alphabets von BLAST.[2]
| Strukturformel | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
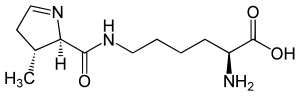 | |||||||||||||
| Allgemeines | |||||||||||||
| Name | Pyrrolysin | ||||||||||||
| Andere Namen |
| ||||||||||||
| Summenformel | C12H21N3O3 | ||||||||||||
| Externe Identifikatoren/Datenbanken | |||||||||||||
| |||||||||||||
| Eigenschaften | |||||||||||||
| Molare Masse | 255,31 g·mol−1 | ||||||||||||
| Sicherheitshinweise | |||||||||||||
| |||||||||||||
| Soweit möglich und gebräuchlich, werden SI-Einheiten verwendet. Wenn nicht anders vermerkt, gelten die angegebenen Daten bei Standardbedingungen. | |||||||||||||
Die Seitenkette von Pyrrolysin ist lipophil.
Gewinnung und Darstellung
L-Pyrrolysin kann in einer mehrstufigen Reaktion ausgehend von (4R,5R)-4-Methylpyrrolin-5-carboxylsäure gewonnen werden.[3]
Translation
Pyrrolysin ist eine der nicht-kanonischen proteinogenen Aminosäuren, die genetisch codiert während der Translation eingefügt als Proteinbaustein in Lebewesen natürlich vorkommen.
Bei einigen methanbildenden Arten von Archaeen der Familie Methanosarcinaceae (z. B. Methanosarcina barkeri) und einzelnen Bakterienarten (z. B. Desulfitobacterium hafniense) ist Pyrrolysin Bestandteil von Enzymen des Methan-Stoffwechsels (Methylamin-Methyltransferasen). Diese Organismen verfügen über eine tRNAPyl sowie eine für deren Beladung spezifische Pyrrolysyl-tRNA-Synthetase und können damit Pyrrolysin bei der Translation am Ribosom einfügen.
Als Codon für diese proteinogene Aminosäure fungiert UAG, dessen übliche Funktion als Stopcodon dabei supprimiert wird. Dies wird bei einigen Archaeenarten anscheinend erleichtert mit besonderen Sequenzen im Kontext des Transkripts, die zur Bildung von Haarnadelstrukturen der mRNA führen, als einem sogenannten PYLIS-Element (einer pyrrolysine insertion sequence). Die Bezeichnung wurde gewählt in Vermutung einer funktionellen Ähnlichkeit mit dem Secis-Element, das bei der Rekodierung eines anderen Stopcodons (UGA) für den Einbau von Selenocystein als proteinogener Aminosäure eine wichtige Rolle spielt. Doch ist hier offenbar eine wirksame Translation des UAG-Tripletts in Pyrrolysin auch ohne Verstärkung seitens solcher cis-agierender kontextueller Elemente möglich – neben dem Ablauf einer Termination.[4]
Evolution
Stammesgeschichtlich handelt es sich bei der Pyrrolysinmaschinerie (Pyl-Maschinerie) nicht um eine neue evolutionäre Errungenschaft. Im Gegenteil, wegen der Einordnung der Pyl-Aminoacyl-tRNA-Synthetase (Pyl-aaRS) zur älteren Klasse IIb ist es wahrscheinlich, dass die Pyl-aaRS aus anderen aaRS-Sequenzen vor der Abspaltung der bakteriellen Stammeslinie von der der Archaeen (letzter gemeinsamer Vorfahre, MRCA: most recent common ancestor) hervorgegangen ist.[5] Damit sind Pyl-aaRS sehr alt.
Warum nur sehr wenige Organismen über die Pyl-Maschinerie verfügen, könnte damit zusammenhängen, dass die Gene in allen anderen Linien im Laufe der Zeit verloren gingen. Dies ist jedoch sehr unwahrscheinlich.
Eine neue Interpretation geht davon aus, dass die Pyl-Maschinerie durch horizontalen Gentransfer aus (mehreren) Donorlinien stammt, die inzwischen ausgestorben sind oder noch nicht entdeckt wurden. Dies setzt aber auch voraus, dass die Donorlinie, aus der die Pyl-Maschinerie stammt, bereits einen gewissen Grad an Diversität zu dem Zeitpunkt erreicht hatte, als noch ein gemeinsamer Vorfahre (MRCA) unserer drei Reiche existierte.
Literatur
- Fournier, G. (2009): Horizontal gene transfer and the evolution of methanogenic pathways. In: Methods Mol Biol. 532; 163–179; PMID 19271184; doi:10.1007/978-1-60327-853-9_9
- Srinivasan, G. et al. (2002): Pyrrolysine encoded by UAG in Archaea: charging of a UAG-decoding specialized tRNA. In: Science. 296(5572), S. 1459–1462 PMID 12029131 doi:10.1126/science.1069588
- Hao, B. et al. (2002): A new UAG-encoded residue in the structure of a methanogen methyltransferase. In: Science. 296(5572), S. 1462–1466. PMID 12029132 doi:10.1126/science.1069556
- Fenske, C. et al. (2003): Wie eindeutig ist der genetische Code? In: Angewandte Chemie. 115(6), S. 626–630. doi:10.1002/ange.200390142
Einzelnachweise
- Dieser Stoff wurde in Bezug auf seine Gefährlichkeit entweder noch nicht eingestuft oder eine verlässliche und zitierfähige Quelle hierzu wurde noch nicht gefunden.
- Release-Notes von BLAST v2.2.15 (Memento des Originals vom 10. März 2016 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis.
- Bing Hao, Gang Zhao, Patrick T. Kang, Jitesh A. Soares, Tsuneo K. Ferguson, Judith Gallucci, Joseph A. Krzycki, Michael K. Chan: Reactivity and Chemical Synthesis of L-Pyrrolysine— the 22nd Genetically Encoded Amino Acid. In: Chemistry & Biology. Band 11, Nr. 9, 2004, ISSN 1074-5521, S. 1317–1324, doi:10.1016/j.chembiol.2004.07.011 (sciencedirect.com).
- D. Longstaff, S. Blight, L. Zhang, K. Green-Church, J. Krzycki: In vivo contextual requirements for UAG translation as pyrrolysine. In: Molecular Microbiology, Band 63, Nr. 1; Januar 2007, S. 229–241; PMID 17140411, doi:10.1111/j.1365-2958.2006.05500.x
- Fournier, G. (2009): Horizontal gene transfer and the evolution of methanogenic pathways. In: Methods Mol Biol. 532; 163–179; PMID 19271184; doi:10.1007/978-1-60327-853-9_9