Anticodon
Ein Anticodon besteht aus den drei Nukleotiden einer tRNA, die als Gegenstück mit den drei Nukleobasen des Codons einer mRNA korrespondieren.
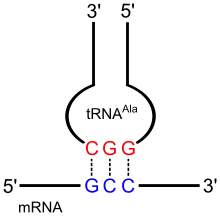
GCC einer mRNA, das für ein Alanin codiert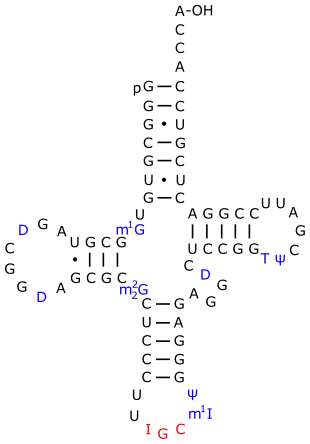
rot hervorgehoben das Triplett Inosin-Guanosin-Cytidin in der 5′→3′ notierten Nukleotidsequenz (neben I an Position 34 des Anticodons auch methyliertes Inosin (m1I) an Position 37)
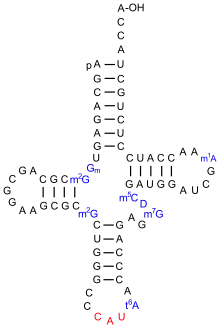
rot hervorgehoben das Anticodon Cytosin-Adenin-Uracil der 5′→3′ notierten Basensequenz; es paart mit dem Codon
AUG als dem StartcodonExponiert auf dem kurzen RNA-Abschnitt der Anticodonschleife eines tRNA-Moleküls finden sich drei aufeinanderfolgende Nukleotide, deren Basen-Abfolge jeweils das charakteristische Anticodon darstellt. Mit diesem Triplett heftet sich die tRNA basenpaarend gegenüber an das Basentriplett eines korrespondierenden Codons der mRNA während der Translation am Ribosom bei der Proteinbiosynthese.
Liegt zum Beispiel auf der mRNA das Triplett GCC als Codon vor, so kann daran eine tRNA über drei komplementäre Basenpaarungen binden mit dem Triplett CGG, als Anticodon (in 5′→3′–Richtung notiert: G-G-C). Ist diese tRNA beladen mit Alanin, so wird diese Aminosäure mit der am Ribosom entstehenden Peptidkette verknüpft.
Einem Codon der mRNA wird damit über das Anticodon einer tRNA eine bestimmte Aminosäure zugeordnet. Die Zuordnung zwischen Codon und Aminosäure bezeichnet man als genetischen Code. Vermittler sind hier die tRNA-Moleküle, die auf der einen Seite ein bestimmtes Anticodon haben und auf der anderen Seite eine spezifische Aminosäure tragen. Mittels der Basenpaarung zwischen Anticodon und Codon wird die codierende Basensequenz eines Polynukleotids wie mRNA abgelesen und übersetzt in die Aminosäuresequenz eines Polypeptids, die Primärstruktur eines Proteins.
Für die Synthese von Proteinen in einer Zelle sind verschiedene tRNA-Moleküle nötig. Unterschiede der tRNA-Spezies bestehen einerseits in der bestimmten Aminosäure, mit der sie je durch spezifische Enzyme – die Aminoacyl-tRNA-Synthetasen – beladen werden, sowie anderseits in ihrem Anticodon. Diese Erkennungsregion stellt das zu einem Codon passende Gegenstück als Sequenz dreier Basen dar. Aber nicht immer ist es erforderlich, dass alle drei Basen je komplementäre Paare mit denen eines Codon-Tripletts bilden; gelegentlich reichen schon zwei Paarungen für die richtige Zuordnung der jeweiligen Aminosäure zu ihrem Codon.
Für Alanin beispielsweise codieren vier Codons (GCU,GCC,GCA,GCG), die alle in 1. und 2. Position gleich sind und damit zugleich von allen anderen 60 Codons unterschieden. Dieser Fall ist nicht ungewöhnlich, sondern für weitere Aminosäuren ähnlich gegeben (Glycin, Prolin, Threonin, Valin etc.). Unter diesen Umständen wird die passende Zuordnung auch dann getroffen, wenn die Paarung nur für die beiden ersten Basen komplementär ist, für die 3. Position des Codons aber etwas wackelig.
So können einige Anticodons mehr als nur ein Codon erkennen, z. B. das Anticodon 3'-CGG-5' neben GCC auch GCU.[1] Abzüglich der drei Stop-Codons enthält der genetische Standard-Code 43 − 3 = 61 verschiedene Codons. Die Zahl an tRNA-Arten in einer Zelle ist oft deutlich geringer. Daraus schloss Francis Crick bereits 1966,[2] dass bestimmte ungenaue Passungen von Codon und Anticodon für die Funktion der tRNA bei der Proteinsynthese ausreichen müssten, und bezeichnete seine Vermutung als Wobble-Hypothese (von englisch wobble ‚wackeln‘).
In allen Organismen finden sich tRNA-Gene (DNA), deren Transkripte (RNA) durch bestimmte Abwandlungen zu reifen tRNA-Molekülen werden (posttranskriptionelle Modifikation). Werden hierbei auch Basen der Anticodonschleife modifiziert, so verändert sich damit das Basenpaarungspotential ihres Anticodons. So kann beispielsweise in vielen eukaryotischen und prokaryotischen Zellen die Nukleinbase Adenin durch besondere Enzyme desaminiert werden zu Hypoxanthin, womit das Nukleosid Adenosin (A) zu Inosin (I) umgewandelt wird (RNA-Editing).[3] Betrifft dies das Anticodon 3'-CGA-5' einer tRNAAla[AGC], das dem Codon GCU komplementär ist, so entsteht ein Anticodon 3'-CGI-5', das nun Wobble-Paarungen bilden kann mit GCU,GCC und auch GCA.
Auf diese Weise können die möglichen Basenpaarungen des Anticodons einer Aminoacyl-tRNA hinsichtlich der dritten Base eines Codons erweitert werden auf solche, die für dieselbe Aminosäure codieren. Doch sind daneben Einschränkungen nötig, um ein korrektes Ablesen der beiden anderen Codonbasen sicherzustellen und Fehlpaarungen auszuschließen. Hier tragen insbesondere die das Anticodon-Triplett flankierenden Basen der Anticodonschleife dazu bei, durch bestimmte Sequenzelemente die translationale Genauigkeit fein abzustimmen,[4] was auch als Entwicklungsprozess verstanden werden kann.[5]
Das Genom im Kern einer menschlichen Zelle enthält an mehr als 600 Orten, verteilt auf nahezu alle Chromosomen, tRNA-Gene (bekannt sind zum Beispiel über dreißig teils unterschiedliche für eine tRNAAla[AGC] und 1 für eine tRNAAla[GGC]).[6] Bezüglich des jeweiligen tRNA-Anticodons werden durch diese genomischen Basensequenzen insgesamt fast alle – ausgenommen sechs – Varianten möglicher Tripletts (aus A, G, C und T) dargestellt. Besondere Bedeutung kommt hier dem DNA-Basentriplett C-A-T zu, das in das Anticodon C-A-U einer tRNA umgeschrieben wird, welches 3'-UAC-5' mit dem Codon AUG paaren kann. Denn dieses codiert für die Aminosäure Methionin und kann, indem eine besondere tRNAiMet als Initiator-tRNA daran bindet, als Startcodon dienen, mit dem dann die Translation beginnt.
Literatur
B. Alberts, A. Johnson, J. Lewis et al.: Molecular Biology of the Cell. 4. Ausgabe. Garland Science, New York 2002, Kapitel: From RNA to Protein (englisch). Auf dem NCBI-Bookshelf online
Einzelnachweise
- B. Mims, N. Prather, E. Murgola: Isolation and nucleotide sequence analysis of tRNAAlaGGC from Escherichia coli K-12. In: Journal of Bacteriology. Band 162 (2), Mai 1985, S. 837; PMC 218931 (freier Volltext).
- F. H. C. Crick: Codon-anticodon pairing: the wobble hypothesis. In: J. Mol. Biol. Band 19, Nummer 2, 1966, S. 548–555 (PMID 5969078; PDF).
- W. Zhou, D. Karcher, R. Bock: Importance of adenosine-to-inosine editing adjacent to the anticodon in an Arabidopsis alanine tRNA under environmental stress. In: Nucleic Acids Research. 41 (5), Januar 2013, S. 3363; PMC 3597679 (freier Volltext).
- S. Ledoux, M. Olejniczak, O. Uhlenbeck: A sequence element that tunes Escherichia coli tRNA(Ala)(GGC) to ensure accurate decoding. In: Nature Structural & Molecular Biology. 16 (4), April 2009, S. 359–364; PMC 2769084 (freier Volltext).
- I. Shepotinovskaya, O. Uhlenbeck: tRNA residues evolved to promote translational accuracy. In: RNA. 19 (4), April 2013, S. 510–516; PMC 3677261 (freier Volltext).
- siehe Einträge für Homo sapiens in der Genomischen tRNA Datenbank (GtRNAdb).