DNA-Methylierung
Bei der DNA-Methylierung handelt es sich um eine chemische Abänderung an Grundbausteinen der Erbsubstanz einer Zelle.[1] Diese Abänderung (Modifikation) wird durch die Übertragung von Methylgruppen durch Enzyme (DNA-Methyltransferasen) auf Nukleobasen an bestimmten Stellen der DNA bewirkt. Da das Grundgerüst der jeweiligen Nukleobase dabei erhalten bleibt, ist die DNA-Methylierung keine genetische Mutation, sondern eine Modifikation.
| Übergeordnet |
| epigenetische Regulation der Genexpression Methylierung von Makromolekülen DNA-Modifikation |
| Gene Ontology |
|---|
| QuickGO |
DNA-Methylierungen kommen in sehr vielen verschiedenen – möglicherweise in allen – Lebewesen vor und haben verschiedene biologische Funktionen. Die Abfolge der DNA-Methylierung kann sich an dem entsprechenden Muster der Mutterzelle orientieren und ist dann Teil des epigenetischen Codes einer Zelle.[2] DNA-Methylierung ist die wichtigste epigenetische Veränderung.[3]
Vorkommen
Organismengruppen
Die DNA-Methylierung ist in Organismen aus allen drei Domänen zu finden. Als Domäne wird hier die oberste Kategorie zur Einteilung der Lebewesen nach Carl R. Woese verwendet. DNA-Methylierungen finden sich also in den beiden Domänen von Lebewesen ohne echten Zellkern, den Bakterien[4] und den Archaeen[5], sowie auch in der Domäne der Lebewesen mit Zellkern, den Eukaryoten.[1]
Die DNA-Methylierung betrifft nicht nur das eigene Erbgut der jeweiligen Zelle, sondern kann auch fremdes Erbgut, z. B. das von Viren betreffen. Darüber hinaus kann sich die DNA-Methylierung von diesen Viren auch auf das Erbgut der Wirtszellen auswirken (z. B. bei Pflanzen[6], dem Menschen[7] oder Bakterien[8]).
Nukleobasen
Bisher (2016) wurden zwei Nukleobasen gefunden, an denen eine natürliche, enzymatische DNA-Methylierung stattfindet: Adenin und Cytosin. Die veränderten Basen sind N6-Methyladenin[9], 5-Methylcytosin[10] und N4-Methylcytosin.[11]
| Grundformen | 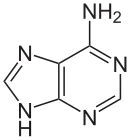 |
 |
||||||
| Adenin, A | Cytosin, C | |||||||
| Veränderte Nukleobasen | 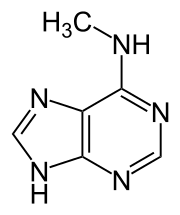 |
 |
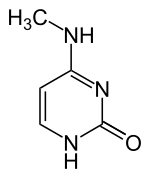 |
|||||
| N6-Methyladenin, 6mA | 5-Methylcytosin, 5mC | N4-Methylcytosin, 4mC | ||||||
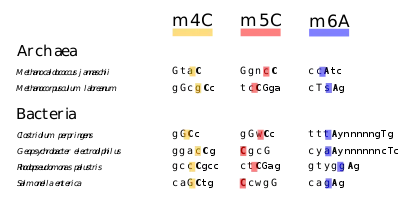
N4-Methylcytosin (m4C), 5-Methylcytosin (m5C) und N6-Methyladenin (m6A). Von den dargestellten sechs Beispielen gehören zwei der Domäne Archaea und vier der Domäne Bacteria an (Angaben aus Blow et al., 2016).[5] In der linken Spalte stehen die Artnamen der Organismen, rechts daneben Beispiele für methylierte DNA-Motive.
(Die vollständigen Namen der Archaeen- bzw. Bakterienstämme lauten nach NCBI-Taxonomie:
„Methanocaldococcus jannaschii DSM 2661“, „Methanocorpusculum labreanum Z“, „Clostridium perfringens ATCC 13127“, „Geopsychrobacter electrodiphilus DSM 16401“, „Rhodopseudomonas palustris CGA009“ und „Salmonella enterica subsp. enterica serovar Paratyphi A str. ATCC 9150“.)
Alle drei Varianten lassen sich in beiden Prokaryoten-Domänen, den Bakterien und den Archaeen finden.[A 1][11] In Eukaryoten ist häufig 5-Methylcytosin vorhanden, das dann an CpG-Stellen auftritt. Allerdings kommt auch N6-Methyladenin vor und wurde zuerst in einigen einzelligen Eukaryoten gefunden. Das betrifft z. B. die Grünalge Chlamydomonas reinhardii und das Wimperntierchen Tetrahymena pyriformis.[12] Das Vorhandensein von N6-Methyladenin in der DNA der Mitochondrien von Säugetieren und den Chromosomen wurde nahezu ausgeschlossen.[13]
Neuere Untersuchungen zeigen, dass N6-Methyladenin als modifizierte Base der DNA bei Eukaryoten eine größere Rolle spielt als zuvor angenommen. Beim Fadenwurm Caenorhabditis elegans[14] und bei der Fruchtfliege Drosophila melanogaster[15][16] ist beispielsweise N6-Methyladenin vorhanden, 5-Methylcytosin jedoch nicht oder kaum.
Luo u. a. (2015) stellen das Vorkommen von N6-Methyladenin und von 5-Methylcytosin bei Eukaryoten gegenüber und machten eine heterogene Verteilung der DNA-Methylierung sichtbar, die wenig an die Verwandtschaftsverhältnisse gekoppelt ist; dabei gibt es Arten, die sowohl N6-Methyladenin als auch 5-Methylcytosin aufweisen, und viele, bei denen nur eine dieser beiden modifizierten Basen in der DNA gefunden werden kann.[17]
Einordnung als epigenetische Modifikation
Die Eukaryoten haben einen Zellkern mit echten Chromosomen. Sie haben Histone, die zusammen mit DNA das Chromatin bilden. Die DNA-Methylierungen befinden sich in enger Wechselwirkung mit den Histon-Modifikationen und der Chromatin-Struktur (z. B. der Packungsdichte der Chromosomen). Das Zusammenwirken von DNA-Methylierungsmustern, Histon-Modifikationen und Chromatin-Struktur ist zentraler Bestandteil der Epigenetik. Die beiden anderen Domänen, die Bakterien und Archaeen sind Prokaryoten. Das heißt, sie besitzen keinen Zellkern und keine echten Chromosomen. Prokaryoten besitzen ein Zellkernäquivalent, das zwar DNA-Methylierungsmuster, aber keine Histone aufweist.
Die Epigenetik ist ein dynamischer Wissenschaftszweig, der auf teilweise vererbbare Phänomene bei Lebewesen mit Zellkern fokussiert ist (Eukaryoten), die nicht direkt an die DNA-Sequenz gekoppelt sind. Je nachdem, wie streng Epigenetik definiert wird, können die Methylierungen von DNA den epigenetischen Zuständen in Zellen (den Epigenomen) zugeordnet werden.
Ein wesentlicher Fakt, der dazu Anlass gibt, auch die DNA-Methylierungen bei Bakterien als epigenetische Veränderungen aufzufassen, ist die Entdeckung der Vererbung von Methylierungszuständen der DNA. Diese Vererbung wurde zuerst bei einem pathogenen Escherichia coli-Bakterium, das Nierenbeckenentzündungen verursachen kann, gefunden.[4] In Kombination mit vielen weiteren Befunden bei Bakterien, die mit der Epigenetik bei Eukaryoten Übereinstimmungen zeigen, wird von bakterieller Epigenetik gesprochen.[18][19][20][21] Allerdings gibt es wesentliche Unterschiede zwischen Bakterien und Eukaryoten (Zellkern und Histone, siehe oben), weshalb der Begriff der bakteriellen Epigenetik zurückhaltend gebraucht wird. Bei Forde u. a. (2015) wird beispielsweise der Ausdruck Methylom (für die Gesamtheit der DNA-Methylierung eines Genoms) verwendet, ohne das Methylom als Teil eines Epigenoms zu bezeichnen.[22]
Bei Ee et al. (2016) wird ebenfalls der Begriff des Methyloms bevorzugt, wenngleich die Analyse der Basen-Modifikationen als epigenomische Analyse bezeichnet wird.[23]
Bisher war vor allem von Organismen mit Zellkern (Domäne Eukaryota) die Rede und den „echten“ Bakterien (Domäne Bacteria). Die Organismen der dritten Domäne, die Archaeen (Archaea, „altertümliche Bakterien“), werden zunehmend Gegenstand der Forschung, da moderne Sequenzierungs- und Analysemethoden die vergleichende Bestimmung von Methylomen erlauben. Blow u. a. (2016) konnten die Methylome von 230 Prokaryoten (Organismen ohne Zellkern) analysieren, neben 217 Bakterien (Bacteria) auch 13 Archaeen. DNA-Methylierungen wurden in 93 % der sequenzierten Organismen gefunden. Die Autoren untersuchten deren DNA-Methylierungs-Muster unter verschiedenen Aspekten und sprechen in der Zusammenschau ihrer Ergebnisse von einer „epigenomischen Landschaft der Prokaryoten“ – hinsichtlich der unterschiedlichen Bindungsspezifitäten von Methyltransferasen, Restriktionsendonuklease-Methyltransferase-Systeme, „verwaisten“ Methyltransferasen [ohne Reastriktionsendonuklease als Partner] und genregulatorische Aktivitäten.[5] Dabei fiel auf, dass DNA-Methylierung ohne zugeordnete Restriktions-Systeme bei Prokaryoten weit verbreitet ist. Die Methylierungsmuster sind zudem evolutionär konserviert, was darauf hindeutet, dass die Methylierung bei der Genom-Regulation eine Rolle spielt.[5]
DNA-Methylierung bei Bakterien
Besonders bei Bakterien hat die Adenin-Methylierung eine wichtige Rolle bei der Fehlerkorrektur der frisch replizierten DNA.[24][25] Innerhalb von GATC-Tetrameren wird das Adenin an der 6-Aminogruppe methyliert (vgl. Bild rechts). Manchmal paart ein Thymin anstelle eines Cytosins mit einem Guanin und wird bei der DNA-Verdopplung irrtümlich eingebaut. Diese und andere Fehlpaarungen können von einem Komplex gefunden werden, der den DNA-Strang absucht und eine Fehlerkorrektur einleitet (proof-reading). Hierbei wird der fehlerhafte Abschnitt im replizierten DNA-Strang herausgeschnitten, der noch keine methylierten Adenine aufweist. Das ausgeschnittene Stück wird alsdann durch ein neu synthetisiertes ersetzt. Ist das proof-reading abgeschlossen, werden die Adenine im neuen Strang methyliert.
Bei Prokaryoten sind DNA-Methylierungen ohne zugeordnete Restriktions-Systeme weit verbreitet. Die Methylierungsmuster sind zudem konserviert, d. h. evolutionär kaum verändert. Das deutet darauf hin, dass die Methylierungen bei der Genom-Regulation eine Rolle spielen.[5]
In einer Überblicksarbeit von Casadesús und Low (2013)[26] wurden Beispiele für Zelldifferenzierung bei Bakterien aufgezählt, die zu verschiedenen Linien führen:
- Sporenbildung durch Bacillus subtilis[27],
- Differenzierung von Rhizobium in stickstofffixierende Bakteroide[28],
- asymmetrische Zellteilung in Caulobacter[29],
- Bildung von Fruchtkörpern durch Myxococcus[30],
- Heterozystenbildung in Cyanobakterien[31] und
- Bildung von Biofilmen bei vielen Bakterienarten.[32][33]
Bei all diesen Phänomenen werden Bakterienzellen mit unterschiedlichen morphologischen und physiologischen Eigenschaften gebildet, während die Genom-DNA-Sequenz intakt bleibt. Casadesús und Low stellten fest,[26] dass lange nach der ersten Anwendung des Begriffs „Epigenese“ durch Conrad Waddington[A 2][34] keine allgemein akzeptierte Definition der Epigenetik vereinbart wurde und favorisierten eine vorläufige Definition, die die Epigenetik als Untersuchung der Zelllinienbildung durch nicht-mutationale Mechanismen anspricht.
Im Bereich bakterieller Epigenetik stellen die Phasenvariationen, die zur Ausbildung von Pili beitragen, Beispiele dar, bei denen die zentrale Beteiligung der DNA-Methylierung gut untersucht ist.[35][36] Wenn eine Methylierungssequenz auf der DNA die Bindungsstelle für ein Protein überlappt, wird die Methylierung dieser Sequenz blockiert, und es kommt zu alternativen Methylierungsmustern.[37][38] Zum Beispiel sind die meisten GATC-Stellen im E. coli-Chromosom vollständig methyliert, außer für eine kurze Zeit nach der DNA-Replikation, in der sie hemimethyliert sind. Einige Stellen sind jedoch aufgrund der Bindung von Proteinen an Stellen, die sich mit einer GATC-Stelle überlappen oder benachbart sind, stabil unmethyliert und konkurrieren mit Dam um Bindung und Blockierung der Methylierung.[35][36][39]
DNA-Methylierung bei Archaeen
Über die DNA-Methylierung bei Archaeen ist weniger bekannt als bei den Bakterien und Eukaryoten. Die technologischen Fortschritte der Gegenwart (2016) machen diese Gruppe zunehmend der umfassenderen Erforschung zugänglich. Nach den bisherigen Ergebnissen dürfte die Methylierung im Prinzip ähnlich umgesetzt werden und ähnliche Aufgaben haben wie bei den Bakterien.[5]
DNA-Methylierung bei Eukaryoten
Bei den Organismen mit echtem Zellkern, den Eukaryoten, ist die Methylierung der zwei Nukleobasen Adenin und Cytosin zu N6-Methyladenin bzw. zu 5-Methylcytosin bekannt.
Hier lassen sich Eukaryoten danach unterscheiden,[17] ob bei ihnen
- die Methylierung von Adenin zu N6-Methyladenin eine Rolle spielt (z. B. der Pinselschimmel Penicillum, der Fadenwurm Caenorhabditis elegans und die Fruchtfliege Drosophila),
- die Methylierung von Cytosin zu 5-Methylcytosin im Vordergrund steht (z. B. die Pflanze Arabidopsis, der Schimmelpilz Neurospora und der Mensch),
- oder sowohl Adenin als auch Cytosin für die DNA-Methylierung genutzt werden (z. B. bei der Grünalge Chlamydomonas).
Wenn Cytosin für die Methylierung der DNA genutzt wird, so handelt es sich in vielen Fällen um die Umwandlung von Cytosin zu 5-Methylcytosin innerhalb von CG-Sequenz-Motiven. Die CG-Methylierung spielt bei der Promotor-Inaktivierung, der Chromatin-Kondensierung, dem genomischen Imprinting und der X-Chromosom-Inaktivierung eine wichtige Rolle (z. B. bei der Acker-Schmalwand[40]).
Bei Wirbeltieren sind zumeist CpG-Dinukleotide jene CG-Sequenz-Motive, die der DNA-Methylierung unterliegen. Das trifft etwa für den Menschen und andere Säugetiere zu. CpG-Inseln sind Regionen im Genom, an denen die CpG-Dinukleotide mit besonderer Häufung vorkommen.[41]
Die Verteilung der CpG-Dinukleotide innerhalb des menschlichen Genoms und die gezielte, selektive Methylierung der Cytosine sind von zentraler Bedeutung für das Verständnis der Epigenetik beim Menschen und die Entstehung von Krankheiten.[42][43]
Bei Pflanzen
Deutliche Fortschritte wurden beim Verständnis der DNA-Methylierung in der Modellpflanze Arabidopsis thaliana erzielt. Die DNA-Methylierung in Pflanzen unterscheidet sich von der von Säugetieren: Während DNA-Methylierung in Säugetieren hauptsächlich am Cytosinnukleotid in einer CpG-Stelle auftritt, kann das Cytosin in Pflanzen an CpG-, CpHpG- und CpHpH-Stellen methyliert werden, wobei H ein beliebiges Nukleotid, aber kein Guanin darstellt. Insgesamt ist die Arabidopsis-DNA hoch methyliert. Durch Massenspektrometrie-Analysen wurde der Anteil modifizierender Cytosine auf 14 % geschätzt.[44]
Die wichtigsten Arabidopsis-DNA-Methyltransferase-Enzyme, die Methylgruppen auf DNA übertragen und kovalent daran binden, sind DRM2, MET1 und CMT3. Sowohl die DRM2- als auch die MET1-Proteine teilen eine signifikante Homologie zu den Säugetier-Methyltransferasen DNMT3 bzw. DNMT1, wohingegen das CMT3-Protein einzigartig für das Pflanzenreich ist.
Es gibt derzeit zwei Klassen von DNA-Methyltransferasen: 1) die De-novo-Klasse von Enzymen, welche Methylierungen auf der DNA neu (de novo) erzeugt und 2) eine „Wartungsklasse“ von Enzymen, welche die Methylierungen als Markierungen auf dem Elternstrang der DNA erkennt und nach der DNA-Replikation entsprechende Methylierungen auf den jeweiligen Tochterstrang überträgt. DRM2 ist das einzige Enzym, das bisher als De-novo-DNA-Methyltransferase betrachtet wird. Es wurde auch gezeigt, dass DRM2 zusammen mit MET1 und CMT3 an der Aufrechterhaltung von Methylierungs-Markierungen durch die DNA-Replikation beteiligt ist.[45] Es werden weitere DNA-Methyltransferasen in Pflanzen exprimiert, die aber keine bekannte Funktion aufweisen (siehe Chromatin-Datenbank).
Es ist nicht klar, wie die Zelle die Orte der De-novo-DNA-Methylierung bestimmt, aber es gibt Hinweise darauf, dass an vielen (wenn auch nicht allen) Stellen RNA-dirigierte DNA-Methylierung (RdDM) beteiligt ist. In RdDM werden spezifische RNA-Transkripte aus einer genomischen DNA-Matrize hergestellt, und diese RNA bildet sekundäre Strukturen, die doppelsträngige RNA-Moleküle genannt werden.[46] Die doppelsträngigen RNAs leiten die De-novo-DNA-Methylierungen der ursprünglichen genomischen Region, die eben diese RNA produziert hat; und zwar entweder über die kleinen interferierenden RNAs (siRNAs) oder über microRNAs (miRNAs).[46] Es wird angenommen, dass diese Art von Mechanismus bei der zellulären Abwehr gegen RNA-Viren und/oder RNA-Transposons wichtig ist, die beide häufig eine doppelsträngige RNA bilden, die für das Wirtsgenom mutagen sein kann. Es wird angenommen, dass diese RNA-Viren und/oder -Transposons durch einen noch wenig verstandenen Mechanismus mithilfe von Methylierung der entsprechenden Orte im Genom abgeschaltet werden und somit nicht länger in der Zelle aktiv sind, wodurch das Genom vor mutagener Wirkung geschützt wäre.
Bei Insekten
Funktionelle DNA-Methylierung wurde in Honigbienen entdeckt.[47][48] Die DNA-Methylierungs-Markierungen befinden sich hauptsächlich innerhalb von Genen. Die gegenwärtige Meinung besagt, dass die DNA-Methylierung bei der Genregulation und dem alternativen Spleißen wirkt.[49] In der Fruchtfliege Drosophila melanogaster ist das DNA-Methylierungs-Niveau für Cytosin fast nicht nachweisbar.[50] Sensitive Methoden, die auf Drosophila-DNA angewendet wurden, schätzen Anteile im Bereich von 0,1-0,3 % des gesamten Cytosins.[51] Dieser niedrige Methylierungsgrad[52] scheint in genomischen Sequenzmustern zu liegen, die sich von den beim Menschen beobachteten Mustern oder anderen Tier- oder Pflanzenarten stark unterscheiden. Die genomische Methylierung in D. melanogaster wurde an spezifischen kurzen Motiven gefunden (konzentriert in spezifischen 5-Basen-Sequenzmotiven, die CA- und CT-reich sind, aber an Guanin abgereichert sind) und ist unabhängig von der DNMT2-Aktivität. Darüber hinaus haben hochsensitive Massenspektrometrie-Ansätze[53] nun gezeigt, dass in den frühesten Stadien der Drosophila-Embryogenese niedrige (0,07 %), aber signifikante Adenin-Methylierungswerte vorliegen.
Bei Pilzen
Viele Pilze haben niedrige Werte (0,1 bis 0,5 %) an Cytosin-Methylierung, während andere Pilze bis zu 5 % des Genoms methyliert haben.[54] Dieser Wert scheint sowohl zwischen den Arten als auch zwischen Isolaten derselben Spezies zu variieren.[55]
Es gibt auch Hinweise darauf, dass die DNA-Methylierung an der zustandspezifischen Kontrolle der Genexpression in Pilzen beteiligt sein könnte. Allerdings wurde bei einer Nachweisgrenze von 250 Attomolen mittels ultra-hochempfindlicher Massenspektrometrie die DNA-Methylierung in einzelzelligen Hefearten wie Saccharomyces cerevisiae oder Schizosaccharomyces pombe nicht bestätigt, was darauf hinweist, dass Hefen diese DNA-Modifikation nicht besitzen.[44] Obwohl Bierhefe (Saccharomyces), Spalthefe (Schizosaccharomyces) und Aspergillus flavus[56] keine nachweisbare DNA-Methylierung aufweisen, hat das Modell des filamentösen Pilzes Neurospora crassa ein gut charakterisiertes Methylierungssystem.[57]
Einige Gene kontrollieren die Methylierung in Neurospora: Eine Mutation der DNA-Methyltransferase (dim-2) eliminiert die gesamte DNA-Methylierung, beeinträchtigt aber weder das Wachstum noch die sexuelle Fortpflanzung. Obgleich das Neurospora-Genom sehr wenig „repeated DNA“ (= Sequenzwiederholungen). aufweist, tritt die Hälfte der Methylierung in solcher DNA auf, die auch Transposon-Relikte und Zentromer-DNA einschließt. Die Möglichkeit, wichtige Phänomene in einem DNA-Methylase-defizienten genetischen Hintergrund zu evaluieren, macht Neurospora zu einem wichtigen System für die Untersuchung der DNA-Methylierung.
Bei niederen Eukaryoten
Die „niederen zellkernhaltigen Organismen“ sind keine phylogenetische Gruppe, also keine Gruppe, in der einheitliche Verwandtschaftsverhältnisse herrschen. Daher ist auch eine Einheitlichkeit hinsichtlich der DNA-Methylierung wenig zu erwarten. Eine DNA-Methylierung ist in Dictyostelium discoidium[58] beispielsweise nahezu nicht vorhanden, da sie mit nur etwa 0,006 % der Cytosine auftritt.[59] Im Gegensatz dazu ist die DNA-Methylierung in Physarum polycephalum[60] weit verbreitet, wo 5-Methylcytosin bis zu 8 % des gesamten Cytosins ausmacht[61]
Epigenetische Veränderungen an CpG-Dinukleotiden
Die wichtigste epigenetische Veränderung ist die Methylierung von Cytosin als Nukleobase der DNA.[62] Dabei werden bei Säugetieren nur solche Cytosine methyliert, die innerhalb von C-G-Dinukleotiden (auch CpG-Dinukleotide oder CpG-Stellen genannt) angetroffen werden. Andere Cytosine werden durch die bekannten menschlichen DNA-Methyltransferasen (DNMT) nicht verändert.[63]
Während der DNA-Verdopplung vor jeder Zellteilung gibt es den alten DNA-Strang, an dem bestimmte Cytosine methyliert sind, während der neugebildete DNA-Strang noch nicht methyliert ist. Das Enzym DNMT3 methyliert jedes Cytosin in einem halbmethylierten CG/CG-Paar. Eine solche CG-Methylierung führt dazu, dass Methyl-CG-erkennende Proteine an diese meCG-Paare binden können. Diese Bindung führt zur Anlagerung weiterer Proteine und zur Verdichtung der Nukleosomen (siehe weiter unten). Dadurch ist die DNA an solchen meCG-Paaren für die RNA-Polymerase nicht ablesbar und das darunterliegende Gen ist inaktiv.
Methylierte Cytosine sind anfällig für Desaminierung, dabei verliert das Cytosin die Aminogruppe an Position 4 des Ringes. Ein desaminiertes, nichtmethyliertes Cytosin ist ein Uracil. Dieses ist keine der vier normalen DNA-Basen Adenin, Cytosin, Guanin oder Thymin. Daher wird ein Uracil in der DNA als Fehler erkannt und ausgetauscht. Wird aber ein 5-Methylcytosin desaminiert, entsteht damit 5-Methyluracil, mit anderem Namen Thymin, das ein übliche DNA-Base ist. In einem DNA-Doppelstrang kann der übliche DNA-Reparaturapparat nicht erkennen, ob das Thymin oder aber das gegenüberliegende Guanin falsch eingebaut ist. Daher ist die Desaminierung eines Methylcytosins in ein Thymin problematisch. Bleibt die Umwandlung erhalten und hat sie in einer Keimzelle stattgefunden, so kann sie auch vererbt werden (als C→T-Punktmutation).
Wenn man auszählt, wie häufig Zweierpaare benachbarter Nukleobasen in den Nukleotiden einer Nukleinsäure insgesamt vorkommen, stellt man fest, dass fast alle Paare etwa gleich häufig sind. Nur Paare von Cytosin und Guanin (CG bzw. GC) kommen wesentlich seltener vor. Als ein Grund dafür wird angenommen, dass methyliertes Cytosin, an dem eine Desaminierung stattfand, nicht repariert wurde, und die Häufigkeit von C daher geringer ist.
Die erhalten gebliebenen CG-Dinukleotide treten gehäuft vor allem in den Genbereichen auf, die für die Steuerung von Genen zuständig sind, den Promotoren. Ein Teil der Promotoren hat eine hohe Dichte an CG-Dinucleotiden, man spricht von einer CpG-Insel (Cytosin phosphat Guanin – Insel). Würden hier CG- in TG-Paare umgewandelt, könnten Zellfunktionen verändert werden oder verloren gehen. Wenn eine solche Veränderung die Existenz der Zelle bzw. des Embryos gefährdet, findet Selektion gegen die Veränderung statt und die Veränderung wird nicht vererbt. CpG-Inseln sind in gesunden Zellen generell unmethyliert, in Promotoren mit niedriger CG Dichte besteht ein qualitativer Zusammenhang zwischen Methylierung und der Aktivität des zugehörigen Gen. Ist ein Promoter methyliert so ist das kontrollierte Gen meist inaktiv.[64]
Cytosin-Methylierungen können durch eine Bisulfit-Sequenzierung bestimmt werden.
| Folgen der Desaminierung—Warum methyliertes Cytosin ein Mutationshotspot ist |
|---|
Szenario und Ziel der Abbildung
Hinweise zu den Bildelementen
|
Abbildung 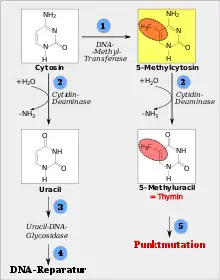 Folgen der Desaminierung, siehe nebenstehenden Text Das nebenstehende Bild wird hier anhand von fünf Punkten erläutert: ( 1 ) Die Nukleobase Cytosin kann durch eine DNA-Methyltransferase methyliert werden, wodurch 5-Methylcytosin entsteht. ( 2 ) Sowohl Cytosin als auch 5-Methylcytosin können, z. B. durch eine Cytidin-Deaminase, desaminiert werden. Wird auf der einen Seite (links) Cytosin desaminiert, entsteht Uracil; wenn auf der anderen Seite (rechts) 5-Methylcytosin desaminiert wird, entsteht 5-Methyluracil = Thymin. ( 3 ) Das links gezeigte Uracil ist DNA-fremd und kann daher sehr gut vom DNA-Reparatur-Apparat erkannt werden. Die falsche Base kann durch Uracil-DNA-Glycosidase ausgeschnitten werden, was zu einer vorübergehenden Lücke in der DNA führt (Basenexzision). ( 4 ) Eine Lücke, die durch die Uracil-DNA-Glycosidase entstanden ist, wird zumeist korrekt mit Cytosin aufgefüllt. Dadurch wird der Ausgangszustand wieder hergestellt und das Ergebnis wäre eine erfolgreiche DNA-Reparatur. ( 5 ) Das rechts gezeigte Thymin ist im Gegensatz zum links gezeigten Uracil eine DNA-eigene Base und wird deshalb vom DNA-Reparatur-Apparat weniger rigoros behandelt als Uracil. Im Fall der Akzeptanz würde der DNA-Reparatur-Apparat dem Thymin im gegenüber liegenden DNA-Strang ein komplementäres Guanin zuordnen und damit die Umwandlung von Cytosin zum Thymin (C→T-Transition) fixieren. Das Ergebnis wäre eine Punktmutation. |
DNA-Methyltransferasen (DNMT)
Bislang sind drei menschliche DNA-Methyltransferasen (DNMT) bekannt: DNMT1, DNMT3a und DNMT3b (DNMT2 methyliert RNA). Für die Erhaltungs-Methylierung (Maintenance-Methylierung) bei der Zellteilung ist DNMT1 zuständig. DNMT3a und DNMT3b methylieren die CG-Dimere, die aufgrund von Zelldifferenzierungen neu methyliert werden (De-novo-Methylierung).
An methylierte DNA können sich Proteine mit spezifischer Methyl-CpG-bindender Domäne (MBD) anlagern,[69] wozu beim Menschen beispielsweise die Proteine MECP2, MBD1, MBD2 und weitere zählen. Eine solche Anlagerung kann wiederum den Keim für zusätzliche Anlagerungen besonderer Proteine bilden, die schließlich auch zur Modifizierung von Histonen führen. Die über Histon-Deacetylasen wie HDAC4 herbeigeführten Histonmodifikationen begünstigen die Kondensierung von Chromatin und können somit die Transkription der DNA erschweren und zur Inaktivierung eines Chromosomenabschnittes beitragen.[70]
DNA-Demethylase
Auch das Methyl-abspaltende Enzym DNA-Demethylase wurde identifiziert.[71] Es war als Methyl-CpG-Domäne-bindendes Protein 2 (MBD2) schon früher beschrieben worden. Damit ist die Methylierung von DNA keine Einbahnstraße, sondern der Methylierungszustand kann zellfunktionsabhängig geregelt werden. Eine solche Situation nennt man plastisch.
Embryonalentwicklung
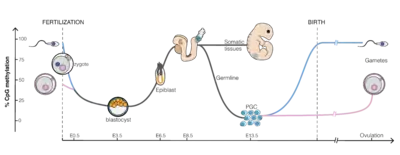
(Erläuternde Verweise zur englischen Beschriftung: CpG methylation=CpG-Methylierung; Fertilization=Befruchtung; Zygote; Blastozyste; Epiblast; Somatic tissues=somatische Zellen im Gewebe; Germline=Keimbahn; PGC=Urkeimzellen; Birth=Geburt; Gametes=Keimzellen; E0.5 ... E13.5 Tage der Embryonal-Entwicklung nach der Befruchtung; Ovulation=Follikelsprung)
Zwischen den Generationen werden die DNA-Methylierungsmuster bei Säugetieren größtenteils gelöscht und wiederhergestellt. Die Löschung betrifft fast alle Methylierungen der Eltern. Diese Demethylierung mit anschließender Remethylierung findet zwei Mal statt: zuerst während der Gametogenese und erneut in der frühen Embryogenese. Die Demethylierung in der frühen Embryogenese erfolgt in der Präimplantationsphase in zwei Stadien – zunächst in der Zygote, dann in den ersten embryonalen Replikationszyklen von Morula und Blastula. Eine Methylierungswelle findet dann während der Implantationsphase des Embryos statt, wobei die CpG-Inseln vor der Methylierung geschützt sind. Dadurch werden die Haushaltsgene in allen Zellen exprimiert. Danach, in der Postimplantationsphase, sind die DNA-Methylierungsmuster stadien- und gewebespezifisch, das heißt, sie weisen die Unterschiede auf, die jeden einzelnen Zelltyp definieren und bleiben über einen langen Zeitraum stabil.[72]
Obwohl die DNA-Methylierung per se für das transkriptionale Silencing (=Abschaltung der Genaktivität) nicht notwendig ist, wird angenommen, dass sie dennoch einen „gesperrten“ Zustand darstellt, der die Transkription definitiv inaktiviert. Insbesondere scheint die DNA-Methylierung entscheidend für die Aufrechterhaltung des monoallelischen Silencing im Kontext des genomischen Imprintings und der Inaktivierung des X-Chromosoms zu sein.[73][74]
In diesen Fällen unterscheiden sich die exprimierten und stummen Allele durch ihren Methylierungsstatus, und der Verlust der DNA-Methylierung führt zu einem Verlust der Prägung und Re-Expression von Xist in somatischen Zellen. Xist (X-inactive specific transcript) ist ein RNA-Gen auf dem X-Chromosom der Plazenta-Säugetiere, das als Haupteffektor des X-Inaktivierungsprozesses fungiert.[75] Während der Embryonalentwicklung verändern nur wenige Gene ihren Methylierungsstatus. Eine wichtige Ausnahme sind viele Gene, die spezifisch in der Keimbahn exprimiert werden.[76]
Die DNA-Methylierung erscheint in differenzierten Zellen unbedingt erforderlich, da der Knock-Out einer der drei wirksamen DNA-Methyltransferasen zu einem Absterben als Embryo oder nach der Geburt führt. Im Gegensatz dazu ist die DNA-Methylierung bei undifferenzierten Zelltypen wie der inneren Zellmasse der Blastozysten, Urkeimzellen oder embryonalen Stammzellen entbehrlich. Da die DNA-Methylierung nur eine begrenzte Anzahl von Genen direkt zu regulieren scheint, bleibt offen, wie genau die Abwesenheit von DNA-Methylierung den Tod differenzierter Zellen verursacht.
Obwohl mütterliche und väterliche Genome bei jedem Durchgang durch die Keimbahn neu programmiert werden, sind sie unterschiedlich. Dieses Phänomen wird genomische Prägung genannt. Während der Gametogenese werden die DNA-Methylierungsmuster in den Urkeimzellen gelöscht und auf der Grundlage des Geschlechts des sendenden Elternteils wiederhergestellt. Nach der Befruchtung werden die väterlichen und mütterlichen Genome erneut demethyliert und remethyliert (mit Ausnahme von differenziell methylierten Regionen, die mit geprägten Genen assoziiert sind). Diese Umprogrammierung ist wahrscheinlich für die Totipotenz des neugebildeten Embryos und die Auslöschung erworbener epigenetischer Veränderungen erforderlich.[77]
Die Rolle der Methylierung von CpG-Stellen für das Gedächtnis
Bei Säugetieren weisen die DNA-Methyltransferasen eine Präferenz für die Cytosine auf, die sich innerhalb von CpG-Stellen befinden.[78] Im Gehirn von Mäusen werden 4,2 % aller Cytosine methyliert, wobei hauptsächlich 5-Methylcytosin an CpG-Stellen (5mCpG ) gebildet wird.[79] Die meisten hypermethylierten 5mCpG-Stellen verstärken die Unterdrückung assoziierter Gene.[79]
Duke et al. beschreiben, dass die DNA-Methylierung in den Neuronen durch neuronale Aktivität verändert wird und dies die Unterdrückung der Expression bestimmter Gene bedingt.[80] Eine neuronale DNA-Methylierung ist für die synaptische Plastizität erforderlich und wird durch Erfahrungen verändert; für die Gedächtnisbildung und -erhaltung ist eine aktive DNA-Methylierung und -Demethylierung erforderlich.[80]
Halder et al. (2016) unterzogen Mäuse[81] und Duke et al. (2017) unterzogen Ratten[80] einer kontextabhängigen Angstkonditionierung, wodurch sich ein besonders starkes Langzeitgedächtnis bildete. 24 Stunden nach der Konditionierung wurde in der Hippocampus-Gehirnregion von Ratten die Expression von 1048 Genen herunterreguliert (was normalerweise mit 5mCpG in Genpromotoren assoziiert ist), und die Expression von 564 Genen wurde hochreguliert (was häufig mit einer Hypomethylierung von CpG-Stellen in Genpromotoren verbunden ist). 24 Stunden nach dem Training waren 9,2 % der Gene im Rattengenom von Hippocampus-Neuronen differentiell methyliert. Der Hippocampus ist zwar für das Lernen neuer Informationen unerlässlich, speichert jedoch selbst keine Informationen. In den Maus-Experimenten von Halder wurden im Hippocampus eine Stunde nach der kontextabhängigen Angstkonditionierung 1206 differentiell methylierte Gene beobachtet, diese veränderten Methylierungen wurden jedoch nach vier Wochen wieder zurück gebildet. Im Gegensatz zum Fehlen langfristiger CpG-Methylierungsänderungen im Hippocampus konnte eine erhebliche differentielle CpG-Methylierung während der Gedächtniserhaltung in kortikalen Neuronen nachgewiesen werden. Vier Wochen nach kontextueller Angstkonditionierung gab es 1223 differentiell methylierte Gene im anterioren cingulären Cortex von Mäusen.
Biologische Funktionen von DNA-Methylierungen
Die DNA-Methylierung ist sehr weit verbreitet (siehe oben) und hat daher eine entsprechende Vielfalt biologischer Funktionen, z. B. auf dem Gebiet der Epigenetik. Im Folgenden wird auf einige funktionelle Aspekte der DNA-Methylierung näher eingegangen:
Bei Prokaryoten
- Schutz vor fremder DNA: Unterscheidung zelleigener DNA von solcher, die von außen in die Zelle gelangt ist.
- Fehlerkorrektur bei der DNA-Synthese: Unterscheidung des ursprünglichen (methylierten) DNA-Strangs vom neusynthetisierten Strang, in welchem die Nukleobasen noch nicht methyliert sind.
Bei Eukaryoten
- Nutzung der DNA als Informationsträger: Markierung von aktiven und inaktiven Bereichen der DNA, unter anderem abhängig vom Lebensalter.[82][83][84][85]
DNA-Methylierung und Schutz vor fremder DNA
DNA ist weit verbreitet. Zellen unterschiedlicher Arten können in unmittelbarer Nachbarschaft existieren. Sowohl bei der Nahrungsaufnahme einer Zelle (z. B. Phagozytose) wie auch bei parasexuellen und sexuellen Prozessen kommt es zur Aufnahme der DNA von einer (lebenden oder toten) Zelle in eine andere Zelle. Darüber hinaus sind viele Zellen in der Lage, unter bestimmten Umständen Fremd-DNA leicht aufnehmen zu können (Zellkompetenz).
Da eine lebende Zelle ihre Integrität nur erhalten kann, wenn die genetische Information sinnvoll ist, sollte sie in der Lage sein, fremde DNA zu erkennen und zu eliminieren. Dies wird häufig durch ein System aus zwei Enzymgruppen gewährleistet: Die DNA-Methyltransferasen und die Restriktionsendonukleasen.
Die Methyltransferasen erkennen eine (meist kurze) DNA-Sequenz und hängen eine Methylgruppe an eine definierte Nukleobase. Dadurch entsteht ein sogenanntes Methylierungsmuster. Daneben erkennen die Restriktionsenzyme jeweils eine (meist kurze) DNA-Sequenz und trennen die DNA an definierten Stellen zwischen Phosphat und Desoxyribose bei bestimmten vorhandenen oder abwesenden Methylierungen. Viele Restriktionsenzyme sind methylierungssensitiv. Das heißt, sie zerschneiden die DNA nur, wenn an bestimmten Stellen Methylierungen vorliegen oder wenn an bestimmten Stellen keine Methylierungen vorhanden sind.
Das System aus Methyltransferasen und Restriktionsenzymen ist in einer lebenden Zelle so abgestimmt, dass die eigene DNA nicht zerschnitten wird. Fremde DNA, die von außen in die betrachtete Zelle gelangt, hat jedoch in den allermeisten Fällen ein anderes Methylierungsmuster. Daher wird sie mit hoher Wahrscheinlichkeit von den Restriktionsenzymen sowie anderen Nukleasen verdaut. In seltenen Fällen wird fremde DNA nicht oder nur zum Teil verdaut und dauerhaft in die zelleigene DNA integriert. Eine Integration fremder DNA wird auch als horizontaler Gentransfer bezeichnet und ist ein Motor der Evolution.
Nachfolgend wird ein einfaches System aus Methyltransferase und Restriktionsenzym als Beispiel erläutert.
Beispiel für DNA-Methylierung und DNA-Restriktion
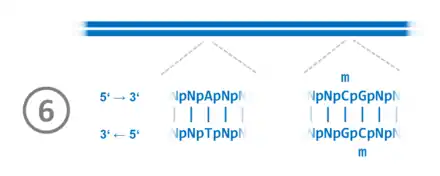
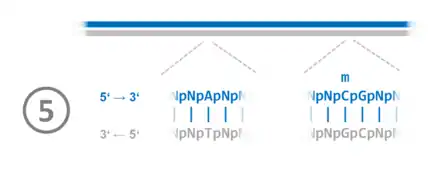
Das Zusammenwirken von DNA-Methylierung und DNA-Restriktion (Spaltung von DNA) soll anhand der Enzyme DpnM (DNA-Methyltransferase) und DpnII (Restriktionsenzym) beschrieben werden. Die Enzyme stammen aus dem Bakterium Diplococcus pneumoniae. Die Methyltransferase DpnM sorgt dafür, dass die palindromische Sequenz GATC im Adenosin methyliert wird:
m
--GATC--
--CTAG--
m
Dadurch kann „frische DNA“ die gerade neu entstanden ist, von der alten DNA, die als Vorlage gedient hat, unterschieden werden:
m --GATC-- --CTAG--
Das ist für die korrekte Reparatur von Fehlern während der DNA-Replikation wichtig. Der sogenannte hemimethylierte Zustand (eine Seite ist methyliert, die andere nicht) wird nachfolgend durch die Methyltransferasen – wie z. B. DpnM – durch Methylierung aufgehoben. Sollte DNA einer anderen Art in die D. pneumoniae-Zelle gelangen, so ist diese DNA in der Sequenz GATC meist nicht methyliert:
--GATC-- --CTAG--
Diese doppelsträngige DNA wird mit großer Wahrscheinlichkeit vom Restriktionsenzym DpnII zerschnitten. Neben anderen Prozessen führt das dazu, dass fremde DNA eher als Nahrung und weniger als Erbsubstanz dient. Dieses ist außerdem ein Mechanismus, mit dem sich Bakterien vor Bakteriophagen schützen – indem sie die eingebrachte DNA in kleine Stücke schneiden.
DNA-Methylierung und Fehlerkorrektur bei der DNA-Neusynthese
Die identische Verdopplung der Desoxyribonukleinsäure (DNA-Replikation) ist eine wesentliche Voraussetzung für die Zellteilung und damit für die Vermehrung. Die DNA-Replikation wird dadurch gewährleistet, dass Enzyme (die DNA-abhängigen DNA-Polymerasen) den bereits vorhandenen alten Strang „lesen“ und dabei den neuen Strang „schreiben“. Dabei können Fehler auftreten. Die Fehlerstellen können durch die DNA-Reparatursysteme einer Zelle erkannt werden, da dort keine komplementäre Basenpaarung vorliegt. Allerdings bliebe unklar, welche der beiden Möglichkeiten die richtige sei, wenn sich alter und neuer DNA-Strang nicht unterschieden. Da der alte Strang jedoch methyliert ist, der neue aber noch nicht, ist eine Unterscheidung möglich. Die DNA-Reparatursysteme von Bakterien können diesen hemimethylierten („halbmethylierten“) Zustand zur postreplikativen Fehlerkorrektur nutzen.
Bei Eukaryoten werden die Reparaturenzyme z. B. durch das Ringklemmenprotein (PCNA – Proliferating-Cell-Nuclear-Antigen) rekrutiert, welches die Stränge während der Replikation auseinander hält. Es gibt allerdings weitere Reparatur-Mechanismen. In den Zellen des Menschen wie auch anderer Säugetiere ist eine kurze Folge aus zwei Grundbausteinen (Nukleosiden) die Grundlage für eine DNA-Methylierung: das CpG-Dinukleotid (Desoxycytidin – Phosphorsäure – Desoxyguanosin). In der Aufeinanderfolge der beiden Nukleobasen Cytosin–Guanin wird das Cytosin methyliert. Bis auf einige Bereiche passiert dies fast in der gesamten menschlichen Erbsubstanz. Auch hier ist (wie bei den Bakterien) ein hemimethylierter Zustand für die postreplikative Reparatur entscheidend. Im Folgenden wird eine solche Reparatur anhand eines Beispiels vereinfacht, in sechs Schritten dargestellt:
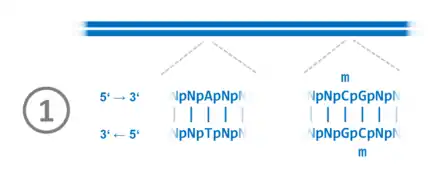 Schritt 1 (6), Abbildungsserie zu halbmethylierten CpG-Stellen, Replikation und Reparatur |
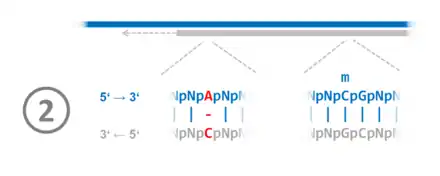 Schritt 2 (6), Abbildungsserie zu halbmethylierten CpG-Stellen, Replikation und Reparatur |
|
1. Vor der DNA-Replikation sind im betrachteten Beispielabschnitt die CpG-Dinukleotide in beiden Strängen am Cytosin methyliert. 2. Während der DNA-Replikation kommt es zum Fehler: Statt eines Thymidintriphosphat-Moleküls wird ein Cytidintriphosphat verwendet. Dadurch wird an der entsprechenden Stelle die komplementäre Basenpaarung aufgehoben. 3. Nach Abschluss der DNA-Replikation im Beispielabschnitt liegt der DNA-Doppelstrang hemimethyliert vor. Das heißt, der alte Strang ist methyliert, der neue nicht. Ein Protein-Komplex bindet an die halbseitig methylierten CpG-Stellen (Hemi-mCpG-Np95-Dnmt1) und ermöglicht ein nachträgliche Reparatur bis in einen Bereich hinein, in welchem der DNA-Doppelstrang bereits beginnt, sich mit den Histonen zum Chromatin zu assemblieren.[86] 4. Die Stelle der Fehlpaarung wird erkannt. Von den beiden Möglichkeiten Adenosinmonophosphat und Cytidinmonophosphat wird das Cytidinmonophosphat ausgeschnitten, das im nichtmethylierten Strang liegt. Tymidintriphosphat wird verwendet, um die Lücke zu schließen. 5. Der reparierte DNA-Doppelstrang liegt im hemimethylierten Zustand vor. 6. Durch die Übertragung von Methylgruppen auf die Nukleobase Cytosin in den CpG-Dinukleotiden wird der Grundzustand wiederhergestellt. |
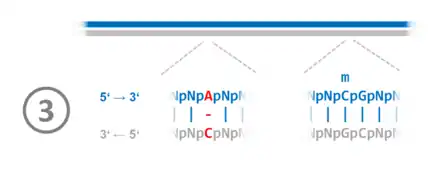 Schritt 3 (von 6), Abbildungsserie zu halbmethylierten CpG-Stellen, Replikation und Reparatur 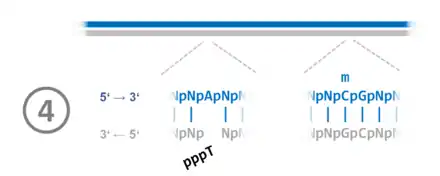 Schritt 4 (von 6), Abbildungsserie zu halbmethylierten CpG-Stellen, Replikation und Reparatur |
 Schritt 6 (von 6), Abbildungsserie zu halbmethylierten CpG-Stellen, Replikation und Reparatur |
 Schritt 5 (von 6), Abbildungsserie zu halbmethylierten CpG-Stellen, Replikation und Reparatur |
DNA-Methylierung und die Nutzung der DNA als Informationsträger
DNA-Methylierungen sind Markierungen, die es der lebenden Zelle gestatten, Bereiche innerhalb der DNA für verschiedene Prozesse selektiv zu nutzen. Die Markierung von DNA kann ähnlich wie Textformatierungen in einem Buch betrachtet werden: Wenn in einem Lexikon ein Stichwort hervorgehoben dargestellt ist, hat es für den Leser eine andere Bedeutung als dasselbe Wort im Fließtext. Es existieren mehrere (sich überschneidende) Möglichkeiten, wie DNA-Methylierungen die Art der Interpretation von Information variieren, welche in der Bausteinabfolge der DNA gespeichert ist.
DNA-Methylierung und Genregulation
In einem Bereich vor einem Gen (stromaufwärts, upstream) sind häufig Stellen vorhanden, die sich hinsichtlich ihres Methylierungsmusters von der Umgebung unterscheiden. Dabei kann in vielen Fällen der Methylierungsgrad in unterschiedlichen Situationen variieren. Dadurch wird eine selektive Lesehäufigkeit des dahinterliegenden Gens möglich, was man als Genregulation oder differenzielle Genexpression bezeichnet. Beispiele für solche Bereiche, die selektiv methyliert sein können, sind CpG-Inseln.
DNA-Methylierung und Imprinting (genomische Prägung)
Die genomische Prägung ist ein Spezialfall einer Genregulation, welche in der Regel durch DNA-Methylierung gesteuert wird. Durch unterschiedliche DNA-Methylierungsmuster in den männlichen und weiblichen Keimzellen können väterliche und mütterliche Allele unterschieden werden. Bei Genen, die dem Imprinting unterliegen, wird nur das mütterliche oder väterliche Allel genutzt. Dadurch ist eine geschlechtsspezifische Ausprägung von phänotypischen Merkmalen möglich.
Medizinische Bedeutung
Da fehlerhafte DNA-Methylierungen auf Zellebene reduzierte oder erhöhte Genaktivität bedingen und diese Aktivitätsveränderungen meist stabil an Tochterzellen vererbt werden, sind sie auf Organismenebene häufig auch Ursache für Krankheiten. So weisen z. B. Tumorzellen oft Methylierungsmuster auf, die von denjenigen gesunder Gewebe signifikant abweichen. Ein Tumor kann dabei sowohl als Folge zu starker Methylierung (Hypomethylierung-Hypermethylierung) von upstream DNA-Bereichen entstehen, als auch bei verringertem Methylierungsgrad.[87] Der regulatorische Bereich vor jedem Gen (Promotorbereich) besteht aus verschiedenen typischen DNA-Sequenzen, die spezielle Bindungsstellen für unterschiedliche Enzyme darstellen. Meistens blockiert eine hypermethylierte upstream DNA den Zugang transkriptionsaktiver Faktoren und Enzyme, wodurch die Genaktivität des nachfolgenden Gens supprimiert wird.
Die DNA-Bereiche, die für die Methylierung von besonderer Bedeutung sind, heißen CpG-Inseln. Ihr GC-Gehalt beträgt etwa 60 % (Gesamtgenom: ca. 40 %), und in diesen Abschnitten liegt das Dinukleotid Cytosin-Guanin (5'-CpG-3') im Vergleich zum restlichen Genom mit zehn- bis zwanzigmal erhöhter Frequenz vor. CpG-Inseln dienen in der humangenetischen Forschung oft der Zuordnung von Genen zu genetischen Erkrankungen. Die Gene und die durch DNA-Methylierung gesteuerten Bereiche vor dem jeweiligen Gen können für die Diagnose von vererbbaren Erkrankungen mit molekulargenetischen Methoden eingesetzt werden.
Eine Therapie von Erkrankungen durch eine gezielte Beeinflussung der DNA-Methylierung ist bisher und auf absehbare Zeit nicht möglich – unter anderem auch deshalb, weil zu wenig über das ‚richtige‘ Methylierungsmuster gesunder Gewebe bekannt ist. Derzeit gibt es nur experimentelle In-vitro-Ansätze, durch sogenannte Zinkfingerproteine (spezielle Klasse von Proteinen, die um ein zentrales Zink-Ion DNA-bindende Domänen besitzen und mit Methylasen oder Demethylasen gekoppelt sein können), um so gezielt bestimmte Sequenzen modifizieren zu können.
Regulation der DNA-Methylierung in Tumoren
Die DNA-Methylierung in Tumorzellen unterscheidet sich von derjenigen in gesunden Zellen.
- Die Analyse der DNA-Methylierung von Tumorzellen hat ergeben, dass in Tumorzellen häufig die Gene für sogenannte Tumorsuppressorproteine im Vergleich zu Normalzellen methyliert sind.
- So ist in der akuten myeloischen Leukämie (AML) häufig die CG-Insel des P15-Proteins (auch CDKN2B oder ink4b genannt) methyliert.
- P15 ist ein hemmender Regulator des Zellzyklus.
- Nach Bildung von meCG in der CG-Insel von P15 wird dessen Transkription und die Biosynthese des P15-Proteins eingestellt.
- Beim Zellzyklus-Regulator P53 ist in 50 % aller menschlichen Tumoren das P53-Gen hypermethyliert und damit inaktiviert.[62]
- Da P53 das proof-reading kontrolliert, wird durch Ausschalten von P53 die Fehlerkontrolle aufgegeben und Mutationen können sich anhäufen, die zur Ausschaltung weiterer Tumorsuppressor-Gene oder zur Aktivierung zellwachstums-fördernder Proteine führen können.
Andererseits ist in Tumorzellen die globale DNA-Methylierung geringer als in Normalzellen. Das führt man darauf zurück, dass das in Normalzellen hochmethylierte Heterochromatin (vor allem die Zentromer-Region) in Tumorzellen geringer methyliert ist.
Seitdem man den Einfluss der Hypermethylierung auf das Tumorwachstum identifiziert hat, hat man nach Wegen gesucht, um durch Demethylierung die im Entstehen begriffenen bzw. auch schon existierende Tumoren wieder der Zellzykluskontrolle zu unterwerfen:[88]
- Substanzen, die auf einer Abwandlung der Base Cytosin beruhen, wie Azacytidin[89] oder Aza-Desoxy-Cytidin,[90] werden in Patienten mit Akuter Myeloischer Leukämie infundiert.
- Diese Stoffe werden in Zellen aufgenommen, deren DNA verdoppelt wird.
- Azacytidin kann in der Zelle in Aza-Desoxy-Cytidin umgewandelt werden.
- Aza-Desoxy-Cytidin wird anstelle von Cytidin in DNA eingebaut.
- Die DNMT3, die die hemimethylierten CGs methylieren will, bindet an das Aza-Analog.
- Der Austausch von Kohlenstoff gegen Stickstoff bewirkt, dass das Enzym bei dem enzymatischen Methyltransfer an der DNA hängenbleibt und keine weiteren Reaktionen durchführen kann.
- Mit diesem Verfahren werden die DNMT3 inaktiviert und eliminiert. Eine Methylierung findet nicht mehr statt.
- Nach der nächsten Zellteilung ist die DNA weniger methyliert. Wenn von dieser De-Methylierung z. B. das P53- oder das P15-Gen betroffen sind, findet wieder Zellzykluskontrolle statt.
- Das Tumorwachstum ist damit unterbunden.
Es wurden klinische Studien veröffentlicht, in denen bei menschlichen Patienten ein hemmender Effekt von Aza-Desoxy-Cytosin auf Tumorentwicklung gezeigt werden konnte.[90] Die Forscher nennen ihr Verfahren Epigenetische Therapie.
Für die Behandlung des Myelo-Dysplastischen Syndroms, das sich häufig zu einer Akuten Myeloischen Leukämie entwickelt, wurde 5-Aza-2'-Desoxy-Cytidin unter dem Namen Dacogen von der FDA im Jahre 2006 als Medikament freigegeben.[91] Ein anderer Name für diese Substanz ist Decitabin.
Abgrenzung von Begriffen im Zusammenhang mit der DNA-Methylierung
Methylierung
Die Methylierung ist eine universelle chemische Abwandlung von Molekülen. Im Bereich anorganischer und organischer Chemie nennt man die abgewandelten Moleküle auch Derivate, bei der biologischen Betrachtung derart abgewandelter Makromoleküle spricht man von Modifikationen.
So können außer den Nukleobasen in der DNA auch Proteine durch Methyltransferasen methyliert werden.
DNA-Methylierung
In engerem Sinn bezeichnet DNA-Methylierung die natürliche, enzymatische Übertragung von Methylgruppen (–CH3) auf die Nukleinbasen der DNA. Weiterhin wird auch das Resultat dieses Vorgangs so bezeichnet: das natürliche Vorkommen von Methylierungen als chemischen Abänderungen an den Grundbausteinen (Nukleinbasen) der Erbsubstanz (DNA). Die DNA-Methylierung ist prinzipiell reversibel (rückführbar). Die Entfernung von Methyl-Gruppen heißt Demethylierung.
In erweitertem Sinn wird der Ausdruck „DNA-Methylierung“ für komplexe biologische Prozesse verwendet, die das Erstellen und Löschen von DNA-Methylierungs-Mustern einschließen (z. B. Embryogenese der Säugetiere).
DNA-Demethylierung
DNA-Demethylierung bezeichnet die Änderung von Nukleobasen, die zuvor methyliert waren und dies anschließend nicht mehr sind. Dabei kann es sich um die tatsächliche Entfernung von Methylgruppen durch entsprechende Enzyme (Demethylasen) handeln, so dass die ursprünglichen Nukleobasen wieder hergestellt werden (Demethylierung in engerem Sinn) oder um eine Umwandlung der Methylgruppen in andere chemische Modifikationen (z. B. Umwandlung von 5-Methylcytosin in 5-Hydroxymethylcytosin, „Demethylierung“ in weiterem Sinn).
Modifikation und Mutation
Der Ausdruck „Modifikation“ wird in der Biologie mit verschiedenen Bedeutungen gebraucht. Von einer phänotypischen Modifikation spricht man, wenn sich die Eigenschaften eines Lebewesens durch geänderte Umweltbedingungen ändern (veränderter Phänotyp), ohne dass die Erbsubstanz in der Abfolge ihrer Grundbausteine verändert wurde (unveränderter Genotyp). Wird der Ausdruck „Modifikation“ ohne weitere Attribute verwendet, ist meist diese phänotypische Modifikation gemeint. Die DNA-Methylierung ist eine DNA-Modifikation und damit eine Modifikation von Makromolekülen. Die „DNA-Modifikation“ und die „phänotypische Modifikation“ sind verschiedene Begriffe, haben allerdings die Gemeinsamkeit, ohne Änderung der Abfolge der Grundbausteine der Desoxyribonukleinsäure zustande zu kommen. Die DNA-Modifikation und die phänotypische Modifikation sind daher keine Mutationen. DNA-Modifikationen können aber Mutationen nach sich ziehen. So steigt die Wahrscheinlichkeit, dass ein Cytosin zum Thymin umgewandelt wird (Punktmutation), wenn dieses Cytosin methyliert auftritt (siehe CpG-Stellen).
Modifikationen der DNA können auch phänotypische Modifikationen nach sich ziehen: Veränderte Umweltbedingungen führen über die Signaltransduktion zu einem veränderten Methylierungsmuster der DNA in bestimmten Bereichen (DNA-Modifikation); dadurch werden die Bedingungen für den Zugriff auf Gene verändert. Damit kann die Nutzung von Genen abgeändert werden (differenzielle Genexpression); dies zeigt sich in einer Änderung der Eigenschaften des Lebewesens (phänotypische Modifikation).
Eine Mutation ist per Definition vererbbar. DNA-Modifikationen sind es nicht oder nur unter gewissen Bedingungen. Ein Beispiel der teilweisen Vererbbarkeit von DNA-Modifikationen ist die genomische Prägung. Dabei findet durch DNA-Methylierung eine Unterscheidung zwischen väterlichem und mütterlichem Allel desselben Gens statt. Bei geprägten Genen ist nur ein Allel aktiv. Die dafür verantwortlichen DNA-Methylierungsmuster werden in die nächste Generation übertragen, also vererbt. Da die Grundbausteine (Nukleobasen) aber bei beiden Allelen erhalten bleiben und die Modifikation durch DNA-Demethylierung rückführbar ist, handelt sich bei der genomischen Prägung weder um Mutationen noch um genetische Vererbung.
Epigenetische Markierung
Je nachdem, welche Definition für Epigenetik angewendet wird, sind Methylierungen an den Nukleobasen der DNA epigenetisch.
Die gegenwärtig (2017) meist genutzten Definitionen beziehen sich auf Lebewesen mit Zellkern (Eukaryoten) und verwenden Begriffe wie Mitose, Meiose bzw. Chromosom; sie schließen dadurch die Lebewesen ohne Zellkern (Bakterien und Archaeen) nicht mit ein.[A 3][92][93] Von Forschern auf dem Gebiet bakterieller Epigenetik wurde vorgeschlagen, eine vorläufige Definition zu verwenden, die die Epigenetik als Untersuchung der Zelllinienbildung durch nicht-mutationale Mechanismen anspricht, solange keine allgemein akzeptierte Definition der Epigenetik vereinbart wurde.[26]
Den Begriff der Epigenetik prägte Conrad Hal Waddington. Anfang der 1940er Jahre definierte er Epigenetik als the branch of biology which studies the causal interactions between genes and their products which bring the phenotype into being – den Zweig der Biologie, der die kausalen Wechselwirkungen untersucht zwischen Genen und ihren Produkten, die den Phänotyp hervorbringen.[A 4][94][95] Nach dieser ursprünglichen und allgemeiner gehaltenen Definition wären auch bei Lebewesen ohne Zellkern die DNA-Methylierungen epigenetisch, die den jeweiligen Phänotyp beeinflussen.
Ohne Nachweis des jeweiligen Kausalzusammenhangs wird oft die Formulierung „epigenetische Markierung“ verwendet, um den Umstand zu beschreiben, dass Teile des Erbguts durch DNA-Methylierung oberhalb der genetischen Ebene markiert werden (altgr. ἐπί epi ‘auf’, ‘zusätzlich’, ‘außerdem’). In diesem Sinne sind die DNA-Methylierungen in allen Lebewesen, in denen sie auftreten, epigenetische Markierungen.
Literatur
- A. Jowaed, I. Schmitt, O. Kaut, U. Wüllner: Methylation regulates alpha-synuclein expression and is decreased in Parkinson's disease patients' brains. In: J. Neurosci. Band 30, Nr. 18, Mai 2010, S. 6355–6359, doi:10.1523/JNEUROSCI.6119-09.2010, PMID 20445061.
- Peter Weigele, Elisabeth A. Raleigh: Biosynthesis and Function of Modified Bases in Bacteria and Their Viruses. In: Chemical Reviews. Band 116, Nr. 20, 26. Oktober 2016, ISSN 1520-6890, S. 12655–12687, doi:10.1021/acs.chemrev.6b00114, PMID 27319741.
Anmerkungen
- Die dritte methylierte Form einer Nukleobase, N4-Cytosin, wurde von Ehrlich et al. (1985, PMID 4000939) in verschiedenen Prokaryoten gefunden. Zu dieser Zeit wurden sie generell als Bakterien bezeichnet und später den verschiedenen Domänen Bacteria und Archaea zugeordnet (Woese et al. 1990, PMID 2112744).
- Von Casadesús & Low (2013, PMID 23592777) wird "Waddington C. H. (1957) The Strategy of the Genes, George Allen and Unwin, London" zitiert; das Werk ist als "C. H. Waddington: The Strategy of the Genes. In: Routledge Library Editions: 20th Century Science, Band 20 (274 Seiten), 7. Mai 2014, ISBN 978-1-13801731-3 (Hardcopy)" erhältlich. Eine Vorschau (Inhaltsüberblick) läßt sich als GooglePreview herunterladen (Abruf 2019-10, https://content.taylorfrancis.com/books/download?dac=C2013-0-26528-7&isbn=9781317657552&format=googlePreviewPdf).
- Zu Mitose und Meiose wird in Egger et al. (2004, PMID 15164071) Folgendes angegeben: „The term epigenetics defines all meiotically and mitotically heritable changes in gene expression that are not coded in the DNA sequence itself.“ und zum Chromosom steht in Bird (2007, PMID 17522671) Folgendes: „... the structural adaptation of chromosomal regions so as to register, signal or perpetuate altered activity states.“
- Der Text mit dem Wortlaut ‘‘the branch of biology which studies the causal interactions between genes and their products which bring the phenotype into being” wird beispielsweise bei Urvalek et al. (2014, PMID 24962884) und bei Spinelli (2017, doi:10.21767/2472-1158.100077) Anfang der 1940er Jahre eingeordnet. Beide Arbeiten verweisen auf Waddington (1968, doi:10.1038/218525a0). Eine weitere, in diesem Zusammenhang aufgegriffene Quelle ist ein Nachdruck (2012, PMID 22186258) zu "Waddington CH (1942) The epigenotype. Endeavour 1: 18-20.", in welchem die Grundaussage des besagten Textes gefunden wird, wenngleich nicht mit dem genauen Wortlaut.
Einzelnachweise
- D. S. Shames, J. D. Minna, A. F. Gazdar: DNA methylation in health, disease, and cancer. In: Curr. Mol. Med. Band 7, Nr. 1, Februar 2007, S. 85–102, PMID 17311535.
- S. Beck, V. K. Rakyan: The methylome: approaches for global DNA methylation profiling. In: Trends Genet. Band 24, Nr. 5, Mai 2008, S. 231–237, doi:10.1016/j.tig.2008.01.006, PMID 18325624.
- L. Shen, R. A. Waterland: Methods of DNA methylation analysis. In: Curr Opin Clin Nutr Metab Care. Band 10, Nr. 5, September 2007, S. 576–581, doi:10.1097/MCO.0b013e3282bf6f43, PMID 17693740.
- B. A. Braaten, X. Nou, L. S. Kaltenbach, D. A. Low: Methylation patterns in pap regulatory DNA control pyelonephritis-associated pili phase variation in E. coli. In: Cell. Band 76, Nummer 3, Februar 1994, S. 577–588. PMID 7906204.
- Matthew J. Blow, Tyson A. Clark, Chris G. Daum, Adam M. Deutschbauer, Alexey Fomenkov, Roxanne Fries, Jeff Froula, Dongwan D. Kang, Rex R. Malmstrom, Richard D. Morgan, Janos Posfai, Kanwar Singh, Axel Visel, Kelly Wetmore, Zhiying Zhao, Edward M. Rubin, Jonas Korlach, Len A. Pennacchio, Richard J. Roberts: The Epigenomic Landscape of Prokaryotes. In: PLoS Genet. 12(2), Feb 2016, S. e1005854. doi:10.1371/journal.pgen.1005854. PMID 26870957. PMC 4752239 (freier Volltext).
- M. M. Pooggin: How can plant DNA viruses evade siRNA-directed DNA methylation and silencing? In: International journal of molecular sciences. Band 14, Nummer 8, Juli 2013, S. 15233–15259, doi:10.3390/ijms140815233, PMID 23887650, PMC 3759858 (freier Volltext) (Review).
- S. Mukherjee, V. C. Vipat, A. K. Chakrabarti: Infection with influenza A viruses causes changes in promoter DNA methylation of inflammatory genes. In: Influenza and other respiratory viruses. Band 7, Nummer 6, November 2013, S. 979–986, doi:10.1111/irv.12127, PMID 23758996, PMC 4634256 (freier Volltext).
- J. E. Shin, C. Lin, H. N. Lim: Horizontal transfer of DNA methylation patterns into bacterial chromosomes. In: Nucleic acids research. Band 44, Nummer 9, 05 2016, S. 4460–4471, doi:10.1093/nar/gkw230, PMID 27084942, PMC 4872104 (freier Volltext).
- D. B. Dunn, J. D. Smith: The occurrence of 6-methylaminopurine in deoxyribonucleic acids. In: Biochem J. 68(4), Apr 1958, S. 627–636. PMID 13522672. PMC 1200409 (freier Volltext).
- B. F. Vanyushin, S. G. Tkacheva, A. N. Belozersky: Rare bases in animal DNA. In: Nature. 225, 1970, S. 948–949. PMID 4391887.
- Melanie Ehrlich, Miguel A. Gama-Sosa, Laura H. Carreira, Lars G. Ljungdahl, Kenneth C. Kuo, Charles W. Gehrke: DNA methylation in thermophilic bacteria: N6-methylcytosine, 5-methylcytosine, and N6-methyladenine. In: Nucleic Acids Research. 13, 1985, S. 1399. PMID 4000939. PMC 341080 (freier Volltext).
- S. Hattman, C. Kenny, L. Berger, K. Pratt: Comparative study of DNA methylation in three unicellular eucaryotes. In: J Bacteriol. 135(3), Sep 1978, S. 1156–1157. PMID 99431. PMC 222496 (freier Volltext).
- D. Ratel, J.-L. Ravanat, M.-P. Charles, N. Platet, L. Breuillaud, J. Lunardi, F. Berger, D. Wion: Undetectable levels of N6-methyl adenine in mouse DNA: Cloning and analysis of PRED28, a gene coding for a putative mammalian DNA adenine methyltransferase. In: FEBS Lett. 580, 2006, S. 3179–3184. PMID 16684535. doi:10.1016/j.febslet.2006.04.074.
- Eric Lieberman Greer, Mario Andres Blanco, Lei Gu, Erdem Sendinc, Jianzhao Liu: DNA Methylation on N6-Adenine in C. elegans. In: Cell. Band 161, Nr. 4, 7. Mai 2015, ISSN 1097-4172, S. 868–878, doi:10.1016/j.cell.2015.04.005, PMID 25936839, PMC 4427530 (freier Volltext).
- Guoqiang Zhang, Hua Huang, Di Liu, Ying Cheng, Xiaoling Liu: N6-methyladenine DNA modification in Drosophila. In: Cell. Band 161, Nr. 4, 7. Mai 2015, ISSN 1097-4172, S. 893–906, doi:10.1016/j.cell.2015.04.018, PMID 25936838.
- F. Capuano, M. Mulleder, R. Kok, H. J. Blom, M. Ralser: Cytosine DNA Methylation Is Found in Drosophila melanogaster but Absent in Saccharomyces cerevisiae, Schizosaccharomyces pombe, and Other Yeast Species. In: Analytical Chemistry. 86, 2014, S. 3697–3702. doi:10.1021/ac500447w. PMID 24640988. PMC PMC4006885 (freier Volltext).
- Guan-Zheng Luo, Mario Andres Blanco, Eric Lieberman Greer, Chuan He, Yang Shi: DNA N6-methyladenine: a new epigenetic mark in eukaryotes? In: Nature Reviews Molecular Cell Biology. 16, 2015, S. 705, doi:10.1038/nrm4076. PMID 26507168. PMC 4763336 (freier Volltext).
- J. Casadesus, D. Low: Epigenetic gene regulation in the bacterial world. In: Microbiology and molecular biology reviews: MMBR. Band 70, Nummer 3, September 2006, S. 830–856, doi:10.1128/MMBR.00016-06. PMID 16959970, PMC 1594586 (freier Volltext).
- D. Wion, J. Casadesus: N6-methyl-adenine: an epigenetic signal for DNA-protein interactions. In: Nature reviews. Microbiology. Band 4, Nummer 3, März 2006, S. 183–192, doi:10.1038/nrmicro1350. PMID 16489347, PMC 2755769 (freier Volltext) (Review).
- R. Kumar, D. N. Rao: Role of DNA methyltransferases in epigenetic regulation in bacteria. In: Epigenetics: Development and Disease. (= Subcellular Biochemistry. 61). 2013, S. 81–102. doi:10.1007/978-94-007-4525-4_4. PMID 23150247.
- J. Casadesus, D. A. Low: Programmed heterogeneity: epigenetic mechanisms in bacteria. In: J Biol Chem. 288(20), 17. Mai 2013, S. 13929–13935. doi:10.1074/jbc.R113.472274. PMID 23592777. PMC 3656251 (freier Volltext).
- B. M. Forde, M. D. Phan, J. A. Gawthorne, M. M. Ashcroft, M. Stanton-Cook, S. Sarkar, K. M. Peters, K. G. Chan, T. M. Chong, W. F. Yin, M. Upton, M. A. Schembri, S. A. Beatson: Lineage-Specific Methyltransferases Define the Methylome of the Globally Disseminated Escherichia coli ST131 Clone. In: mBio. Band 6, Nummer 6, November 2015, S. e01602–e01615, doi:10.1128/mBio.01602-15. PMID 26578678, PMC 4659465 (freier Volltext).
- Robson Ee, Yan-Lue Lim, Wai-Fong Yin, Wah-Seng See-Too, Richard J. Roberts, Kok-Gan Chan: Novel Methyltransferase Recognition Motif Identified in Chania multitudinisentens RB-25T gen. nov., sp. nov. In: Frontiers in Microbiology. 7, 2016, doi:10.3389/fmicb.2016.01362.
- F. Barras, M. G. Marinus: The great GATC: DNA methylation in E. coli. In: Trends Genet. Band 5, Nr. 5, 1989, S. 139–143. PMID 2667217.
- M. G. Marinus, J. Casadesus: Roles of DNA adenine methylation in host-pathogen interactions: mismatch repair, transcriptional regulation, and more. In: FEMS Microbiol Rev. Band 33, Nr. 3, 2009, S. 488–503. PMID 19175412, doi:10.1111/j.1574-6976.2008.00159.x.
- J. Casadesús, D. A. Low: Programmed heterogeneity: epigenetic mechanisms in bacteria. In: The Journal of Biological Chemistry. Band 288, Nummer 20, Mai 2013, S. 13929–13935, doi:10.1074/jbc.R113.472274, PMID 23592777, PMC 3656251 (freier Volltext) (Review).
- J. Errington: Regulation of endospore formation in Bacillus subtilis. In: Nature reviews. Microbiology. Band 1, Nummer 2, November 2003, S. 117–126, doi:10.1038/nrmicro750, PMID 15035041 (Review).
- K. E. Gibson, H. Kobayashi, G. C. Walker: Molecular determinants of a symbiotic chronic infection. In: Annual Review of Genetics. Band 42, 2008, S. 413–441, doi:10.1146/annurev.genet.42.110807.091427, PMID 18983260, PMC 2770587 (freier Volltext) (Review).
- C. L. Kirkpatrick, P. H. Viollier: Decoding Caulobacter development. In: FEMS Microbiology Reviews. Band 36, Nummer 1, Januar 2012, S. 193–205, doi:10.1111/j.1574-6976.2011.00309.x, PMID 22091823 (Review).
- D. Kaiser: Myxococcus-from single-cell polarity to complex multicellular patterns. In: Annual Review of Genetics. Band 42, 2008, S. 109–130, doi:10.1146/annurev.genet.42.110807.091615, PMID 18605899 (Review).
- E. Flores, A. Herrero: Compartmentalized function through cell differentiation in filamentous cyanobacteria. In: Nature Reviews. Microbiology. Band 8, Nummer 1, Januar 2010, S. 39–50, doi:10.1038/nrmicro2242, PMID 19966815 (Review).
- P. S. Stewart, M. J. Franklin: Physiological heterogeneity in biofilms. In: Nature Reviews Microbiology. Band 6, Nummer 3, März 2008, S. 199–210, doi:10.1038/nrmicro1838, PMID 18264116 (Review).
- Y. Chai, F. Chu, R. Kolter, R. Losick: Bistability and biofilm formation in Bacillus subtilis. In: Molecular Microbiology. Band 67, Nummer 2, Januar 2008, S. 254–263, doi:10.1111/j.1365-2958.2007.06040.x, PMID 18047568, PMC 2430929 (freier Volltext).
- C. H. Waddington: The Strategy of the Genes. Verlag: George Allen and Unwin, London.
- L. B. Blyn, B. A. Braaten, D. A. Low: Regulation of pap pilin phase variation by a mechanism involving differential dam methylation states. In: The EMBO Journal. Band 9, Nummer 12, Dezember 1990, S. 4045–4054, PMID 2147413, PMC 552177 (freier Volltext).
- M. X. Wang, G. M. Church: A whole genome approach to in vivo DNA-protein interactions in E. coli. In: Nature. Band 360, Nummer 6404, Dezember 1992, S. 606–610, doi:10.1038/360606a0, PMID 1334233.
- M. van der Woude, W. B. Hale, D. A. Low: Formation of DNA methylation patterns: nonmethylated GATC sequences in gut and pap operons. In: Journal of Bacteriology. Band 180, Nummer 22, November 1998, S. 5913–5920, PMID 9811649, PMC 107665 (freier Volltext).
- D. E. Waldron, P. Owen, C. J. Dorman: Competitive interaction of the OxyR DNA-binding protein and the Dam methylase at the antigen 43 gene regulatory region in Escherichia coli. In: Molecular Microbiology. Band 44, Nummer 2, April 2002, S. 509–520, PMID 11972787.
- S. Ringquist, C. L. Smith: The Escherichia coli chromosome contains specific, unmethylated dam and dcm sites. In: Proceedings of the National Academy of Sciences. Band 89, Nummer 10, Mai 1992, S. 4539–4543, PMID 1584789, PMC 49118 (freier Volltext).
- Olivier Mathieu, Georges Picard, Sylvette Tourmente: Methylation of a euchromatin-heterochromatin transition region in Arabidopsis thaliana chromosome 5 left arm. In: Chromosome Research: An International Journal on the Molecular, Supramolecular and Evolutionary Aspects of Chromosome Biology. Band 10, Nr. 6, 2002, ISSN 0967-3849, S. 455–466, PMID 12489828.
- M. Gardiner-Garden, M. Frommer: CpG islands in vertebrate genomes. In: Journal of molecular biology. Band 196, Nummer 2, Juli 1987, S. 261–282. PMID 3656447.
- G. D. Ginder, D. C. Williams: Readers of DNA methylation, the MBD family as potential therapeutic targets. In: Pharmacology & therapeutics. [elektronische Veröffentlichung vor dem Druck] November 2017, doi:10.1016/j.pharmthera.2017.11.002. PMID 29128342 (Review).
- P. D. Fransquet, P. Lacaze, R. Saffery, J. McNeil, R. Woods, J. Ryan: Blood DNA methylation as a potential biomarker of dementia: A systematic review. In: Alzheimer's & dementia : the journal of the Alzheimer's Association. [elektronische Veröffentlichung vor dem Druck] November 2017, doi:10.1016/j.jalz.2017.10.002. PMID 29127806 (Review).
- F. Capuano, M. Mülleder, R. Kok, H. J. Blom, M. Ralser: Cytosine DNA methylation is found in Drosophila melanogaster but absent in Saccharomyces cerevisiae, Schizosaccharomyces pombe, and other yeast species. In: Analytical chemistry. Band 86, Nummer 8, April 2014, S. 3697–3702, doi:10.1021/ac500447w. PMID 24640988, PMC 4006885 (freier Volltext).
- X. Cao, S. E. Jacobsen: Locus-specific control of asymmetric and CpNpG methylation by the DRM and CMT3 methyltransferase genes. In: Proceedings of the National Academy of Sciences. Band 99 Suppl 4, Dezember 2002, S. 16491–16498, doi:10.1073/pnas.162371599. PMID 12151602, PMC 139913 (freier Volltext).
- W. Aufsatz, M. F. Mette, J. van der Winden, A. J. Matzke, M. Matzke: RNA-directed DNA methylation in Arabidopsis. In: Proceedings of the National Academy of Sciences. Band 99 Suppl 4, Dezember 2002, S. 16499–16506, doi:10.1073/pnas.162371499. PMID 12169664, PMC 139914 (freier Volltext).
- Y. Wang, M. Jorda, P. L. Jones, R. Maleszka, X. Ling, H. M. Robertson, C. A. Mizzen, M. A. Peinado, G. E. Robinson: Functional CpG methylation system in a social insect. In: Science. Band 314, Nummer 5799, Oktober 2006, S. 645–647, doi:10.1126/science.1135213. PMID 17068262.
- Y. Wang, H. Li-Byarlay: Physiological and Molecular Mechanisms of Nutrition in Honey Bees. In: Advances in Insect Physiology. Band 49, Kapitel 2, August 2015, S. 25–85, doi:10.1016/bs.aiip.2015.06.002.
- H. Li-Byarlay, Y. Li, H. Stroud, S. Feng, T. C. Newman, M. Kaneda, K. K. Hou, K. C. Worley, C. G. Elsik, S. A. Wickline, S. E. Jacobsen, J. Ma, G. E. Robinson: RNA interference knockdown of DNA methyl-transferase 3 affects gene alternative splicing in the honey bee. In: Proceedings of the National Academy of Sciences. Band 110, Nummer 31, Juli 2013, S. 12750–12755, doi:10.1073/pnas.1310735110. PMID 23852726, PMC 3732956 (freier Volltext).
- S. S. Smith, C. A. Thomas: The two-dimensional restriction analysis of Drosophila DNAs: males and females. In: Gene. Band 13, Nummer 4, Mai 1981, S. 395–408. PMID 6266924.
- F. Lyko, B. H. Ramsahoye, R. Jaenisch: DNA methylation in Drosophila melanogaster. In: Nature. Band 408, Nummer 6812, November 2000, S. 538–540, doi:10.1038/35046205. PMID 11117732.
- S. Takayama, J. Dhahbi, A. Roberts, G. Mao, S. J. Heo, L. Pachter, D. I. Martin, D. Boffelli: Genome methylation in D. melanogaster is found at specific short motifs and is independent of DNMT2 activity. In: Genome research. Band 24, Nummer 5, Mai 2014, S. 821–830, doi:10.1101/gr.162412.113. PMID 24558263, PMC 4009611 (freier Volltext).
- G. Zhang, H. Huang, D. Liu, Y. Cheng, X. Liu, W. Zhang, R. Yin, D. Zhang, P. Zhang, J. Liu, C. Li, B. Liu, Y. Luo, Y. Zhu, N. Zhang, S. He, C. He, H. Wang, D. Chen: N6-methyladenine DNA modification in Drosophila. In: Cell. Band 161, Nummer 4, Mai 2015, S. 893–906, doi:10.1016/j.cell.2015.04.018. PMID 25936838.
- F. Antequera, M. Tamame, J. R. Villanueva, T. Santos: DNA methylation in the fungi. In: The Journal of biological chemistry. Band 259, Nummer 13, Juli 1984, S. 8033–8036. PMID 6330093.
- Thomas Binz, Nisha D'Mello, Paul A. Horgen: A Comparison of DNA Methylation Levels in Selected Isolates of Higher Fungi. In: Mycologia. 90, 1998, S. 785, doi:10.2307/3761319.
- S. Y. Liu, J. Q. Lin, H. L. Wu, C. C. Wang, S. J. Huang, Y. F. Luo, J. H. Sun, J. X. Zhou, S. J. Yan, J. G. He, J. Wang, Z. M. He: Bisulfite sequencing reveals that Aspergillus flavus holds a hollow in DNA methylation. In: PloS one. Band 7, Nummer 1, 2012, S. e30349, doi:10.1371/journal.pone.0030349. PMID 22276181, PMC 3262820 (freier Volltext).
- E. U. Selker, N. A. Tountas, S. H. Cross, B. S. Margolin, J. G. Murphy, A. P. Bird, M. Freitag: The methylated component of the Neurospora crassa genome. In: Nature. Band 422, Nummer 6934, April 2003, S. 893–897, doi:10.1038/nature01564. PMID 12712205.
- S. S. Smith, D. I. Ratner: Lack of 5-methylcytosine in Dictyostelium discoideum DNA. In: The Biochemical journal. Band 277 ( Pt 1), Juli 1991, S. 273–275. PMID 1713034, PMC 1151219 (freier Volltext).
- J. L. Steenwyk, J. St-Denis, J. Dresch, D. Larochelle, R. A. Drewell: Whole genome bisulfite sequencing reveals a sparse, but robust pattern of DNA methylation in the Dictyostelium discoideum genome. In: bioRxiv. 2017, 166033. doi:10.1101/166033.
- J. G. Reilly, R. Braun, C. A. Thomas: Methjylation in Physarum DNA. In: FEBS letters. Band 116, Nummer 2, Juli 1980, S. 181–184. PMID 6250882.
- H. H. Evans, T. E. Evans: Methylation of the deoxyribonucleic acid of Physarum polycephalum at various periods during the mitotic cycle. In: The Journal of biological chemistry. Band 245, Nummer 23, Dezember 1970, S. 6436–6441. PMID 5530731.
- A. Jeltsch: Beyond Watson and Crick: DNA methylation and molecular enzymology of DNA methyltransferases. In: ChemBioChem Band 3, Nr. 4, 2002, S. 274–293. PMID 11933228.
- A. Jeltsch: Molecular enzymology of mammalian DNA methyltransferases. In: Curr Top Microbiol Immunol. Band 301, 2006, S. 203–225. PMID 16570849.
- S. Seisenberger, J. R. Peat, T. A. Hore, F. Santos, W. Dean, W. Reik: Reprogramming DNA methylation in the mammalian life cycle: building and breaking epigenetic barriers. In: Philosophical transactions of the Royal Society of London. Series B, Biological sciences. Band 368, Nummer 1609, Januar 2013, S. 20110330, doi:10.1098/rstb.2011.0330. PMID 23166394. PMC 3539359 (freier Volltext).
- M. J. Snider, L. Reinhardt, R. Wolfenden, W. W. Cleland: 15N kinetic isotope effects on uncatalyzed and enzymatic deamination of cytidine. In: Biochemistry. Band 41, Nummer 1, Januar 2002, S. 415–421, PMID 11772041.
- M. J. Snider, R. Wolfenden: Site-bound water and the shortcomings of a less than perfect transition state analogue. In: Biochemistry. Band 40, Nummer 38, September 2001, S. 11364–11371, PMID 11560484.
- Vincent Caval, Rodolphe Suspène, Jean-Pierre Vartanian, Simon Wain-Hobson: Orthologous Mammalian APOBEC3A Cytidine Deaminases Hypermutate Nuclear DNA. In: Molecular Biology and Evolution. 31, 2014, S. 330, doi:10.1093/molbev/mst195.
- N. Schormann, R. Ricciardi, D. Chattopadhyay: Uracil-DNA glycosylases-structural and functional perspectives on an essential family of DNA repair enzymes. In: Protein science : a publication of the Protein Society. Band 23, Nummer 12, Dezember 2014, S. 1667–1685, doi:10.1002/pro.2554, PMID 25252105, PMC 4253808 (freier Volltext) (Review).
- X. Nan, R. Meehan, A. Bird: Dissection of the methyl-CpG binding domain from the chromosomal protein MeCP2. In: Nucleic Acids Research. Band 21, Nr. 21, 25 Oktober 1993, S. 4886–4892; DOI:10.1093/nar/21.21.4886, PMID 8177735, PMC 311401 (freier Volltext).
- Q. Du, P. Luu, C. Stirzaker, S. Clark: Methyl-CpG-binding domain proteins: readers of the epigenome In: Epigenomics. Band 7, Nr. 6, 30. April 2015, S. 1051–1073; DOI:10.2217/epi.15.39, PMID 25927341.
- N. Cervoni, S. Bhattacharya, M. Szyf: DNA demethylase is a processive enzyme. In: The Journal of Biological Chemistry. Band 274, Nr. 13, 26. März 1999, ISSN 0021-9258, S. 8363–8366, doi:10.1074/jbc.274.13.8363, PMID 10085064.
- H. Cedar, Y. Bergman: Programming of DNA methylation patterns. In: Annual review of biochemistry. Band 81, 2012, S. 97–117, doi:10.1146/annurev-biochem-052610-091920, PMID 22404632 (Review).
- C. Beard, E. Li, R. Jaenisch: Loss of methylation activates Xist in somatic but not in embryonic cells. In: Genes & development. Band 9, Nummer 19, Oktober 1995, S. 2325–2334, PMID 7557385.
- E. Li, C. Beard, R. Jaenisch: Role for DNA methylation in genomic imprinting. In: Nature. Band 366, Nummer 6453, November 1993, S. 362–365, doi:10.1038/366362a0, PMID 8247133.
- Entrez Gene: XIST X (inactive)-specific transcript
- J. Borgel, S. Guibert, Y. Li, H. Chiba, D. Schübeler, H. Sasaki, T. Forné, M. Weber: Targets and dynamics of promoter DNA methylation during early mouse development. In: Nature Genetics. 42, 2010, S. 1093, doi:10.1038/ng.708.
- S. Seisenberger, J. R. Peat, T. A. Hore, F. Santos, W. Dean, W. Reik: Reprogramming DNA methylation in the mammalian life cycle: building and breaking epigenetic barriers. In: Philosophical transactions of the Royal Society of London. Series B, Biological sciences. Band 368, Nummer 1609, Januar 2013, S. 20110330, doi:10.1098/rstb.2011.0330, PMID 23166394, PMC 3539359 (freier Volltext) (Review).
- M. J. Ziller, F. Müller, J. Liao, Y. Zhang, H. Gu, C. Bock, P. Boyle, C. B. Epstein, B. E. Bernstein, T. Lengauer, A. Gnirke, A. Meissner: Genomic distribution and inter-sample variation of non-CpG methylation across human cell types. In: PLoS genetics. Band 7, Nummer 12, Dezember 2011, S. e1002389, doi:10.1371/journal.pgen.1002389, PMID 22174693, PMC 3234221 (freier Volltext).
- M. Fasolino, Z. Zhou: The Crucial Role of DNA Methylation and MeCP2 in Neuronal Function. In: Genes. Band 8, Nummer 5, Mai 2017, S. , doi:10.3390/genes8050141, PMID 28505093, PMC 5448015 (freier Volltext) (Review).
- C. G. Duke, A. J. Kennedy, C. F. Gavin, J. J. Day, J. D. Sweatt: Experience-dependent epigenomic reorganization in the hippocampus. In: Learning & memory. Band 24, Nummer 7, 07 2017, S. 278–288, doi:10.1101/lm.045112.117, PMID 28620075, PMC 5473107 (freier Volltext).
- R. Halder, M. Hennion, R. O. Vidal, O. Shomroni, R. U. Rahman, A. Rajput, T. P. Centeno, F. van Bebber, V. Capece, J. C. Garcia Vizcaino, A. L. Schuetz, S. Burkhardt, E. Benito, M. Navarro Sala, S. B. Javan, C. Haass, B. Schmid, A. Fischer, S. Bonn: DNA methylation changes in plasticity genes accompany the formation and maintenance of memory. In: Nature Neuroscience. Band 19, Nummer 1, Januar 2016, S. 102–110, doi:10.1038/nn.4194, PMID 26656643.
- H. Heyn, N. Li u. a.: Distinct DNA methylomes of newborns and centenarians. In: Proceedings of the National Academy of Sciences. Band 109, Nummer 26, Juni 2012, S. 10522–10527. doi:10.1073/pnas.1120658109. PMID 22689993.
- A. A. Johnson, K. Akman, S. R. Calimport, D. Wuttke, A. Stolzing, J. P. de Magalhães: The role of DNA methylation in aging, rejuvenation, and age-related disease. In: Rejuvenation research. Band 15, Nummer 5, Oktober 2012, S. 483–494, doi:10.1089/rej.2012.1324. PMID 23098078. PMC 3482848 (freier Volltext).
- J. A. Hackett, M. A. Surani: DNA methylation dynamics during the mammalian life cycle. In: Philosophical transactions of the Royal Society of London. Series B, Biological sciences. Band 368, Nummer 1609, Januar 2013, S. 20110328, doi:10.1098/rstb.2011.0328. PMID 23166392. PMC 3539357 (freier Volltext).
- M. Berdasco, M. Esteller: DNA methylation in stem cell renewal and multipotency. In: Stem cell research & therapy. Band 2, Nummer 5, 2011, S. 42, doi:10.1186/scrt83. PMID 22041459. PMC 3308039 (freier Volltext).
- Keh-Yang Wang, Chun-Chang Chen, Shih-Feng Tsai, Che-Kun James Shen: Epigenetic Enhancement of the Post-replicative DNA Mismatch Repair of Mammalian Genomes by a Hemi-mCpG-Np95-Dnmt1 Axis. In: Scientific Reports. 6, 2016, S. 37490, doi:10.1038/srep37490. PMC 5122852 (freier Volltext). PMID 27886214.
- E. Daura-Oller, M. Cabre, M. A. Montero, J. L. Paternain, A. Romeu: Specific gene hypomethylation and cancer: new insights into coding region feature trends. In: Bioinformation. Band 3, Nr. 8, 2009, S. 340–343, PMID 19707296, PMC 2720671 (freier Volltext).
- R. L. Momparler, V. Bovenzi: DNA methylation and cancer. In: J. Cell Physiol. Band 183, Nr. 2, 2000, S. 145–154. PMID 10737890.
- Myelodysplastische Syndrome (MDS). Abgerufen am 5. Dezember 2020.
- P. V. Wijermans u. a.: An epigenetic approach to the treatment of advanced MDS; the experience with the DNA demethylating agent 5-aza-2'-deoxycytidine (decitabine) in 177 patients. In: Ann. Hematol. Band 84, Nr. l, 2005, S. 9–17. PMID 16211386, doi:10.1007/s00277-005-0012-1.
- Dacogen-Freigabe durch die FDA.
- Gerda Egger, Gangning Liang, Ana Aparicio, Peter A. Jones: Epigenetics in human disease and prospects for epigenetic therapy. In: Nature. Band 429, Nr. 6990, 27. Mai 2004, ISSN 1476-4687, S. 457–463, doi:10.1038/nature02625, PMID 15164071.
- Adrian Bird: Perceptions of epigenetics. In: Nature. Band 447, Nr. 7143, 24. Mai 2007, ISSN 1476-4687, S. 396–398, doi:10.1038/nature05913, PMID 17522671.
- C. H. Waddington: Towards a Theoretical Biology. In: Nature. Band 218, Nr. 5141, Mai 1968, ISSN 0028-0836, S. 525–527, doi:10.1038/218525a0.
- C. H. Waddington: The epigenotype. 1942. In: International Journal of Epidemiology. Band 41, Nr. 1, Februar 2012, ISSN 1464-3685, S. 10–13, doi:10.1093/ije/dyr184, PMID 22186258.