Histon
Histone sind basische Proteine, die im Zellkern von Eukaryoten vorkommen wie außerdem in bestimmten Archaeen, insbesondere Euryarchaeota und Proteoarchaeota.[1]
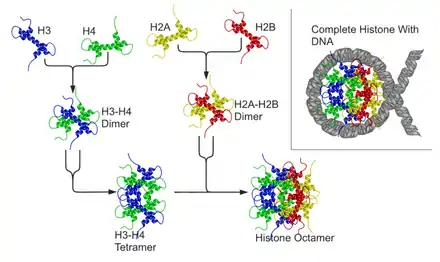
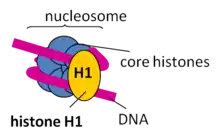
Als Bestandteil des Chromatins sind Histone von essentieller Bedeutung für die Verpackung der DNA und auch für die Expression mancher auf ihr codierten Gene (siehe Epigenetik). Das große Genom im Zellkern von eukaryotischen Zellen ist in Chromosomen aufgeteilt, deren kleinste Verpackungseinheiten Nukleosomen sind. Ein Nukleosom ähnelt einer Spule, bei der sich der DNA-Strang um einen Proteinkern wickelt, der aus Histonen besteht. Je zwei Kopien der Histone H2A, H2B, H3 und H4 bilden gemeinsam einen Proteinkomplex aus acht Histonen. Um dieses Histonoktamer ist die DNA gewickelt, etwa anderthalbmal (1,65-mal), auf einer Länge von 146 DNA-Basenpaaren. Die zwischen benachbarten Nukleosomen verbindende DNA wird Linker-DNA genannt. Ein weiteres Histon, H1, bindet DNA direkt neben Nukleosomen und erlaubt die nächsthöhere Verpackungseinheit der DNA.
Histone bestehen aus einem globulären Zentrum und flexiblen endständigen Armen (englisch histone tails), die zahlreiche basische, positiv geladene Aminosäuren aufweisen. Die DNA ist hingegen negativ geladen, so dass eine elektrostatische Anziehung besteht.
Entdeckung
Die Histon-Proteine wurden 1884 vom deutschen Mediziner und Physiologen Albrecht Kossel entdeckt. Der Begriff Histon lässt sich aus griechisch histanai oder histos herleiten. Bis in die frühen 1990er Jahre wurden Histone als reines Packmaterial nukleärer DNA verkannt. Erst in den letzten beiden Jahrzehnten konnte ihre Bedeutung für epigenetische Mechanismen beschrieben werden.[2]
Histonklassen
Beim Menschen sind fünf Haupt-Histon-Proteine (Histone-Klassen, englisch histone families) bekannt:
| Überfamilie superfamily |
Familie family |
Unterfamilie subfamily |
|---|---|---|
| Linker (Gerüst) |
H1 | H1F |
| H1H1 | ||
| Core (Spule) |
H2A | H2AF |
| H2A1 | ||
| H2A2 | ||
| H2B | H2BF | |
| H2B1 | ||
| H2B2 | ||
| H3 | H3A1 | |
| H3A2 | ||
| H3A3 | ||
| H4 | H41 | |
| H44 |
Die Histonproteine H2A und H2B lagern sich zu Dimeren zusammen. Dasselbe gilt für H3 und H4. Zwei H3/H4-Dimere lagern sich zu einem Tetramer zusammen, an das wiederum zwei H2A/H2B-Dimere angelagert werden. Dadurch entsteht der oktamere Nukleosomenkern (englisch core particle), um den sich die DNA in ca. zwei großen linksgängigen Windungen legen kann. Das fünfte Histon, H1, wird möglicherweise benötigt, um eine 30-nm-Faser zu bilden – eine übergeordnete Struktur, die einer Helix aus Nukleosomen entspricht. Dadurch wird die DNA Packung weiter verstärkt. Die Komprimierung des DNA-Moleküls beträgt ohne H1 Faktor 7 und wird mit H1 auf Faktor 40–50 erhöht, d. h. ein unkomprimierter DNA-Strang enthält 3 Mio. Nukleotide/mm, komprimiert ohne H1 20 Mio. Nukleotide/mm und mit H1 120 Mio. Nukleotide/mm.
Neben den oben genannten Histonproteinen gibt es außerdem Varianten, die sehr spezifische Funktionen bei der Regulation der Genexpression und der Strukturierung der Chromosomen übernehmen. Ein Beispiel ist Makro-H2A, welches das Histon H2A partiell auf dem inaktivierten X-Chromosom von weiblichen Säugern ersetzt. Ein anderes Beispiel ist CENP-A, eine Variante des Histons H3, die nur im Bereich des Zentromers zu finden ist und für die spezifische Struktur dieser Chromosomenregion essentiell ist. Im Großen und Ganzen sind die Core-Histone H2A, H2B, H3 und H4 in der Evolution streng konserviert worden. Lediglich das Histon H1 ist in seiner Struktur sehr variabel, wie der Vergleich unterschiedlicher Organismen zeigt. Bei der Bäckerhefe (Saccharomyces cerevisiae) fehlt dieses Histon sogar völlig. Die Bindung der DNA an die Histone kann die Transkription positiv oder negativ beeinflussen. Für die Vorgänge der Transkription, Replikation und DNA-Reparatur müssen die Histone von der DNA gelöst oder auf dem DNA-Strang verschoben werden – ein Vorgang, den man als Nukleosomenremodelling bezeichnet.[3]
Die Histone der Archaeen enthalten nur eine H3-H4-ähnliche dimere Struktur, die aus demselben Protein besteht. Solche dimeren Strukturen können sich zu einer großen Superhelix (‚Supernukleosom‘) stapeln, auf der sich die DNA ähnlich wie bei den Spulen der Nukleosomen aufwickelt.[4] Nur einige archaeale Histone haben endständige Arme.[5]
Histonmodifikationen
Das N-terminale Ende eines Histons kann von Enzymen modifiziert werden. Diese Histonmodifikationen können Methylierung, Phosphorylierung, Sumoylierung, Ubiquitinylierung, Acetylierung, Propionylierung und Butyrylierung sowie deren Rückreaktionen umfassen. Hieraus ergibt sich der spezifische Histon-Code einer Zelle. Diese Modifikationen haben Einfluss auf das Chromatingerüst des Zellkerns und somit auf die Genregulation.[6][7][8][9][10][11]
Die Funktion der Methylierung von Histonen wird derzeit intensiv erforscht und steht überwiegend in Beziehung zur epigenetischen Inaktivierung von Genen. So kann eine regionale Trimethylierung des Lysinseitenrestes (K9) am Histon 3 eines Promoters zu einer Kondensierung der Chromatinstruktur in diesem Bereich führen, dies hat dann eine Inaktivierung der Genexpression des auf diesem Abschnitt liegenden Gens zur Folge. Darüber hinaus existieren Verbindungen zum inaktivierenden Prozess der DNA-Methylierung. Die Phosphorylierung von Histonproteinen erhöht in den meisten Fällen die Zugänglichkeit der DNA und spielt unter anderem eine wichtige Rolle bei der Regulation der Transkription während der Mitose und Meiose. Die Acetylierung setzt, wie die Phosphorylierung, in den meisten Fällen die Bindefähigkeit der Histone für die DNA herab, indem die Ladung der Histone negativ wird und sich die negative DNA abstößt. Sie ist deshalb Voraussetzung für die Transkription der mit Histonen assoziierten DNA.[12][13]
Histongene
Histone sind offensichtlich evolutionär sehr alte und sehr bedeutende Moleküle, sodass sie hoch konserviert sind. Sie gehören zu den am stärksten konservierten Proteinen in Eukaryoten, was ihre wichtige Rolle in der Biologie des Zellkerns unterstreicht.[14]:939 Histongene sind S-Phase-abhängig, werden also nur exprimiert, wenn neue DNA gebildet wird, und zwar dann extrem stark.
Die Histongene liegen ähnlich wie die rDNA häufig in Clustern wiederholt vor, sind also gekoppelt, sie besitzen jedoch jeweils einen eigenen Promotor. Die Zahl der Wiederholungen kann recht stark schwanken (Saccharomyces cerevisiae hat 2 Cluster, der Grüne Wassermolch hat 700, beim Menschen sind es 10–24).
Die Reihenfolge der einzelnen Histongene im Cluster ist recht unterschiedlich, teilweise sind die Gene zudem gegenläufig, teilweise auch in gleicher Richtung angeordnet. In jedem Cluster jedoch sind alle 5 Histone vorhanden.
Die Histongene selbst haben zudem eine regelrecht altertümliche Struktur. Sie besitzen weder Introns, noch erhalten sie nach der Transkription einen poly(A)-Schwanz. Der 3'-UTR ist sehr kurz und enthält zwei gegenläufig gerichtete Wiederholungseinheiten (inverted repeats). Diese bilden eine Haarnadelstruktur aus, was man ansonsten nur von Genen von Prokaryoten kennt. Die Haarnadelstruktur ist für die koordinierte Reifung der Histon-mRNA und für die Regelung ihrer Lebenszeit in der S-Phase wichtig.
Bei Säugetieren und Vögeln sind die Cluster nicht repetitiv angeordnet, sondern etwas verteilt. Einige Histongene sind S-Phase-korreliert und liegen recht nahe beieinander. Sie entsprechen vom Aufbau her den „normalen“ Histongenen. Andere hingegen sind sogenannte Ersatzhistongene, die defekte Histonproteine ersetzen können, auch außerhalb der S-Phase. Diese liegen abseits der Histoncluster und besitzen lange 3'-UTRs und einen poly(A)-Schwanz.
Entwicklungsgeschichte der Histone
Wie bereits erwähnt, finden sich Histone in den Kernen eukaryotischer Zellen und in bestimmten Archaeen – bei Proteoarchaea und Euryarchaea, (bis auf H1, s. u.) aber nicht in Bakterien.[1] Die bei Archaeen gefundenen Histone scheinen den evolutionären Vorläufern eukaryotischer Histone sehr ähnlich sein.[1] Diese Ergebnisse stützen moderne Varianten der Eozyten-Theorie (die Ursprünge der Eukaryoten sind unter den Proteoarchaeota zu suchen), und damit einen zentralen Aspekt der Endosymbiontentheorie.
Im Gegensatz dazu finden in reife Samenzellen hauptsächlich Protamine Verwendung, um Kern-DNA zu packen. Dies liegt höchstwahrscheinlich daran, dass dadurch eine noch höhere Packrate erreicht werden kann.[15]
Eine Zeit lang wurde angenommen, dass Dinoflagellaten die einzigen Eukaryoten sind, denen Histone völlig fehlen.[16] Spätere Studien zeigten jedoch, dass ihre Kern-DNA immer noch Histon-Gene enthält.[17]
Histonähnliche Proteine bei Bakterien
In Bakterien findet man keine Core-Histone (mit Ausnahme des Lysin-reichen H1, auch als Nukleoprotein HC1/HC2 bezeichnet).[18] Stattdessen ist bei Bakterien und DNA-haltigen Organellen wie Mitochondrien und Plastiden (insbesondere Chloroplasten) die DNA normalerweise in Nuceloiden (Kernäquivalenten) verdichtet, wofür so genannte ‚histonähnliche Proteine‘ (HLPs, nach englisch histone like proteins, auch Bacterial DNA binding proteins) sorgen. Diese sind untereinander homolog, zu den echten Histonen im Zellkern der eukaryotischen Zellen (Euzyten) aber nur funktionell ähnlich (analog).[19][20] Die Bezeichnungen sind:
- HU in Bakterien (mit Beispiel H-NS)
- Abf2 in Mitochondrien
- HC (englisch histone-like protein of chloroplast) in den Chloroplasten von Rotalgen wie Cyanidioschyzon merolae (Cyanidiales).[21][22]
Diese Beziehungen der Histone und HLPs unterstützen ebenfalls die Endosymbiontentheorie.
Siehe auch
- Epigenetik – Rolle von Histonmodifikationen bei der Regulation des Genoms
- Nukleosom – der Komplex, den die Histone mit der DNA bilden
- Histon-Code
- Histon-Deacetylase
- Sirtuine
- Imprinting
- Transkription (Biologie)
- Polycomb-Körper
Literatur
- M. G. Goll, T. H. Bestor: Histone modification and replacement in chromatin activation. Genes Dev 16(14): S. 1739-1742 (2002) PMID 12130533
- P. A. Grant: A tale of histone modifications. Genome Biol 2(4):REVIEWS0003 (2001) PMID 11305943
- J. M. Eirín-López, L. J. Frehlick, J. Ausió: Protamines, in the footsteps of linker histone evolution. J Biol Chem 281(1): S. 1-4 (2006) PMID 16243843
- S. Inouye, F. I. Tsuji: Monitoring gene expression in Chinese hamster ovary cells using secreted apoaequorin. Anal Biochem 201(1): S. 114-118 (1992) PMID 1621948
- C. Weiller C, F. Chollet, K. J. Friston, R. J. Wise, R. S. Frackowiak: Functional reorganization of the brain in recovery from striatocapsular infarction in man. Ann Neurol 31(5): S. 463-472 (1992) PMID 1596081
- M. Hampsey, D. Reinberg: Tails of intrigue: phosphorylation of RNA polymerase II mediates histone methylation. Cell 113(4): S. 429-432 (2003) PMID 12757703
- B. M. Turner: Cellular memory and the histone code. Cell 111(3): S. 285-291 (2002) PMID 12419240
- A. J. Bannister, R. Schneider, T. Kouzarides: Histone methylation: dynamic or static? Cell 109(7): S. 801-806 (2002) PMID 12110177
- P. Cheung, C. D. Allis, P. Sassone-Corsi: Signaling to chromatin through histone modifications. Cell 103(2): S. 263-271 (2000) PMID 11057899
- C. L. Peterson, M. A. Laniel: Histones and histone modifications. Curr Biol 14(14): S. R546-51 (2004) PMID 15268870
- S. Belikov, V. Karpov: Linker histones: paradigm lost but questions remain. FEBS Lett 441(2): S. 161-164 (1998) PMID 9883876
- C. Von Holt, W. N. Strickland, W. F. Brandt, M. S. Strickland: More histone structures. FEBS Lett 100(2): S. 201-218 (1979) PMID 378692
- Y. Shi, J. R. Whetstine: Dynamic regulation of histone lysine methylation by demethylases. Mol Cell 25(1): S. 1-14 (2007) PMID 17218267
Weblinks
- Video: Der molekulare Aufbau eines Histonoktamers (Animation). Institut für den Wissenschaftlichen Film (IWF) 2007, zur Verfügung gestellt von der Technischen Informationsbibliothek (TIB), doi:10.3203/IWF/C-13106.
Einzelnachweise
- B Henneman, C van Emmerik, H van Ingen, RT Dame: Structure and function of archaeal histones.. In: PLoS genetics. 14, Nr. 9, September 2018, S. e1007582. doi:10.1371/journal.pgen.1007582. PMID 30212449. PMC 6136690 (freier Volltext).
- Informationen der Nobelstiftung zur Preisverleihung 1910 an Albrecht Kossel (englisch).
- Kamakaka RT, Biggins S: Histone variants: deviants?. In: Genes Dev.. 19, Nr. 3, Februar 2005, S. 295–310. doi:10.1101/gad.1272805. PMID 15687254.
- F Mattiroli, S Bhattacharyya, PN Dyer, AE White, K Sandman, BW Burkhart, KR Byrne, T Lee, NG Ahn, TJ Santangelo, JN Reeve, K Luger: Structure of histone-based chromatin in Archaea.. In: Science. 357, Nr. 6351, 11. August 2017, S. 609–612. doi:10.1126/science.aaj1849. PMID 28798133. PMC 5747315 (freier Volltext).
- Alva V, Ammelburg M, Söding J, Lupas AN: On the origin of the histone fold. In: BMC Structural Biology. 7, März 2007, S. 17. doi:10.1186/1472-6807-7-17. PMID 17391511. PMC 1847821 (freier Volltext).
- Bártová E, Krejcí J, Harnicarová A, Galiová G, Kozubek S: Histone modifications and nuclear architecture: a review. In: J. Histochem. Cytochem.. 56, Nr. 8, August 2008, S. 711–21. doi:10.1369/jhc.2008.951251. PMID 18474937. PMC 2443610 (freier Volltext).
- Eberharter A, Becker PB: Histone acetylation: a switch between repressive and permissive chromatin. Second in review series on chromatin dynamics. In: EMBO Rep.. 3, Nr. 3, März 2002, S. 224–9. doi:10.1093/embo-reports/kvf053. PMID 11882541. PMC 1084017 (freier Volltext).
- Zhang Y: Transcriptional regulation by histone ubiquitination and deubiquitination. In: Genes Dev.. 17, Nr. 22, November 2003, S. 2733–40. doi:10.1101/gad.1156403. PMID 14630937.
- Mersfelder EL, Parthun MR: The tale beyond the tail: histone core domain modifications and the regulation of chromatin structure. In: Nucleic Acids Res.. 34, Nr. 9, 2006, S. 2653–62. doi:10.1093/nar/gkl338. PMID 16714444. PMC 1464108 (freier Volltext).
- Suganuma T, Workman JL: Crosstalk among Histone Modifications. In: Cell. 135, Nr. 4, November 2008, S. 604–7. doi:10.1016/j.cell.2008.10.036. PMID 19013272.
- Weake VM, Workman JL: Histone ubiquitination: triggering gene activity. In: Mol. Cell. 29, Nr. 6, März 2008, S. 653–63. doi:10.1016/j.molcel.2008.02.014. PMID 18374642.
- Cloos PA, Christensen J, Agger K, Helin K: Erasing the methyl mark: histone demethylases at the center of cellular differentiation and disease. In: Genes Dev.. 22, Nr. 9, Mai 2008, S. 1115–40. doi:10.1101/gad.1652908. PMID 18451103. PMC 2732404 (freier Volltext).
- Zhang Y, Reinberg D: Transcription regulation by histone methylation: interplay between different covalent modifications of the core histone tails. In: Genes Dev.. 15, Nr. 18, September 2001, S. 2343–60. doi:10.1101/gad.927301. PMID 11562345.
- Michael Cox, David R. Nelson, Albert L Lehninger: Lehninger Principles of Biochemistry. W.H. Freeman, San Francisco 2005, ISBN 978-0-7167-4339-2.
- H. J. Clarke: Nuclear and chromatin composition of mammalian gametes and early embryos. In: Biochemistry and Cell Biology. 70, Nr. 10–11, 1992, S. 856–866. doi:10.1139/o92-134. PMID 1297351.
- P. J. Rizzo: Those amazing dinoflagellate chromosomes. In: Cell Research. 13, Nr. 4, August 2003, S. 215–217. doi:10.1038/sj.cr.7290166. PMID 12974611.
- P. B. Talbert, S. Henikoff: Chromatin: Packaging without Nucleosomes. In: Current Biology. 22, Nr. 24, 2012, S. R1040–R1043. doi:10.1016/j.cub.2012.10.052. PMID 23257187.
- HAROLD E. KASINSKY, JOHN D. LEWIS, JOEL B. DACKS, JUAN AUSIÓ: Origin of H1 linker histones. In: The FASEB Journal. 15, Nr. 1, Januar 2001, S. 34–42. doi:10.1096/fj.00-0237rev. PMID 11149891.
- Drlica K, Rouviere-Yaniv J: Histonelike proteins of bacteria. In: Microbiological Reviews. 51, Nr. 3, September 1987, S. 301–19. PMID 3118156. PMC 373113 (freier Volltext).
- Pettijohn DE: Histone-like proteins and bacterial chromosome structure. In: The Journal of Biological Chemistry. 263, Nr. 26, September 1988, S. 12793–6. PMID 3047111.
- T. Kobayashi, M. Takahara, S. Y. Miyagishima, H. Kuroiwa, N. Sasaki, N. Ohta, M. Matsuzaki, T. Kuroiwa: Detection and Localization of a Chloroplast-Encoded HU-Like Protein That Organizes Chloroplast Nucleoids. In: The Plant Cell Online. 14, Nr. 7, 2002, S. 1579–1589. doi:10.1105/tpc.002717. PMID 12119376. PMC 150708 (freier Volltext).
- Biology, 8th Edition, Campbell & Reece. Benjamin Cummings (Pearson), 2009, ISBN 978-0-321-54325-7, S. 516.