Epigenetik
Die Epigenetik (von altgriechisch ἐπί epi „dazu, außerdem“ und -genetik) ist das Fachgebiet der Biologie, das sich mit der Frage befasst, welche Faktoren die Aktivität eines Gens und damit die Entwicklung der Zelle zeitweilig festlegen. Sie untersucht die Änderungen der Genfunktion, die nicht auf Veränderungen der Sequenz der Desoxyribonukleinsäure (DNA), etwa durch Mutation oder Rekombination, beruhen und dennoch an Tochterzellen weitergegeben werden.
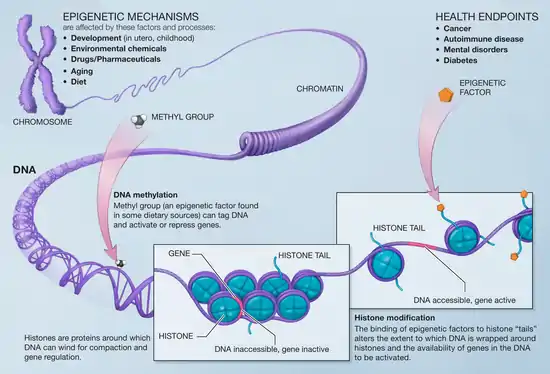
Grundlage sind chemische Veränderungen am Chromatin, der Proteine, die an DNA binden, oder auch Methylierung der DNA selbst, die Abschnitte oder ganze Chromosomen in ihrer Aktivität beeinflussen können. Man spricht auch von epigenetischer Veränderung bzw. epigenetischer Prägung. Da die DNA-Sequenz nicht verändert wird, können epigenetische Effekte nicht im Genotyp (DNA-Sequenz), sehr wohl aber im Phänotyp nachgewiesen und beobachtet werden.
Einführung
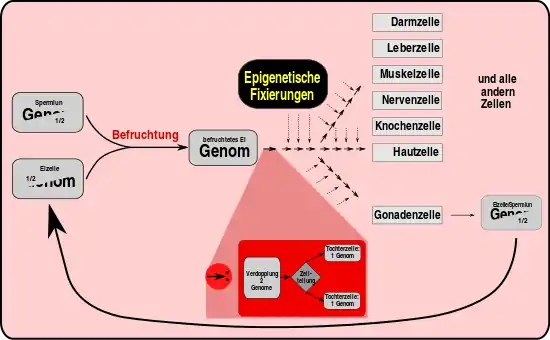
Bei der Vererbung wird Erbgut weitergegeben. Epigenetische Fixierung bewirkt, dass die totipotenten Zellen reifen und sich spezialisieren. Der Reifungsprozess ist normalerweise nicht umkehrbar. (Jeder Pfeil deutet eine Zellteilung an. Dabei wird die Zelle verändert. Diese Veränderungen werden mit dem Erbgut an die Tochterzellen weitergegeben. Es handelt sich dabei nicht um Sequenzveränderungen der DNA.)
Nach der Befruchtung teilt sich die Eizelle. Bis zum 8-Zell-Stadium sind alle Tochterzellen gleichwertig. Man bezeichnet sie als totipotent, weil jede von ihnen noch alleine in der Lage ist, einen kompletten Organismus hervorzubringen. Danach finden sich Zellen mit einem unterschiedlichen inneren Programm, deren Entwicklungspotenzial von nun an eingeschränkt – d. h. mehr und mehr spezialisiert – wird. Wenn der Körper fertig ausgebildet ist, sind die meisten Körperzellen für ihre Funktion fest programmiert (lediglich die sogenannten adulten Stammzellen behalten eine gewisse Flexibilität). Dabei bleibt die Sequenz des Erbguts unverändert (abgesehen von wenigen zufälligen, genetischen Veränderungen = Mutationen). Die funktionelle Festlegung erfolgt durch verschiedene Mechanismen, einer davon beruht auf biochemischen Modifikationen an einzelnen Basen der Sequenz oder der die DNA verpackenden Histone oder beiden. Solche Veränderungen führen dazu, dass bestimmte Bereiche des Erbguts „stillgelegt“, andere dafür leichter transkribiert (in RNA für Proteine umgeschrieben) werden können. Diese Modifizierungen sehen in Körperzellen ganz anders aus als in Stammzellen oder in Keimzellen (Eizellen und Spermien; auch Krebszellen haben meist abweichende [und dabei spezifische] Modifikationsmuster). Die wichtigsten Modifikationen sind die Methylierung von Cytidin-Basen an einem CpG-Dinukleotid (Cytosin-Guanosin-Nukleotid-Dimer, DNA-Methylierung) sowie die Seitenketten-Methylierung und -Acetylierung von Histonen.
Neben Methylierung haben Telomere eine wichtige epigenetische Bedeutung. Telomere schützen die Enden der Chromosomen bei der Zellteilung vor dem Abbau. Das Enzym Telomerase stellt dabei sicher, dass die Chromosomen intakt bleiben. Psychische Belastung kann die Aktivität dieses Enzyms verringern, was zu einer beschleunigten Verkürzung der Telomere im Alterungsprozess führen kann (Nobelpreis für Medizin 2009 an Elizabeth Blackburn).
Begriff
Epigenetisch sind alle Prozesse in einer Zelle, die als „zusätzlich“ zu den Inhalten und Vorgängen der Genetik gelten. Conrad Hal Waddington hat den Begriff Epigenetik erstmals benutzt. Im Jahr 1942 (als die Struktur der DNA noch unbekannt war) definierte er Epigenetik als „the branch of biology which studies the causal interactions between genes and their products which bring the phenotype into being“ („der Zweig der Biologie, der die kausalen Wechselwirkungen zwischen Genen und ihren Produkten, die den Phänotyp hervorbringen, untersucht“). Zur Abgrenzung vom allgemeineren Konzept der Genregulation sind heutige Definitionen meist spezieller, zum Beispiel: „Der Begriff Epigenetik definiert alle meiotisch und mitotisch vererbbaren Veränderungen in der Genexpression, die nicht in der DNA-Sequenz selbst codiert sind.“[1] Andere Definitionen, wie die von Adrian Peter Bird, einem der Pioniere der Epigenetik, vermeiden die Einschränkung auf generationsübergreifende Weitergabe. Epigenetik beschreibe „die strukturelle Anpassung chromosomaler Regionen, um veränderte Zustände der Aktivierung zu kodieren, zu signalisieren oder zu konservieren.“[2] In einer Überblicksarbeit zum Thema Epigenetik bei Bakterien wurde von Casadesús und Low vorgeschlagen, eine vorläufige Definition zu benutzen, solange es keine allgemein akzeptierte Definition der Epigenetik gibt: „Eine vorläufige Definition könnte jedoch sein, dass die Epigenetik die Untersuchung der Zelllinienbildung durch nicht-mutationale Mechanismen anspricht.“[3]
Epigenese
Mit dem Ausdruck Epigenese werden die graduellen Prozesse der embryonalen Morphogenese von Organen beschrieben. Diese beruhen auf Mechanismen auf der Ebene von Zellen und Zellverbänden, das sind Turing-Mechanismen oder allgemein Musterbildungsprozesse in der Biologie. Beispiele hierfür findet man etwa bei der Erklärung der embryonalen Extremitätenentwicklung der Wirbeltiere.
Zugehörige Begriffe
Zu den epigenetischen Prozessen zählt man die Paramutation, das Bookmarking, das Imprinting, das Gen-Silencing, die X-Inaktivierung, den Positionseffekt, die Reprogrammierung, die Transvection, maternale Effekte (paternale Effekte sind selten, da wesentlich weniger nicht-genetisches Material mit dem Spermium „vererbt“ wird), den Prozess der Karzinogenese, viele Effekte von teratogenen Substanzen, die Regulation von Histonmodifikationen und Heterochromatin sowie technische Limitierungen beim Klonen.
Epigenetik im Vergleich zur Genetik
Man kann den Begriff Epigenetik verstehen, wenn man sich den Vorgang der Vererbung vor Augen führt:
- Vor einer Zellteilung wird die Erbsubstanz verdoppelt. Jeweils die Hälfte des verdoppelten Genoms wird dann auf eine der beiden Tochterzellen übertragen. Bei der sexuellen Vermehrung des Menschen, der Fortpflanzung, werden von der Eizelle die Hälfte des mütterlichen Erbguts und vom Spermium die Hälfte des väterlichen Erbguts miteinander vereint.
- Die Molekulargenetik beschreibt die Erbsubstanz als Doppelhelix aus zwei Desoxyribonukleinsäure-Strängen, deren Rückgrat aus je einem Phosphat-Desoxyribosezucker-Polymer besteht. Die genetische Information ist durch die Reihenfolge der vier Basen Adenin (A), Cytosin (C), Guanin (G) und Thymin (T) bestimmt, die jeweils an einen der Desoxyribose-Zucker angehängt sind.
- Die Basen des einen Stranges paaren sich fast immer mit einer passenden Base des zweiten Stranges. Adenin paart sich mit Thymin, und Cytosin paart sich mit Guanin.
- In der Reihenfolge der Bausteine A, C, G, T (der Basensequenz) ist die genetische Information verankert.
Einige Phänomene der Vererbung lassen sich nicht mit dem gerade beschriebenen DNA-Modell erklären:
- Bei der Zelldifferenzierung entstehen im Verlauf von Zellteilungen Tochterzellen mit anderer Funktion, obwohl das Erbgut in allen Zellen gleich ist. Die Festlegung der funktionellen Identität einer Zelle ist ein Thema der Epigenetik.
- Es gibt Eigenschaften, die nur vom Vater her (paternal) „vererbt“ werden, so wie es Eigenschaften gibt, die nur von der Mutter (maternal) stammen und die nicht mit der Basensequenz in Zusammenhang stehen.
- Bei der Rückumwandlung von funktionell festgelegten Zellen (terminal differenzierte Zellen) in undifferenzierte Zellen, die sich wieder in verschiedene Zellen entwickeln können und die bei der Klonierung von Individuen (z. B. von Dolly) eingesetzt werden, müssen epigenetische Fixierungen aufgehoben werden, damit eine Zelle nicht auf eine einzige Funktion festgelegt bleibt, sondern wieder alle oder viele Funktionen erwerben und vererben kann.
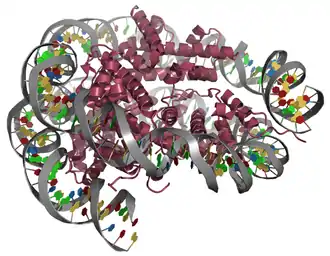
Die DNA ist um den Kern aus acht Histon-Untereinheiten (je zwei H2a, H2b, H3 und H4) gewickelt und macht etwa 1,7 Umdrehungen. An das Stück DNA zwischen zwei Nukleosomen bindet Histon 1 (H1). Die Enden der Histone sind für epigenetische Modifizierung verfügbar: Methylierung, Acetylierung oder Phosphorylierung. Dadurch wird die Verdichtung oder Ausdehnung des Chromatins beeinflusst.[4]
Histone und ihre Rolle bei der epigenetischen Fixierung
DNA liegt im Zellkern nicht nackt vor, sondern ist an Histone gebunden. Acht verschiedene Histonproteine, jeweils zwei Moleküle von Histon 2A, Histon 2B, Histon 3 und Histon 4 bilden den Kern eines Nukleosoms, auf das 146 Basenpaare eines DNA-Stranges aufgespult sind. Die Enden der Histonstränge ragen aus dem Nukleosom heraus und sind Ziel von Histon-modifizierenden Enzymen. Vor allem Methylierungen und Acetylierungen an Lysin, Histidin oder Arginin, außerdem Phosphorylierungen an Serinen sind die bekannten Modifizierungen. Außerdem spielt es eine Rolle, ob die Lysin-Seitenkette mit ein, zwei oder drei Methyl-Gruppen belegt ist. Durch vergleichende Analyse postuliert man eine Art von „Histon-Code“, der in direktem Zusammenhang mit der Aktivität des von den Histonen jeweils gebundenen Gens stehen soll.
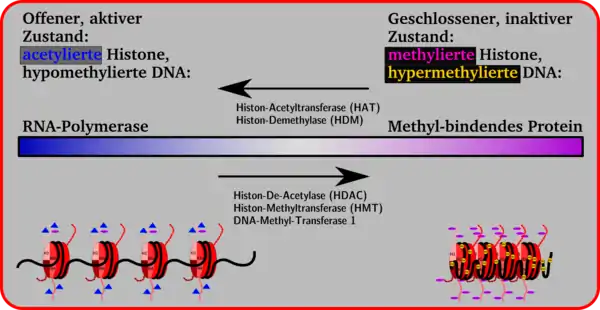
Die Histonseitenketten in den Nukleosomen können enzymatisch verändert werden. Dadurch ändert sich das Volumen eines Gensegments. Kleinere Volumina, geschlossene Konformation, Chromosomkondensierung und Inaktivität eines Gens stehen auf der einen Seite, größere Volumina, offene Konformation und Gen-Aktivität auf der anderen. Zwischen beiden Seiten ist ein Übergang möglich, der durch Anheftung und Abspaltung von Methylgruppen an Cytidin-Basen, durch Methylierung, Demethylierung, Acetylierung oder Deacetylierung mit Hilfe von Enzymen bewirkt wird.
Generell gilt, dass die Anheftung von Acetyl-Gruppen an die Lysin-Seitenketten der Histone zur Öffnung der Nukleosomen-Konformation führt, wodurch das Gen für die Transkription durch die RNA-Polymerase verfügbar wird. Durch eine verstärkte Anheftung von Methyl-Gruppen an Lysin-Seitenketten werden Proteine angeheftet wie z. B. das Methyl-bindende Protein MeCB, das die Genexpression unterdrückt. Diese Repressorproteine führen dazu, dass die Histon-Konformation geschlossen wird und keine Transkription mehr möglich ist.
Methoden der epigenetischen Forschung
Restriktionsendonukleasen, die nur an demethylierten CG-Dimeren schneiden
HpaII (die zweite Restriktionsendonuklease aus Haemophilus parainfluenza) schneidet CCGG-Palindrome nur, wenn die CG-Dimere nicht methyliert sind, im Vergleich zu BsiSI (aus Bacillus), die auch methylierte CmeCGG-Palindrome schneidet. Tryndiak und Mitarbeiter zeigen damit, dass bei Zellen auf dem Weg zum Mammakarzinom ein fortschreitender globaler Verlust von DNA-Methylierung mit einer fehlgeleiteten Bildung der DNMT1, meCG-bindender Proteine und Veränderungen in den Histonen einhergeht.[5]
Bisulfit-Sequenzierung
Durch Behandlung von DNA mit Natriumhydrogensulfit (alter Name „Bisulfit“) wird Cytosin (C) in Uracil (U) umgewandelt. Bei einer anschließenden Sequenzierung findet man daher an den Stellen, wo vorher ein C war, nun ein U/T. Da bisulfit-behandelte DNA sehr labil ist, wird das Gen, das man analysieren möchte, mittels PCR wieder amplifiziert. Bei der nachfolgenden Sequenzierung werden dann T bzw. TG (Thymin-Guanosin-Dimere) identifiziert, wo in der unbehandelten DNA Cytosin bzw. CG-Dimere existierten.
Für die epigenetische Analyse ist wichtig, dass nur nicht-methylierte C-Basen konvertiert werden, während meC in CG-Dimeren nicht in Thymin konvertiert werden. Man kann daher mit dieser Methode exakt analysieren, welche CG-Dimere in einer bestimmten Zelle methyliert waren. Indem man das bisulfit-behandelte Genstück, das man analysieren möchte, nach der PCR-Amplifikation kloniert und verschiedene Klone sequenziert, erhält man eine Abschätzung, ob ein bestimmtes CG-Dimer gar nicht, vollständig oder nur partiell methyliert war. Bei der Methode des Pyrosequencing ist dieses Verfahren noch verfeinert und erlaubt genauere quantitative Aussagen: Man kann zum Beispiel den Schweregrad einer Tumorentartung mit dem Methylierungsgrad von CG-Inseln einzelner sogenannter Tumor-Suppressorproteine vergleichen und stellt fest, dass in bestimmten Tumoren des blutbildenden Systems (Hämatopoietisches System) bestimmte meCG-Dimere mit steigendem Schweregrad immer stärker methyliert sind.
Chromatin-Immunpräzipitation
Bei dieser Methode kann bestimmt werden, ob ein bestimmtes Protein an ein gegebenes DNA-Stück bindet: Durch Behandlung der Zellen oder biologischen Gewebes mit Formaldehyd werden die bindenden Proteine mit der DNA kovalent verknüpft. Die aus den Zellen extrahierte DNA wird anschließend durch Behandlung mit Ultraschall in Bruchstücke von 50 bis 1000 Basenpaaren fragmentiert, an denen die gebundenen Proteine verbleiben. In einem nächsten Schritt wird mit einem Antikörper das interessierende Protein zusammen mit den daran gebundenen DNA Fragmenten extrahiert, und anschließend wird die kovalente Bindung zwischen Protein und DNA durch Hitzebehandlung in 300 mM Kochsalzlösung wieder aufgelöst. Die damit abgetrennten DNA-Fragmente können anschließend identifiziert bzw. quantifiziert werden (mittels genspezifischer PCR oder genomweiter NGS). Aus der Häufigkeit, mit der dabei ein bestimmtes DNA Fragment identifiziert wird, lässt sich schlussfolgern, ob bzw. wie stark das Protein in der lebenden Zelle mit dem betreffenden DNA-Abschnitt assoziiert war. Je nachdem, welches Protein man mit Antikörpern versucht zu präzipitieren, kann man z. B. sagen:
- Die RNA-Polymerase hat an das Gen gebunden, daher wurde es transkribiert, das Gen war aktiv.
- Das meCG-bindende Protein (MeCP) war an das Gen gebunden, daher wurde dieses nicht transkribiert und war ruhig gestellt (engl. silencing).
Electrophoretic Mobility Shift Assay
Die unterschiedlichen DNA-Moleküle weisen ein unterschiedliches Laufverhalten in einer Gelelektrophorese auf.
Epigenomik und Methoden der epigenetischen Forschung
Epigenomik (engl. Epigenomics) oder Epigenomforschung ist ein Teilgebiet der Epigenetik, das die Untersuchung des möglichst vollständigen Satzes epigenetischer Modifikationen am genetischen Material einer Zelle zum Ziel hat. Solche zusammenhängenden Sätze von epigenetischen Modifikationen werden Epigenome genannt.
Der Begriff Epigenomics wurde analog zu anderen -omics, wie Genomics und Proteomics, gebildet und wurde populär, als Methoden zur Verfügung standen, epigenetische Modifikationen im größeren Stil zu untersuchen. Die Initiierung des Human Epigenome Project im Jahr 1999 hat dazu einen wesentlichen Beitrag geleistet.[6][7]
Epigenomik und Epigenetik schließen sich nicht aus. Die umfassende und effiziente Erforschung der Epigenetik auf globaler Ebene wird durch Hochdurchsatz-Methoden ermöglicht. Die Verwendung des Begriffs Epigenomforschung bzw. Epigenomik kennzeichnet diese Vorgehensweise spezifischer als die Verwendung des Begriffs Epigenetik.
Zwei der am meisten charakterisierten epigenetischen Markierungen sind DNA-Methylierungen und Histonmodifikationen. Eine Gesamtbestimmung von DNA-Methylierungen zu einer bestimmten Zeit, in einem bestimmten Gewebe usw. wird häufig als Methylom oder DNA-Methylierungsmuster bezeichnet, ein zusammenhängender Satz von Histonmodifikationen wird häufig Histon-Code genannt. Sowohl Methylome (bzw. DNA-Methylierungsmuster) als auch Histon-Codes sind Teil-Epigenome.
Beispielhaft seien hier für die Untersuchung von Epigenomen die Bisulfit-Sequenzierung und die ChIP-Seq genannt. Die Bisulfit-Sequenzierung ermöglicht eine umfassende Analyse von "Methylomen" (DNA-Methylierungsmustern) und die ChIP-Seq kann für die Interaktion von Histonen mit der DNA eingesetzt werden.
Es ist anzumerken, dass die Epigenomforschung zwar als Teilgebiet der Epigenetik angesehen werden kann, jedoch ein sehr interdisziplinäres Fach ist, das beispielsweise Schnittmengen mit der Genetik, der Molekularbiologie, allen -omik-Gebieten, der Systembiologie und der Bioinformatik aufweist.[8]
Epigenetische Veränderungen im Lebenslauf
Epigenetik beschränkt sich nicht auf Vererbungsfälle. Zunehmende Beachtung finden epigenetische Forschungsergebnisse im Zusammenhang mit anhaltenden Veränderungen im Lebenslauf sowie im Zusammenhang mit der Ausbildung von Krankheiten. So konnte an 80 eineiigen Zwillingen nachgewiesen werden, dass sie im Alter von drei Jahren epigenetisch noch in hohem Maß übereinstimmen, nicht mehr aber im Alter von 50 Jahren, wenn sie wenig Lebenszeit miteinander verbrachten und/oder eine unterschiedliche medizinisch-gesundheitliche Geschichte hinter sich hatten. So war der Methylierungsgrad bei dem einen Zwilling bis zu zweieinhalb mal höher als bei dem anderen Zwilling, sowohl in absoluten Zahlen als auch, was die Verteilung der epigenetischen Marker angeht. Ältere eineiige Zwillinge sind demnach trotz ihrer genetischen Identität epigenetisch umso verschiedener, je unterschiedlicher das Leben der Zwillinge verläuft. Der Grund liegt neben der erlebten Umwelt auch in der Ungenauigkeit bei der Übertragung von Methylgruppenmustern bei jeder Zellteilung. Schleichende Veränderungen summieren sich damit im Lauf eines Lebens immer stärker auf.[9]
Die Umstellung der Ernährung bei Arbeiterbienen nach Ablauf der ersten Wochen des Larvenstadiums auf eine einfache Pollen- und Honigkost im Vergleich zur Königin verursacht eine hochgradige epigenetische Umprogrammierung des Larvengenoms. Mehr als 500 Gene wurden identifiziert, die von den umweltspezifisch verursachten Methylierungsveränderungen betroffen sind. Nicht nur die Aktivierung bzw. Nichtaktivierung von Genen ist die Folge des Ernährungswandels, sondern sogar alternatives Splicing und veränderte Genprodukte.[10]
„Vererbung“ epigenetischer Prägungen?
Eine Vererbung epigenetischer Prägungen wurde 2003 von Randy Jirtle und Robert Waterland mittels Mäuseexperimenten vorgeschlagen.[11] Weiblichen Agoutimäusen wurde vor der Paarung und während der Schwangerschaft eine bestimmte Zusammensetzung an Nährstoffen verabreicht. Es zeigte sich, dass ein Großteil der Nachkommen nicht den typischen Phänotyp aufwies.
Verborgene Erblichkeit
Bei der Suche nach erblichen Faktoren von Krankheiten, zum Beispiel mit Hilfe von genomweiten Assoziationsstudien (GWAS), wurde oft beobachtet, dass DNA-Abweichungen nur einen kleinen Teil der vermuteten Erblichkeit erklären konnten. Inzwischen geht man davon aus, dass die Erklärung für diese "Blindheit" die verborgene Erblichkeit (engl. missing heritability) ist, die auf einer Vielzahl epigenetischer Prozesse beruht.[12] Die Aufklärung – oder auch nur eine Abschätzung – dieser Prozesse ist eine besondere Herausforderung, nicht nur wegen ihrer Vielfältigkeit, sondern auch wegen ihrer Veränderlichkeit (Dynamik). Im Lebenslauf einer Zelle oder eines Organismus sowie im Verlauf über mehrere Generationen können epigenetische Einstellungen entstehen, gelöscht werden und wieder neu entstehen – je nach dem Wechsel innerer und äußerer Einflüsse.[13]
Anwendung bei Krebstherapie
Die Eigenschaft, dass die epigenetischen Einstellungen grundsätzlich umkehrbar sind, hat in jüngster Zeit zu völlig neuen Therapiemöglichkeiten – unter anderem gegen Krebs – geführt. Es ist nunmehr möglich, gezielt Moleküle zu entwickeln, die bestimmte Krebs begünstigende, schädliche epigenetische Regulatoren entschärfen und somit die Krebserkrankung bekämpfen.[14][15][16]
Anwendung bei Suchttherapie
Eine Vielzahl epigenetischer Veränderungen im Belohnungszentrum des Gehirns durch den Konsum von Rauschdrogen ist bekannt. Auch die Vererbbarkeit mancher dieser Veränderungen konnte nachgewiesen werden. Die gezielte Beeinflussung der suchtfördernden epigenetischen Einstellungen wurde in vorklinischen Studien in Tiermodellen zwar schon erreicht, erwies sich jedoch als so kompliziert, dass Anwendungen in der Suchttherapie beim Menschen in naher Zukunft noch nicht absehbar waren.[17]
Der Konsum von Cannabis von Seiten der Mutter oder des Vaters vor einer Schwangerschaft sowie von Seiten der Mutter während der Schwangerschaft führt bei Neugeborenen zu epigenetischen Veränderungen, die dafür bekannt sind, dass sie in einem Zusammenhang stehen mit erhöhter Anfälligkeit für psychiatrische Störungen wie Autismus, ADHS, Schizophrenie, Suchtverhalten und andere. Eine entsprechende Information der Konsumenten wurde daher empfohlen.[18][19][20][21]
Epigenetik bei Bakterien
Während die Epigenetik bei Eukaryoten, insbesondere bei den vielzelligen Tieren (Metazoa), von grundlegender Bedeutung ist, spielt sie bei Bakterien eine andere Rolle. Am wichtigsten ist dabei, dass Eukaryoten epigenetische Mechanismen in erster Linie zur Regulierung der Genexpression verwenden, was die Bakterien selten tun. Allerdings ist die Nutzung der postreplikativen DNA-Methylierung für die epigenetische Kontrolle von DNA-Protein-Wechselwirkungen bei Bakterien weit verbreitet. Außerdem verwenden Bakterien DNA-Adenin-Methylierung (anstelle von DNA-Cytosin-Methylierung) als epigenetisches Signal. DNA-Adenin-Methylierung ist bei Bakterienvirulenz in Organismen wie Escherichia coli, Salmonella, Vibrio, Yersinia, Haemophilus und Brucella wichtig. In den Alphaproteobakterien reguliert die Methylierung von Adenin den Zellzyklus und verbindet die Gentranskription mit der DNA-Replikation. In Gammaproteobakterien liefert die Adeninmethylierung Signale für die DNA-Replikation, Chromosomensegregation, Fehlpaarungsreparatur, Verpackung von Bakteriophagen, Transposaseaktivität und Regulation der Genexpression.[22][23] Es gibt einen genetischen Schalter, der Streptococcus pneumoniae (Pneumokokken) kontrolliert und es dem Bakterium erlaubt, seine Eigenschaften zufällig so zu ändern, dass einer von sechs alternativen Zuständen eintritt. Das könnte den Weg zu verbesserten Impfstoffen ebnen. Jede Form wird zufällig durch ein phasenvariables Methylierungssystem erzeugt. Die Fähigkeit der Pneumokokken, tödliche Infektionen zu verursachen, ist in jedem dieser sechs Zustände unterschiedlich. Ähnliche Systeme existieren in anderen Bakteriengattungen.[24]
Literatur
Handbücher
- Trygve Tollefsbol (Hrsg.): Handbook of Epigenetics. The New Molecular and Medical Genetics, Academic Press, London 2017, ISBN 9780128054772.
- Suming Huang, Michael D Litt, C. Ann Blakey (Hrsg.): Epigenetic Gene Expression and Regulation, Academic Press, London 2016, ISBN 9780128004715.
- Trygve Tollefsbol (Hrsg.): Transgenerational Epigenetics: Evidence and Debate, Elsevier, London 2014, ISBN 9780124059221.
Monographien
- Jan Baedke: Above the Gene, Beyond Biology: Toward a Philosophy of Epigenetics. University of Pittsburgh Press, Pittsburgh 2018.
- Joachim Bauer: Das Gedächtnis des Körpers: wie Beziehungen und Lebensstile unsere Gene steuern. Eichborn, Frankfurt am Main 2002; Erweiterte Taschenbuchausgabe: Piper, München 2004 (10. Aufl. 2007), ISBN 978-3-492-24179-3.
- Peter Spork: Gesundheit ist kein Zufall. Wie das Leben unsere Gene prägt. Die neuesten Erkenntnisse der Epigenetik. DVA, München 2017, ISBN 978-3-421-04750-2.
Aufsätze
- Bradbury, J. (2003): Human Epigenome Project—Up and Running. In: PLoS Biol. Bd. 1, S. e82 doi:10.1371/journal.pbio.0000082; PDF (freier Volltextzugriff, engl.).
- B. M. Maia, R. M. Rocha, G. A. Calin: Clinical significance of the interaction between non-coding RNAs and the epigenetics machinery: challenges and opportunities in oncology. In: Epigenetics. Band 9, Nummer 1, Januar 2014, S. 75–80, doi:10.4161/epi.26488, PMID 24121593, PMC 3928188 (freier Volltext) (Review).
- C. D. Allis, T. Jenuwein: The molecular hallmarks of epigenetic control. In: Nature Reviews Genetics. Band 17, Nummer 8, 08 2016, S. 487–500, doi:10.1038/nrg.2016.59, PMID 27346641 (Review), PDF.
- E. Jablonka: The evolutionary implications of epigenetic inheritance. In: Interface focus. Band 7, Nummer 5, Oktober 2017, S. 20160135, doi:10.1098/rsfs.2016.0135, PMID 28839916, PMC 5566804 (freier Volltext) (Review).
- Delcuve, GP. et al. (2009): Epigenetic control. In: J Cell Physiol. Bd. 219, Nr. 2, S. 243–250, PMID 19127539.
- Morgan, HD. et al. (2005): Epigenetic reprogramming in mammals. In: Human molecular genetics. (Hum Mol Genet.) Bd. 14, Nr. 1, R47–58, PMID 15809273; PDF (freier Volltextzugriff, engl.)
- Jan Baedke (2019): Philosophische Probleme der Epigenetik. Bericht. In: Information Philosophie Heft 2/2019, S. 22–31. Literaturangaben in Information Philosophie.
Weblinks
Portale
Einzelbeiträge
- Epigenetik: wie Umwelt und Verhalten Gene steuern. Audio auf: funkkolleg-biologie.de, 18. November 2017, abgerufen am 7. Januar 2021.
- „Epigenetik“ bei Planet Wissen, abgerufen am 3. Oktober 2020.
- Warum ist das allen brillanten Genetikern in hundert Jahren nicht aufgefallen? In: Spektrum, 26. März 2014.
- Bruch des bösen Zaubers. In: Der Spiegel. Nr. 32, 2008 (online).
Einzelnachweise
- “The term ‘epigenetics’ defines all meiotically and mitotically heritable changes in gene expression that are not coded in the DNA sequence itself.” In: Gerda Egger u. a.: Epigenetics in human disease and prospects for epigenetic therapy. In: Nature 429, S. 457–463 (2004).
- „… the structural adaptation of chromosomal regions so as to register, signal or perpetuate altered activity states.“ In: Adrian Peter Bird: Perceptions of epigenetics. In: Nature. Band 447, Nummer 7143, Mai 2007, S. 396–398, doi:10.1038/nature05913, PMID 17522671.
- “However, a tentative definition may be that epigenetics addresses the study of cell lineage formation by non-mutational mechanisms.” In: Josep Casadesús, David A. Low: Programmed heterogeneity: epigenetic mechanisms in bacteria. In: The Journal of biological chemistry. Band 288, Nummer 20, Mai 2013, S. 13929–13935, doi:10.1074/jbc.R113.472274, PMID 23592777, PMC 3656251 (freier Volltext) (Review).
- Abbildung von Clapier et al., Proteindatenbank 2PYO; Clapier, CR. et al. (2007): Structure of the Drosophila nucleosome core particle highlights evolutionary constraints on the H2A-H2B histone dimer. In: Proteins 71 (1); 1–7; PMID 17957772; PMC 2443955 (freier Volltext)
- Tryndiak, VP. et al. (2006): Loss of DNA methylation and histone H4 lysine 20 trimethylation in human breast cancer cells is associated with aberrant expression of DNA methyltransferase 1, Suv4-20H2 histone methyltransferase and methyl-binding proteins. In: Cancer Biol Ther. 5(1), 65–70; PMID 16322686; PDF (freier Volltextzugriff, engl.)
- "... The Human Epigenome Project, for example, was established in 1999, when researchers in Europe teamed up to identify, catalogue and interpret genomewide DNA methylation patterns in human genes. ..." In: L. Bonetta: Epigenomics : Detailed analysis. In: Nature. Band 454, 2008, S. 796, doi:10.1038/454795a.
- "... Over time, the field of epigenetics gave rise to that of epigenomics, which is the study of epigenetic modifications across an individual's entire genome. Epigenomics has only become possible in recent years because of the advent of various sequencing tools and technologies, such as DNA microarrays, cheap whole-genome resequencing, and databases for studying entire genomes (Bonetta, 2008) ..." In: L. Bonetta: "Epigenomics: The new tool in studying complex diseases." Nature Education. Band 1, 2008, S. 178, Weblink.
- K. A. Janssen, S. Sidoli, B. A. Garcia: Recent Achievements in Characterizing the Histone Code and Approaches to Integrating Epigenomics and Systems Biology. In: Methods in enzymology. Band 586, 2017, S. 359–378, doi:10.1016/bs.mie.2016.10.021, PMID 28137571, PMC 5512434 (freier Volltext) (Review).
- Mario F. Fraga, Esteban Ballestar, Maria F. Paz, Santiago Ropero, Fernando Setien, Maria L. Ballestar, Damia Heine-Suñer, Juan C. Cigudosa, Miguel Urioste, Javier Benitez, Manuel Boix-Chornet, Abel Sanchez-Aguilera, Charlotte Ling, Emma Carlsson, Pernille Poulsen, Allan Vaag, Zarko Stephan, Tim D. Spector, Yue-Zhong Wu, Christoph Plass, and Manel Esteller. Epigenetic differences arise during the lifetime of monozygotic twins. Proceedings of the National Academy of Sciences. 2005. July 26, 2005. Vol. 102. No. 30
- Frank Lyko, Sylvain Foret u. a.: The Honey Bee Epigenomes: Differential Methylation of Brain DNA in Queens and Workers. In: PLoS Biology. 8, 2010, S. e1000506, doi:10.1371/journal.pbio.1000506.
- Waterland, RA. und Jirtle, RL. (2003): Transposable elements: targets for early nutritional effects on epigenetic gene regulation. In: Mol Cell Biol. 23(15); 5293-5300; PMID 12861015; PMC 165709 (freier Volltext)
- M. Trerotola, V. Relli, P. Simeone, S. Alberti: Epigenetic inheritance and the missing heritability. In: Human genomics. Band 9, Juli 2015, S. 17, doi:10.1186/s40246-015-0041-3, PMID 26216216, PMC 4517414 (freier Volltext) (Review).
- W. W. Burggren: Dynamics of epigenetic phenomena: intergenerational and intragenerational phenotype 'washout'. In: The Journal of experimental biology. Band 218, Pt 1Januar 2015, S. 80–87, doi:10.1242/jeb.107318, PMID 25568454 (freier Volltext) (Review).
- R. L. Bennett, J. D. Licht: Targeting Epigenetics in Cancer. In: Annual review of pharmacology and toxicology. Band 58, 01 2018, S. 187–207, doi:10.1146/annurev-pharmtox-010716-105106, PMID 28992434, PMC 5800772 (freier Volltext) (Review).
- L. Villanueva, D. Álvarez-Errico, M. Esteller: The Contribution of Epigenetics to Cancer Immunotherapy. In: Trends in immunology. Band 41, Nummer 8, 08 2020, S. 676–691, doi:10.1016/j.it.2020.06.002, PMID 32622854 (freier Volltext) (Review).
- A. Roberti, A. F. Valdes, R. Torrecillas, M. F. Fraga, A. F. Fernandez: Epigenetics in cancer therapy and nanomedicine. In: Clinical Epigenetics. Band 11, Nummer 1, 05 2019, S. 81, doi:10.1186/s13148-019-0675-4, PMID 31097014, PMC 6524244 (freier Volltext) (Review).
- R. C. Pierce, B. Fant, S. E. Swinford-Jackson, E. A. Heller, W. H. Berrettini, M. E. Wimmer: Environmental, genetic and epigenetic contributions to cocaine addiction. In: Neuropsychopharmacology. Band 43, Nummer 7, 06 2018, S. 1471–1480, doi:10.1038/s41386-018-0008-x, PMID 29453446, PMC 5983541 (freier Volltext) (Review).
- A. Smith, F. Kaufman, M. S. Sandy, A. Cardenas: Cannabis Exposure During Critical Windows of Development: Epigenetic and Molecular Pathways Implicated in Neuropsychiatric Disease. In: Current environmental health reports. Band 7, Nummer 3, 09 2020, S. 325–342, doi:10.1007/s40572-020-00275-4, PMID 32441004, PMC 7458902 (freier Volltext) (Review).
- Y. L. Hurd, O. J. Manzoni, M. V. Pletnikov, F. S. Lee, S. Bhattacharyya, M. Melis: Cannabis and the Developing Brain: Insights into Its Long-Lasting Effects. In: Journal of Neuroscience. Band 39, Nummer 42, 10 2019, S. 8250–8258, doi:10.1523/JNEUROSCI.1165-19.2019, PMID 31619494, PMC 6794936 (freier Volltext) (Review).
- H. Szutorisz, Y. L. Hurd: High times for cannabis: Epigenetic imprint and its legacy on brain and behavior. In: Neuroscience and biobehavioral reviews. Band 85, 02 2018, S. 93–101, doi:10.1016/j.neubiorev.2017.05.011, PMID 28506926, PMC 5682234 (freier Volltext) (Review).
- R. Schrott, S. K. Murphy: Cannabis use and the sperm epigenome: a budding concern? In: Environmental epigenetics. Band 6, Nummer 1, Januar 2020, S. dvaa002, doi:10.1093/eep/dvaa002, PMID 32211199, PMC 7081939 (freier Volltext) (Review).
- Jörg Tost: Epigenetics. Caister Academic Press, Norfolk, England 2008, ISBN 1-904455-23-9.
- Casadesús J, Low D: Epigenetic gene regulation in the bacterial world. In: Microbiol. Mol. Biol. Rev.. 70, Nr. 3, September 2006, S. 830–56. doi:10.1128/MMBR.00016-06. PMID 16959970. PMC 1594586 (freier Volltext).
- Manso AS, Chai MH, Atack JM, Furi L, De Ste Croix M, Haigh R, Trappetti C, Ogunniyi AD, Shewell LK, Boitano M, Clark TA, Korlach J, Blades M, Mirkes E, Gorban AN, Paton JC, Jennings MP, Oggioni MR: A random six-phase switch regulates pneumococcal virulence via global epigenetic changes. In: Nature Communications. 5, September 2014, S. 5055. doi:10.1038/ncomms6055. PMID 25268848. PMC 4190663 (freier Volltext).