Spleißen (Biologie)
Als Spleißen bzw. Splicing (englisch splice ‚miteinander verbinden‘, ‚zusammenkleben‘) wird ein wichtiger Schritt der Weiterverarbeitung (Prozessierung) der Ribonukleinsäure (RNA) bezeichnet, der im Zellkern von Eukaryoten stattfindet und bei dem aus der prä-mRNA die reife mRNA entsteht.
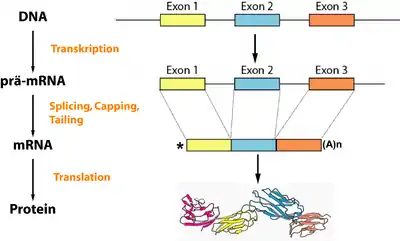
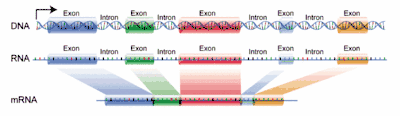
Die zunächst in der Transkription gebildete prä-mRNA enthält noch Introns und Exons. Durch das Splicing werden die Introns entfernt und die angrenzenden Exons miteinander zur fertigen mRNA verknüpft.
Splicing findet zusammen mit der Polyadenylierung (Tailing) des 3'-Endes nach der Transkription statt, ist also ein posttranskriptioneller Vorgang. Im Unterschied dazu ist das Capping des 5'-Endes ein cotranskriptioneller Vorgang.
Geschichte der Entdeckung
Frühe genetische Untersuchungen konnten zeigen, dass Gen, mRNA und Protein colinear sind, was durch die direkte Abschrift bzw. Übersetzung naheliegend war. Sehr gut ist dies in prokaryotischen Organismen zu beobachten, wo Transkription und Translation nicht durch eine Kompartimentierung der Zelle voneinander getrennt sind. Noch während die RNA-Polymerase die mRNA an der DNA synthetisiert, können bereits Ribosomen die naszierende Kette binden und mit der Translation beginnen, wodurch es zur Ausbildung sogenannter Polysomen kommt.
In Eukaryoten ist eine Kopplung von Transkription und Translation aber nicht möglich, da eine Kernmembran die beiden Prozesse räumlich voneinander trennt.
Darüber hinaus konnten Chow et al.[1] und Berget et al.[2] in sehr anschaulichen elektronenmikroskopischen Untersuchungen von RNA:DNA-Hybriden am Beispiel von Adenoviren zeigen, dass die mRNA in Eukaryoten offenbar einer zusätzlichen Prozessierung unterliegen muss, da ihr interne Bereiche fehlen, die aber in der DNA vorkommen. Indirekt konnte eine Reifung anhand der im Vergleich zu zytoplasmatischen RNAs kurzen Halbwertszeit von primären Transkripten, den sogenannten heterogenen nukleären RNAs (hnRNAs) gezeigt werden.
Richard John Roberts und Phillip A. Sharp entwickelten auf dieser Basis das Konzept der split genes und des prä-mRNA-Spleißens, das 1993 mit dem Nobelpreis für Medizin belohnt wurde.[3] Grundlegend neu dabei war, dass der Bereich eines eukaryotischen Gens auf der DNA immer wieder von Sequenzen unterbrochen wird, die nicht in Aminosäuren des späteren Proteins translatiert werden. Diese sogenannten intervening sequences, auch als Introns bezeichnet, werden in einem als prä-mRNA-Spleißen bezeichneten Prozess aus dem Primärtranskript, der prä-mRNA (precursor-mRNA), ausgeschnitten und degradiert. Dabei werden gleichzeitig die beiden angrenzenden proteincodierenden Sequenzabschnitte, oder kurz Exons für expressed sequences, miteinander verknüpft.
Ein Gen kann dabei bis zu über 60 Introns mit Längen zwischen 35 und 100.000 Nukleotiden enthalten. Darüber hinaus tritt das Splicing nicht nur bei den genannten Eukaryoten auf, sondern auch in Mitochondrien, Archaea und einigen der bereits erwähnten viralen RNAs.
Autokatalytisches Spleißen (self-splicing)
Einige RNAs können Introns ohne die Hilfe eines großen Spliceosoms (siehe unten) entfernen. Die chemische Aktivität dazu besitzen sie selbst, d. h., es handelt sich um Ribozyme, die nur in einigen Fällen (Gruppe II Introns) die Hilfe von Proteinen für eine korrekte Faltung benötigen.
1981 konnten T. Cech et al. für den Vorläufer der 26S rRNA von Tetrahymena thermophila nachweisen, dass für die Prozessierung eines etwa 400 Nukleotide langen Introns keine Proteinkomponenten benötigt werden, sondern dass die Aktivität von der RNA selber herrührt. Man spricht daher auch vom autokatalytischen Splicing oder self-splicing. Für diese Entdeckung eines ersten Ribozyms und damit der katalytischen Aktivität von RNA, was zur Postulierung einer RNA-Welt in der sehr frühen Phase der Entstehung des Lebens führte, wurde Thomas R. Cech zusammen mit Sidney Altman 1989 mit dem Nobelpreis für Chemie ausgezeichnet.[4] Spätere Untersuchungen konnten zeigen, dass Self-splicing-Introns in vielen weiteren Organismen auftreten. Entsprechend der Reaktionsmechanismen und der konservierten Sequenzelemente der RNAs können zwei Arten des self-splicings unterschieden werden, die sogenannten Gruppe-I und Gruppe-II-Introns. Auch wenn schlüssig gezeigt werden konnte, dass die RNA die katalytische Aktivität besitzt, scheinen in vivo an den Reaktionen beider Gruppen von Introns zusätzlich Proteine beteiligt zu sein, die wahrscheinlich das Ausbilden der aktiven Struktur der RNA fördern. Da bei den im Folgenden geschilderten Reaktionen die Gesamtzahl der Phosphodiesterbindungen stets gleich bleibt, weil es sich um Umesterungen handelt, sind keine energieliefernden Cofaktoren notwendig.
Gruppe-I-Introns treten in der prä-rRNA einfacher Eukaryoten, wie z. B. dem bereits erwähnten Ciliaten T. thermophila, sowie in einigen prä-mRNAs von Zellorganellen wie Mitochondrien und Chloroplasten auf. Die Exzision des Introns findet in einem Zweischrittmechanismus statt, wobei ein als Cofaktor für die Reaktion essentielles Guanosin, das durch die Struktur der RNA in die geeignete Position gebracht wird, zunächst einen nucleophilen Angriff auf die 5’-splice-site durchführt. Die nucleofuge Gruppe dieser Reaktion, die 3’-Hydroxygruppe des 5’ gelegenen Exons, greift nun seinerseits als Nucleophil die 3’-splice-site an, wodurch die beiden Exons unter Freisetzung des Introns miteinander verknüpft werden. In darauf folgenden Reaktionen schließt sich das Intron schließlich zu einem Ring. Stereochemische Untersuchungen an chiralen Substraten lassen vermuten, dass hier ein einziges katalytisches Zentrum beide Teilreaktionen des Splicings in Form einer Hin- und Rückreaktion katalysiert.
Gruppe-II-Introns finden sich dagegen in prä-mRNAs der Mitochondrien von Hefen und anderen Pilzen und in manchen RNA-Vorläufern der Chloroplasten mancher einzelliger Eukaryoten wie Chlamydomonas. Ein Guanosin-Cofaktor ist hier nicht notwendig, vielmehr wird durch die Struktur der RNA ein 7 oder 8 Nukleotide upstream der 3’ splice-site gelegenes Adenosin in die Lage versetzt die 5’ splice-site nucleophil mit seiner 2’ Hydroxy-Gruppe angreifen zu können. Dies führt zur Knüpfung einer ungewöhnlichen 2’ 5’ Phosphodiesterbindung und damit zur Ausbildung einer lassoartigen Struktur des Introns, dem sogenannten Lariat. In einer zweiten Reaktion, ähnlich der der Gruppe-I-Introns, greift schließlich die 5’-splice-site die 3’ splice-site nucleophil an, was zur Verknüpfung beider Exons und dem Freisetzen des Introns führt.
Beim spliceosomalen Prozessieren von mRNAs (siehe unten) findet sich ein dem der Gruppe-II-Introns identischer Reaktionsmechanismus, was zu einer Reihe von Spekulationen geführt hat, ob beide Prozesse evolutionär auseinander hervorgegangen sind (siehe unten bei der „Intron-early“-Hypothese), beispielsweise durch Fragmentierung eines Gruppe-II-Introns, oder ob es sich um konvergente Entwicklungen bedingt durch die katalytische Optimierung der gleichen chemischen Reaktion handelt.
Analog zum Spleißen von RNA ist das Proteinspleißen definiert.
Splicen von tRNAs
Das enzymatische Splicing von tRNAs findet sich sowohl bei Archaea als auch bei Eukaryoten, in Bakterien dagegen werden Introns in tRNAs nach einem autokatalytischen Mechanismus prozessiert, der im vorangegangenen Abschnitt beschrieben wurde. Die Introns in den für die tRNAs codierenden Genen finden sich meist in der Anticodonschleife direkt 3’ des Anticodons – seltener in der Dihydrouracilschleife – und besitzen eine Länge von 14 bis 60 Basen. Beim enzymatischen Splicing werden sie im Gegensatz zum Splicing von prä-mRNAs nicht über ihre Sequenz, sondern über eine übergeordnete Struktur des Gesamtmoleküls erkannt (beispielsweise das bulge-helix-bulge-motif – BHB-motif – bei Archaea) und in drei Schritten entfernt. Dabei wird die prä-tRNA zunächst zweimal durch eine Endonuklease geschnitten, die das Intron und zwei sogenannte tRNA-Halbmoleküle freisetzt. Das dabei entstehende zyklische 2’ 3’ Phosphat des 5’ Halbmoleküls wird anschließend zu einem 2’ Phosphat und einer 3’ OH-Gruppe hydrolysiert, während die 5’ OH Gruppe des 3’ Halbmoleküls unter GTP-Verbrauch phosphoryliert wird. Dies ermöglicht eine Ligation durch eine RNA-Ligase unter ATP-Hydrolyse. Schließlich wird im letzten Schritt das 2’ Phosphat entfernt, was ungewöhnlicherweise unter NAD-Verbrauch und Freisetzung von Nicotinamid vonstattengeht. Auch einige mRNAs werden nach einem ähnlichen, für sie eigentlich sehr untypischen Mechanismus aus zwei endonukleolytischen Spaltungen mit anschließender Ligation durch eine tRNA-Ligase prozessiert.
Splicing im Spliceosom
Das Splicen findet in den meisten Fällen in einem großen Komplex aus RNA und Proteinen statt, dem sogenannten Spliceosom, welches die Reaktion in zwei aufeinanderfolgenden Umesterungen katalysiert. Die Mehrzahl der Introns wird auf diese Art und Weise entfernt. Die Anzahl der Bindungen bleibt bei der Reaktion insgesamt gleich, Energie wird nur für den Aufbau und die Umlagerung der Maschinerie für die Katalyse (Spliceosom) benötigt. Die beiden Einzelreaktionen unterscheiden sich chemisch nicht voneinander, lediglich die Positionen der beteiligten Gruppen in der prä-mRNA sind unterschiedlich. Bei beiden Reaktionen findet eine nukleophile Substitution (SN2) an einem Phosphat statt, das Nukleophil ist jeweils eine Hydroxygruppe einer Ribose.
Im ersten Schritt greift das Sauerstoffatom der 2'-OH-Gruppe eines Adenosins aus der sogenannten "branch point sequence" (BPS) ein Phosphoratom einer Phosphodiesterbindung in der 5'-splice site an. Dies führt zur Freisetzung des 5'-Exons und zur Zirkularisierung des Introns (aufgrund der lassoartigen Struktur „Lariat“ genannt). Im zweiten Schritt greift nun der Sauerstoff der frei gewordenen 3'-OH-Gruppe des 5'-Exons die 3'-splice site an, was zur Verknüpfung der beiden Exons und zur Freisetzung des Intron-Lariats führt.[5]
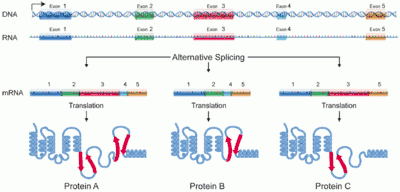
Das Splicing-Muster kann sich aufgrund der Gewebeart und unter Umwelteinflüssen unterscheiden. Man spricht von alternativem Splicing, einer wichtigen Grundlage für eine große Diversität von Proteinen. Das Splicing findet cotranskriptionell statt, was bedeutet, dass Introns bereits entfernt werden, noch während die Polymerase das Gen abliest.
Weitere wichtige Prozesse die während der Reifung einer prä-mRNA zur mRNA auftreten sind das
- Capping: Modifikation des 5'-Endes der RNA mit einem 7-Methylguanosin für eine bessere Stabilität der RNA und wichtig für die Translation am Ribosom.
- Tailing: Nach Erreichen des Genendes wird die RNA circa 15 Nukleotide nach einer speziellen Basensequenz (AAUAAA) geschnitten und mit einem ca. 150–200 Nukleotide langen Poly-A-Schwanz versehen. Auch hier spielt eine Vielzahl von Proteinen eine Rolle (CPSF-Komplex, Cstf-Komplex, CFI, CFII, PABP2, PAP etc.), die neben der erwähnten A2UA3-Sequenz weitere Elemente der RNA binden und das Prozessieren regulieren. Eine Termination der Transkription – ein leider nur sehr wenig verstandener Prozess in Eukaryoten – erfolgt wenig später downstream der Polyadenylierungs-Stelle u. a. durch den TREX-Komplex.
Schließlich wird die reife mRNA durch die Kernporen (nuclear pore complex, NPC) aus dem Zellkern in das Zytosol exportiert, wo sie anschließend im Verlauf der Translation dazu genutzt wird Proteine zu synthetisieren.
Splicing und Krankheiten
Auch bei einigen Krankheitsbildern spielt das Splicing eine große Rolle. Mutationen in Introns haben keinen direkten Effekt auf die Sequenz des Proteins, das durch ein Gen codiert wird. In einigen Fällen jedoch betreffen Mutationen Sequenzen, die für das Splicing wichtig sind und führen so zu einem falschen Prozessieren der prä-mRNA. Die so entstehenden RNAs codieren für unfunktionelle oder sogar schädliche Proteine und führen damit zu erblichen Erkrankungen.
Ein klassisches Beispiel sind einige Formen der β-Thalassämie, einer erblichen Hämoglobinopathie, bei denen eine Punktmutation die 5' splice-site von Intron 1 des HBB-Gens verändert und somit unbrauchbar macht. Dies führt dazu, dass nahegelegene „kryptische“ splice-sites erkannt werden und das Spliceosom verkürzte oder verlängerte mRNAs erzeugt, die in inaktive Proteine translatiert werden. Eine weitere gut untersuchte Mutation in Intron 2 des gleichen Gens führt zum Beibehalten einer kurzen Intron-Sequenz in der fertigen mRNA. In beiden Fällen kommt es zu einer stark reduzierten Hämoglobinsynthese des Allels in den erythrozytären Vorstufen. Sind beide Allele von einer solchen Mutation betroffen, entsteht das Krankheitsbild einer β-Thalassämia major, was u. a. zu einer deutlichen Blutarmut und einem ständigen Transfusionsbedarf führt.[6][7]
Weitere Fälle sind z. B. das Ehlers-Danlos-Syndrom (EDS) Typ II (Mutation eines branch-points im Gen COL5A1) und die spinale Muskelatrophie (Mutation eines Splicing-Enhancers/Silencers im Gen SMN1).
Die „RNA-Fabrik“ (RNA-factory)
In den letzten Jahren zeigte sich immer deutlicher, dass Transkription, Prozessierung der RNA (also Splicing, Capping und Tailing), RNA-Export in das Zytoplasma, RNA-Lokalisierung, Translation und RNA-Abbau einander beeinflussen und regulieren. Das Prozessieren der prä-mRNA findet noch während der Transkription statt – man spricht vom cotranskriptionellen RNA-Prozessieren – und die unterschiedlichen Maschinerien nehmen dabei miteinander Kontakt auf. Aus diesem Grund wurde kürzlich der Begriff „RNA-Fabrik“ (RNA-factory) geprägt, der dies veranschaulichen soll. Das Splicing kann darüber hinaus selbst auf die Prozesse Einfluss nehmen, die räumlich getrennt im Zytoplasma stattfinden. Ein Proteinkomplex, der durch das Spliceosom auf der fertigen mRNA abgesetzt wird (der Exon-Junction-Complex, EJC), ermöglicht einen effektiven Export aus dem Zellkern und übermittelt dabei zusätzlich eine Information, die eine spätere Qualitätskontrolle der RNA während der Translation ermöglicht (Nonsense-mediated mRNA decay, [NMD]). Eine weitere Implikation, die sich daraus ergibt ist: eine vollständige prä-mRNA (wie sie in der Abbildung oben dargestellt ist) kommt in der lebenden Zelle eigentlich nicht vor, denn Introns werden wie eben geschildert während der Transkription entfernt.
Splicing und Evolution
Viele Exons kodieren einen funktionellen Teil eines Proteins, der autonom faltet, eine sogenannte Domäne. Dies ist die Grundlage für die Theorie, dass ein modularer Aufbau eines Gens aus Exons, die solche Proteindomänen codieren, die Möglichkeit mit sich bringt, eine einmal evolutionär „erfundene“ Domäne durch Kombination mit anderen vielseitig einzusetzen. So kann durch einfache Rekombination von Exons nach einem Baukastenprinzip eine große Proteinvielfalt mit unterschiedlichsten Funktionen und Eigenschaften geschaffen werden, was als exon-shuffling bezeichnet wird. Ein klassisches Beispiel dafür ist das Gen für das Protein Fibronectin, das zum einen bei der Zelladhäsion, zum anderen aber auch bei Zellmigration, -proliferation und -differenzierung eine Rolle spielt. Das Protein besteht vorwiegend aus Wiederholungen dreier Proteindomänen, die darüber hinaus auch im Plasminogen-Aktivatorprotein (Typ I), in Proteinen der Blutgerinnung (Typ II), Zelloberflächenrezeptoren und Proteinen der extrazellulären Matrix (Typ III) gefunden werden können.
Darüber hinaus gibt es Vermutungen, dass Introns schon im letzten gemeinsamen, universellen Vorfahren (last universal common ancestor, ein Organismus aus dem sich die drei Reiche Bacteria, Archaea und Eukaryoten entwickelt haben) bereits vorhanden gewesen sein könnten. Diese Intron-early-Hypothese wird gestützt durch die Entdeckung unterschiedlicher Introns in den Genomen von Mitochondrien, Archaea und Viren. Bacteria müssten laut dieser Theorie ihre Introns eingebüßt haben, was durch eine Optimierung des Genoms für eine rasche Proliferation und kurze Generationsdauer erklärt werden könnte. Im Gegensatz dazu scheinen zumindest einige der Introns dieser Theorie nicht zu entsprechen, da sie sich vermutlich aus anderen Vorläufersequenzen entwickelt haben. Demnach mag es kein „Ur-Intron“ gegeben haben (aus dem alle heutigen Introns hervorgegangen sind), sondern vielmehr mehrere Sequenzen als Vorfahren für die heute bekannten Introns. Somit wären Introns nicht monophyletisch, sondern würden am ehesten einer polyphyletischen Gruppe entsprechen. Dieser Zusammenhang wird in der Intron-late-Hypothese formuliert.
Siehe auch
Literatur
- James E. Darnell, Harvey Lodish, David Baltimore: Molekulare Zellbiologie. de Gruyter, Berlin u. a. 1993, ISBN 3-11-011934-X (4. Auflage. Harvey Lodish: Molekulare Zellbiologie. Spektrum Akademischer Verlag, Heidelberg u. a. 2001, ISBN 3-8274-1077-0).
- Benjamin Lewin: Molekularbiologie der Gene. Spektrum Akademischer Verlag, Heidelberg u. a. 1998, ISBN 3-8274-0234-4.
- William S. Klug, Michael R. Cummings, Charlotte A. Spencer: Genetik. 8., aktualisierte Auflage. 2007, ISBN 978-3-8273-7247-5.
Weblinks
- mRNA splicing (Animation) aus Alberts u. a., Essential Cell Biology.
Einzelnachweise
- Louise T.Chow, James M.Roberts, James B.Lewis, Thomas R.Broker (1977): "An amazing sequence arrangement at the 5' ends of adenovirus 2 messenger RNA". Cell. 12 (1), 1–8. doi:10.1016/0092-8674(77)90180-5
- Susan M. Berget, Claire Moore, Phillip A. Sharp (1977): "Spliced segments at the 5' terminus of adenovirus 2 late mRNA". Proceedings of the National Academy of Sciences of the United States of America. 74 (8), 3171–3175. doi:10.1073/pnas.74.8.3171
- „Nobelpreis in Medizin, 1993“ Offizielle Website des Nobelpreiskomitees. Abgerufen am 28. Mai 2021.
- „Nobelpreis in Chemie, 1989“ Offizielle Website des Nobelpreiskomitees. Abgerufen am 18. Juni 2010.
- Sebastian M. Fica, Nicole Tuttle, Thaddeus Novak, Nan-Sheng Li, Jun Lu: RNA catalyses nuclear pre-mRNA splicing. In: Nature. Band 503, Nr. 7475, 14. November 2013, ISSN 0028-0836, S. 229–234, doi:10.1038/nature12734, PMID 24196718, PMC 4666680 (freier Volltext) – (nature.com [abgerufen am 5. Mai 2016]).
- Raffaella Origa: Beta-Thalassemia. In: GeneReviews®. University of Washington, Seattle, Seattle (WA) 1993, PMID 20301599 (nih.gov [abgerufen am 5. April 2020]).
- Antonio Cao, Renzo Galanello: Beta-thalassemia. In: Genetics in Medicine. Band 12, Nr. 2, Februar 2010, ISSN 1530-0366, S. 61–76, doi:10.1097/GIM.0b013e3181cd68ed (nature.com [abgerufen am 5. April 2020]).