Histonmodifikation
Histonmodifikationen sind chemische Veränderungen an Histon-Proteinen, die unter anderem Einfluss auf die Transkription haben.
Arten von Modifikationen
Histonmodifikationen wurden sowohl an den unstrukturierten N- und C-terminalen Enden der Histon-Proteine als auch in dem globulären Bereich innerhalb des Nukleosomen-Kerns gefunden.
Nomenklatur
Um Histonmodifikationen zu bezeichnen, hat sich folgende Nomenklatur entwickelt:
- Der Name des Histons (z. B. H3)
- Die betroffene Aminosäure in ihrem Einbuchstabencode (z. B. K für Lysin) mit der Position der Aminosäure im Protein
- Die Art der Modifikation (me: Methyl, P: Phosphat, Ac: Acetyl, Ub: Ubiquitin)
- Bei Methylierung kann zusätzlich die Anzahl der Methylgruppen (bei Lysinen und Argininen) als auch die Symmetrie (bei dimethylierten Argininen) angegeben werden.
Beispiele:
- Trimethylierung des Lysins an Position 4 des Histon 3: H3K4me3
- symmetrische Methylierung des Arginins 8 am Histon 3: H3R8me2s
- Acetylierung des Lysins an Position 20 am Histon 4: H4K20Ac
Acetylierung
Histon-Acetylierung findet ausschließlich an Lysinen statt (z. B. H3K9Ac, H3K27Ac, H4K16Ac, H4K20Ac). Die Hauptwirkung der Acetylgruppe ist die Neutralisierung der positiven Ladung des Lysins. Die Konsequenz ist eine Verringerung der elektrostatischen Wechselwirkung zwischen dem Lysin und den negativen Ladungen an der DNA. Dies führt zu einer Öffnung der 30 nm-Faser (Solenoidstruktur), was das Binden von Transkriptionsfaktoren sowie der Transkriptionsmaschinerie erlaubt, und so die Transkription begünstigt. Histon-Acetylierungen werden erzeugt durch Histon-Acetyltransferasen (HAT) und wieder entfernt durch Histon-Deacetylasen (z. B. HDAC4).
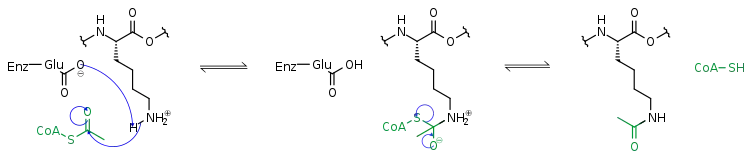
Methylierung
Histon-Methylierung findet man sowohl an Lysinen als auch an Argininen. Histon-Methylierung kann sowohl positiv als auch negativ mit Transkription korrelieren, je nachdem, welches Lysin/Arginin man betrachtet. Außerdem können Lysine mit ein, zwei oder drei und Arginine mit bis zu zwei Methylgruppen modifiziert sein. Diese verschiedenen Methylierungszustände sind oft unterschiedlich im Genom verteilt und haben daher wahrscheinlich auch unterschiedliche biologische Funktionen. Histon-Methylierung wird erzeugt durch Histon-Methyltransferasen (HMT) und entfernt durch Histon-Demethylasen (HDM). Mechanistisch liegt dem Prozess ein nucleophiler Angriff der Aminogruppe des Restes zugrunde. Der Methyldonor ist dabei meist (S)-Adenosylmethionin (SAM).[1]
Wichtige Methylierungen sind:
- H3K4me2/3 (findet man an den Promotoren von aktiv transkribierten Genen)
- H3K27me3 (findet man an reprimierten Genen)
- H3K9me3 (findet man im Heterochromatin)
- H3K4me1/2 (findet man an aktiven Enhancern)
- H3K36me3, H4K20me1 (findet man im Gen-Körper von aktiv transkribierten Genen)
Phosphorylierung
Histon-Phosphorylierung kann an Aminosäuren mit einer Hydroxygruppe stattfinden, also an Serinen, Threoninen und Tyrosinen. Histon-Phosphorylierungen sind in ihrer Funktion ähnlich divers wie Histon-Methylierungen.
Weitere bekannte Modifikationen
- Propionylierung
- Butyrylierung
- Ubiquitinylierung
- ADP-Ribosylierung
- Sumoylierung
- Carbonylierung
- Glycosylierung
- Biotinylierung
- cis-trans-Isomerisierung an Prolinen
- Citrullinierung an Argininen
Histon-Code
Neben dem direkten Einfluss auf die Chromatin-Struktur, wie zum Beispiel durch Acetylierung, scheinen viele Histonmodifikationen nur indirekt Einfluss auf biologische Prozesse zu haben. Die Entdeckung einer Vielzahl von Proteinen, die bestimmte Histonmodifikationen (insbesondere Methylierungen) erkennen können ("Histone Reader"), lassen den Schluss zu, dass viele Modifikationen als Bindestelle für Proteine dienen, die die Information in nachfolgende Prozesse übersetzen. Da jedes Nukleosom eine große Zahl potentieller Modifikationsstellen hat, und diese wiederum mehrere verschiedene Modifikationen aufweisen können (z. B. kann ein Lysin unmodifiziert, acetyliert, mono-, di- oder trimethyliert sein), kann ein einzelnes Nukleosom eine enorme Anzahl von verschiedenen Kombinationen besitzen. Man spricht in diesem Zusammenhang auch von der Histon-Code-Hypothese. Die Hypothese besagt, dass die Kombination verschiedener Histon-Modifikationen durch bindende Proteine abgelesen und deren Zusammenwirken zu bestimmten biologischen Prozessen führt. Die Richtigkeit dieser Hypothese ist derzeit Gegenstand intensiver Diskussion.
| Histon | Modifikation[Anm. 1] | Heterochromatin | Euchromatin | Kombinatorik und Folgen |
|---|---|---|---|---|
| H3 | Lys4me | + | ||
| H3 | Lys9me(1-3) | + | HP1-(PcG-)Bindung[Anm. 2]; fördert DNAme, hemmt Ser10p, hemmt generell Lys-ac | |
| H3 | Lys-ac | + | ||
| H3 | Ser10p | (+)[Anm. 3] | (+) | hemmt Lys9me u.u.; fördert Lys9, 14ac und umgekehrt |
| H3 | Lys14ac | |||
| H3 | Lys27me | |||
| H3 | Lys36me | |||
| H4 | Arg3me | + | EsaI Bindung[Anm. 4]; fördert Lys5ac und umgekehrt | |
| H4 | Lys5ac | |||
| H4 | Lys12ac | + | ||
| H4 | Lys16ac | fördert His18p und umgekehrt | ||
| H4 | His18p | hemmt Lys20me | ||
| H4 | Lys20me | fördert Lys16ac |
- Bedeutung der Abkürzungen siehe bei Nomenklatur.
- PcG: Polycomb Group Protein
- Wo „+“-Symbole geklammert sind, hängt der Effekt von der Kombinatorik verschiedener Modifikationen ab
- Esa1: Acetyltransferase, ein aktivierendes Enzym
Die Interaktion von Histonen und DNA wird durch Histonmodifikationen gesteuert. Diese Modifikationen können die Chromatinstruktur verändern und so zu Veränderungen der Genaktivität führen. Somit können diese epigenetischen Mechanismen die Transkription einzelner Gene oder ganzer Gruppen von Genen beeinflussen.
Quellen
- T. Jenuwein, C. D. Allis: Translating the Histone Code In: Science. 293(5532), 2001, S. 1074–1080. PMID 11498575
- B. D. Strahl, C. D. Allis: The language of covalent histone modifications. In: Nature. 403(6765), 2000, S. 41–45. PMID 10638745
- Yu-Chieh Wang, Suzanne E Peterson, Jeanne F Loring: Protein post-translational modifications and regulation of pluripotency in human stem cells. In: Cell Research. Band 24, Nr. 2, Februar 2014, ISSN 1001-0602, S. 143–160, doi:10.1038/cr.2013.151, PMID 24217768, PMC 3915910 (freier Volltext) – (nature.com [abgerufen am 17. Februar 2021]).