Exon
Ein Exon (von engl. exon, gebildet von intron und der Vorsilbe ex des Ausdrucks expressed [region])[1] ist der Teil eines eukaryotischen Gens, der nach Spleißen (Splicing) erhalten bleibt. Demgegenüber stehen die Introns (englisch intragenic regions), die beim Spleißen herausgeschnitten und abgebaut werden. Das typische humane Gen enthält durchschnittlich acht Exons mit einer mittleren Länge der internen Exons von 145 Nukleotiden. Introns sind im Durchschnitt mehr als 10-mal so lang, in einigen Fällen sind sie sogar noch wesentlich länger.[2]
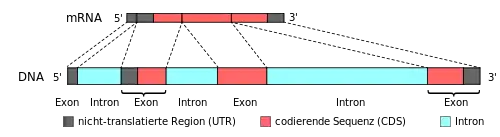
Die Exons Protein-codierender Gene enthalten den offenen Leserahmen (englisch open reading frame, ORF) und zusätzlich den 5' und 3' untranslatierten Bereich (englisch untranslated region, UTR) aus den terminalen Exons. Nur etwa 1,5 Prozent der gesamten genomischen Desoxyribonukleinsäure (DNA) codieren für Proteine (das haploide humane Genom beläuft sich auf rund 23.000 Protein-codierende Gene[3]), während der Rest aus Genen für nichtcodierenden Desoxyribonukleinsäuren (englisch non-coding DNA), sowie Introns, regulatorischer DNA und nichtcodierenden Desoxyribonukleinsäuren (sogenannte „junk“-DNA) besteht.[4] Da viele der Protein-codierenden Gene u. a. durch alternatives Splicing des Primärtranskripts (Präkursor-mRNA, prä-mRNA) eines Gens mehr als ein Protein produzieren, kommen im menschlichen Körper aber weit mehr als nur 23.000 verschiedene Proteine vor. Hierbei entscheidet sich erst während des Spleißvorgangs, welche DNA-Sequenzen Introns und welche Exons sind. Mit anderen Worten: Da nicht immer nach dem gleichen festen Muster gespleißt wird (vgl. Alternatives Spleißen), ist die genaue Angabe von Exons nur bedingt möglich, da je nach fertiger mRNA unterschiedliche Teile eines Gens als Exons definiert werden können. Eine genaue Voraussage von Exons mittels der Bioinformatik ist daher äußerst schwierig (vgl. Spleißstelle und Exon Trapping).
Darüber hinaus kennt man heute Proteine, die aus Exonen von Genen aus räumlich weit entfernten Regionen, mitunter sogar unterschiedlichen Chromosomen, aufgebaut sind.[5] Mithin ist die traditionelle Ein-Gen-ein-Enzym-Hypothese (auch: Ein-Gen-eine-mRNA-ein-Protein-Hypothese) für höhere Organismen heute nicht mehr haltbar.[6][7]
Polycistronische mRNA besteht aus multiplen ORFs in einem Transkript, mit kurzen Regionen von UTRs zwischen den ORFs.
Die regelmäßige Abfolge von Exons und Introns macht die typische Struktur der eukaryotischen Gene aus – das sogenannte Mosaikgen (englisch split gene) – für dessen Entdeckung Richard John Roberts und Phillip Allen Sharp 1993 mit dem Nobelpreis für Physiologie oder Medizin ausgezeichnet wurden.
Die Gesamtheit der Exone eines Organismus wird als Exom bezeichnet.
Geschichte
Der Begriff Exon wurde 1978 von dem Biochemiker Walter Gilbert geprägt: "The notion of the cistron… must be replaced by that of a transcription unit containing regions which will be lost from the mature messenger – which I suggest we call introns (for intragenic regions) – alternating with regions which will be expressed – exons."[8] Die Definition wurde ursprünglich für proteincodierende Transkripte eingeführt, später jedoch für ribosomale RNA (rRNA),[9] Transfer-RNA (tRNA)[10] und Transspleißen (englisch trans Splicing)[11] erweitert.
Weblinks
Einzelnachweise
- "The notion of the cistron [i.e., gene] ... must be replaced by that of a transcription unit containing regions which will be lost from the mature messenger – which I suggest we call introns (for intragenic regions) – alternating with regions which will be expressed – exons." (Gilbert 1978)
- E. S. Lander u. a.: Initial sequencing and analysis of the human genome. In: Nature. Band 15, Nr. 409, 2001, S. 860–921, PMID 11237011.
- International Human Genome Sequencing Consortium: Finishing the euchromatic sequence of the human genome. In: Nature. Band 431, Nr. 7011, 2004, S. 931–945, doi:10.1038/nature03001, PMID 15496913. nature.com
- International Human Genome Sequencing Consortium: Initial sequencing and analysis of the human genome. In: Nature. Band 409, Nr. 6822, 2001, S. 860–921, doi:10.1038/35057062, PMID 11237011. nature.com
- P. Kapranov u. a.: Examples of the complex architecture of the human transcriptome revealed by RACE and high-density tiling arrays. In: Genome Res. Band 15, Nr. 7, 2005, S. 987–997, PMID 15998911.
- J. L. Rupert: Genomics and environmental hypoxia: what (and how) we can learn from the transcriptome. In: High Alt Med Biol. Band 9, Nr. 2, 2008, S. 115–122, PMID 18578642.
- E. Pennisi: Genomics. DNA study forces rethink of what it means to be a gene. In: Science. Band 15, Nr. 316, 2007, S. 1556–1557, PMID 17569836.
- W. Gilbert: Why genes in pieces? In: Nature. Band 271, Nr. 5645, 1978, S. 501, doi:10.1038/271501a0, PMID 622185.
- K. P. Kister, W. A. Eckert: Characterization of an authentic intermediate in the self-splicing process of ribosomal precursor RNA in macronuclei of Tetrahymena thermophila. In: Nucleic Acids Research. Band 15, Nr. 5, März 1987, S. 1905–1920, doi:10.1093/nar/15.5.1905, PMID 3645543, PMC 340607 (freier Volltext).
- P. Valenzuela, A. Venegas, F. Weinberg, R. Bishop, W. J. Rutter: Structure of yeast phenylalanine-tRNA genes: an intervening DNA segment within the region coding for the tRNA. In: Proceedings of the National Academy of Sciences of the United States of America. Band 75, Nr. 1, Januar 1978, S. 190–194, doi:10.1073/pnas.75.1.190, PMID 343104, PMC 411211 (freier Volltext).
- A. Y. Liu, L. H. Van der Ploeg, F. A. Rijsewijk, P. Borst: The transposition unit of variant surface glycoprotein gene 118 of Trypanosoma brucei. Presence of repeated elements at its border and absence of promoter-associated sequences. In: Journal of Molecular Biology. Band 167, Nr. 1, Juni 1983, S. 57–75, doi:10.1016/S0022-2836(83)80034-5, PMID 6306255.