Proteom
Die Gesamtheit aller Proteine in einem Lebewesen, einem Gewebe, einer Zelle oder einem Zellkompartiment, unter exakt definierten Bedingungen und zu einem bestimmten Zeitpunkt, wird als Proteom bezeichnet (zum Beispiel Proteom des Menschen, der Kartoffelknolle, der Bakterienzelle, des Zellkerns).
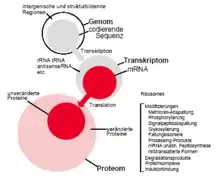
Das Proteom steht in einem Gleichgewicht ständiger Neusynthese von Proteinen bei gleichzeitigem Abbau nicht mehr benötigter Proteine. Damit ist das Proteom im Gegensatz zum relativ statischen Genom ständig Änderungen in seiner Zusammensetzung unterworfen. Diese Änderungen werden im Zuge der spatiotemporalen Genexpression über komplexe Regulationsprozesse gesteuert und werden maßgeblich durch Umweltstimuli, Krankheiten, Wirkstoffe und Medikamente beeinflusst. Das Proteom ist somit Spiegel seiner Umwelt und hoch dynamisch.
In Bakterienzellen umfasst das Proteom je nach Art um die 1000 bis 10.000 verschiedene Arten von Proteinmolekülen, beim Menschen rechnet man mit 500.000 bis 1.000.000 Proteinspezies. Es kann durchaus vorkommen, dass von einer proteincodierenden Gensequenz durch mRNA Splicing (nicht bei Bakterien) und nachträgliche Modifikationen des Primärproteins durch Enzyme bis zu mehrere Hundert Proteinspezies (Modifikationsformen) synthetisiert werden. Diese sogenannten posttranslationalen (nach der Proteinsynthese stattfindenden) Modifikationen können mit Hilfe von Techniken der Proteomanalyse erforscht werden.
Methoden
Für die Erforschung von Proteomen steht eine Vielzahl von Techniken zur Verfügung. Dabei kann zwischen Methoden für die Separation der einzelnen Proteinspezies und solchen für die Charakterisierung und Identifizierung derselben unterschieden werden. Die Wissenschaft von der Erforschung des Proteoms wird Proteomik (engl. proteomics) genannt.
Separation
- serielle Extraktionen
- serielle Fällungen
- Chromatographie
- Elektrophorese
- Zentrifugation
Identifikation und Charakterisierung
- Massenspektrometrie
- NMR-Spektroskopie
- Proteinsequenzierung per Edman-Abbau
- Anfärbung mit Antikörpern oder anderen selektiven Liganden
- direkte oder gekoppelte enzymatische Nachweise (z. B. colorimetrisch mit chromogenen Substraten)
- phänotypische Nachweise
Mit der Erforschung des Proteoms beschäftigt sich das Human Proteome Project (kurz HUPO) sowie in Deutschland die Deutsche Gesellschaft für Proteomforschung. Kommerziell wird die Protein-Muster-Diagnostik, außerhalb der klinischen Praxis, im Rahmen von Krebsdiagnostik und zur Diagnostik von Nierenerkrankungen angewandt.
Nach der weitestgehend abgeschlossenen Sequenzierung des gesamten menschlichen Genoms verspricht man sich von der Erforschung des Proteoms z. B. eine tiefe Einsicht in die Entstehung vieler Krankheiten und in weiterer Folge die Entwicklung kausal wirkender Medikamente. Die Ergebnisse der Proteomforschung finden sich in speziellen Proteom-Datenbanken, z. B. UniProt, GenBank, KEGG, Protein Data Bank, Reactome, Mascot und IEDB.
Geschichte
Der Begriff Proteom wurde 1994 vom Australier Marc R. Wilkins auf einem wissenschaftlichen Kongress zur Proteomforschung im italienischen Siena, in Anlehnung und Analogie zu den Begriffen Genom und Transkriptom, definiert.
Proteom des Menschen
In einer ersten umfassenden Studie wurde das Proteom von sieben menschlichen Zelllinien untersucht und die Schnittmenge daraus bestimmt, das so genannte zentrale Proteom. Dieses umfasste 1124 verschiedene Proteine oberhalb der Nachweisgrenze. Von zehn Prozent der Proteine war bei der Veröffentlichung die Funktion nicht bekannt. Die vom zentralen Proteom abgedeckten Stoffwechselbestandteile sind Proteinbiosynthese, Primärstoffwechsel, Zellzyklus und Apoptose. Die Daten werden auf proteomecommons.org zur Verfügung gestellt.[1]
2014 haben Forscher in Deutschland und den USA nahezu zeitgleich und unabhängig voneinander erstmals einen Katalog mit fast allen vom Menschen hergestellten Proteinen vorgelegt. In mehr als 30 Geweben untersuchten beide Forschergruppen, welche Proteine in den jeweiligen Zellen hergestellt werden. Dabei erfassten die Wissenschaftler der Johns Hopkins University School of Medicine in Baltimore (Maryland) 17.294 Proteine und damit etwa 84 % des Proteoms[2] und die deutsche Gruppe der TU München mehr als 18.000 Proteine und damit 92 % desselben[3]. Inzwischen ist bekannt, dass der Mensch etwa 20.344[4] – 20.356[5] vermutete proteincodierende Gene besitzt, darunter 8.874 in allen Geweben vorkommende Haushaltsgene, einige in allen Geweben vorkommende regulierte Gene und 6.942 gewebsspezifische Gene.[4] Etwa 80 % der proteincodierenden Gene erzeugen darüber hinaus Spleißvarianten.[6] Nach der Lokalisation eingeteilt sind es etwa 3.171 Gene (16 %) von sezernierten Proteinen, 5.570 (27 %) von Membranproteinen, 12.467 (61 %) von intrazellulären Proteinen und 852 Gene von Proteinen, die sowohl membrangebundene als auch sezernierte Isoformen besitzen.[5]
Bislang ist die Frage nicht vollständig aufgeklärt, welche Gene den Bauplan für genau welche Proteine bereithalten, unter welchen Bedingungen sie abgelesen werden. Damit sollen vertiefende Erkenntnisse über das Zusammenspiel von DNA, RNA und Proteinen als den molekularen Hauptakteuren des Lebens gewonnen werden. Etwa 1832 Proteine wurden nicht aufgefunden,[4] die laut Genkarte eigentlich existieren sollten. Es wird vermutet, dass eine Reihe dieser Proteine unter anderem nur in der Embryonalentwicklung vorhanden sind, viele bekannte Gene aber auch funktionslos geworden sind und nicht mehr als Vorlage für die Proteinsynthese dienen. Nach jetziger Erkenntnis trifft letzteres unter anderem auf Geruchsrezeptoren zu, weil der Geruchssinn für den Menschen nicht mehr überlebenswichtig sei. Andererseits sind fast 200 Proteine entdeckt worden, deren Funktion und Eigenschaften weiterhin unbekannt sind und die nicht von den bekannten proteinkodierenden Genen, sondern von DNA-Bereichen außerhalb dieser Gene produziert werden. Diese oft als "Junk-DNA" bezeichneten Bereiche des Genoms sollten nach bisherigen Erkenntnissen zwar eine regulierende Funktion für die Genaktivität haben, nicht aber ihrerseits eigene Proteine kodieren. Nach Akhilesh Pandey könnte das bedeuten, dass bisher noch nicht vollständig verstanden ist, wie Zellen die DNA lesen[7]. Des Weiteren wurden 24 Krebsmedikamente untersucht, deren Wirksamkeit auf 35 Krebszelllinien klar mit deren Proteinprofilen in Zusammenhang stand. Die Forscher hoffen, dass mit Kenntnis des Proteinprofils eines Tumors Medikamente zukünftig zielgerichteter eingesetzt werden könnten.
Weblinks
Einzelnachweise
- Thomas R. Burkard u. a.: Initial characterization of the human central proteome. In: BMC systems biology. Band 5, 2011, S. 17, doi:10.1186/1752-0509-5-17, PMID 21269460.
- Min-Sik Kim1, Sneha M. Pinto, Derese Getnet, et al.: A draft map of the human proteome. In: Nature. 29. Mai 2014, Nr. 509, S. 575–581, doi:10.1038/nature13302.
- Mathias Wilhelm, Judith Schlegl, Hannes Hahne1 et al.: Mass-spectrometry-based draft of the human proteome. In: Nature. 29. Mai 2014, Nr. 509, S. 582–587, doi:10.1038/nature13319.
- The human tissue specific proteome, Proteinatlas.org. Abgerufen am 7. November 2014.
- The human secretome and membrane proteome, Proteinatlas.org. Abgerufen am 7. November 2014.
- The human isoform proteome, Proteinatlas.org. Abgerufen am 7. November 2014.
- Nadja Podbregar: Erster Blick ins Proteom des Menschen. Auf: wissenschaft.de vom 28. Mai 2014.