Intron
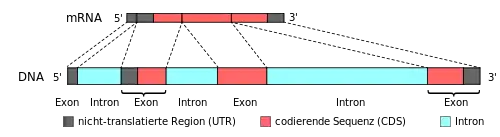
Introns (englisch intragenic regions) sind die nicht codierenden Abschnitte der DNA innerhalb eines Gens (intragen), die benachbarte Exons trennen. Introns werden transkribiert, aber dann aus der prä-mRNA herausgespleißt, bevor diese zur Translation aus dem Zellkern herausgeschleust wird. Die in der reifen mRNA verbleibenden Teile des Gens nennt man Exons. Die Aufteilung des Gens in Intron und Exon gehört zu den Hauptcharakteristika von eukaryotischen Zellen.
Introns können „alten Code“ enthalten, also (duplizierte) Teile eines Gens, die im Verlauf der Stammesgeschichte funktionslos geworden sind. Da sie keine direkte Bedeutung für die Struktur der Translationsprodukte besitzen, tendieren sie in höherem Maße zur Akkumulation von Mutationen als Exons. Bei höheren Eukaryoten nehmen sie einen beträchtlichen Anteil des Genomes ein. Manche nehmen an, dass es sich um Überbleibsel von viralen Infekten handelt, bzw. um "defekte" endogene (Retro)viren. Beim Menschen machen diese Bestandteile mindestens 50 % des Genoms aus.[1]
Introns spielen eine Rolle beim Alternativen Spleißen eines Gens, so dass ein Gen mehrere, in Abschnitten unterschiedliche Proteine hervorbringen kann. In diesen Fällen entscheidet erst der Spleißprozess, ob eine DNA-Sequenz als Intron oder Exon behandelt wird.
Eine Spezialrolle kommt den selbstspleißenden Introns (Ribozymen) zu, die sich quasi selbst aus der mRNA entfernen.
Das Verhältnis von intronischer zu exonischer DNA variiert stark zwischen unterschiedlichen Arten. Der Kugelfisch Takifugu rubripes zum Beispiel wurde aufgrund seines sehr geringen Anteils an Introns auch im Vergleich zu verwandten Arten schon früh sequenziert.
Man kann die Introns als eine Teilmenge der so genannten noncoding DNA betrachten, die die Gesamtmenge aller nichtcodierenden DNA-Anteile ist[2].
Die Mosaikgene, bei denen codierende DNA-Bereiche (Exon) durch nichtcodierende (Intron) DNA-Bereiche getrennt sind, wurden 1977 unabhängig voneinander durch Richard J. Roberts und Phillip Allen Sharp erkannt, wofür sie 1993 den Nobelpreis für Medizin erhielten.[3]
Geschichte
Der Begriff Intron wurde 1978 von dem Biochemiker Walter Gilbert geprägt: "The notion of the cistron… must be replaced by that of a transcription unit containing regions which will be lost from the mature messenger – which I suggest we call introns (for intragenic regions) – alternating with regions which will be expressed – exons."[4] Die Definition wurde ursprünglich für proteincodierende Transkripte eingeführt, später jedoch für rRNA[5], tRNA[6] und trans Spleißen[7] erweitert.
Intronphasen
Introns können an quasi jeder Stelle des Transkriptes liegen, auch mitten in einem Dreierblock, der in der Translation als Codon fungiert. Liegt ein Intron zwischen der dritten Base eines Codons und der ersten des nächsten Codons (also zwischen zwei Codons), so spricht man von Phase-0-Introns. Liegt das Intron zwischen dem ersten und dem zweiten Nukleotid eines Codons, so spricht man von Phase-1-Introns und zwischen der zweiten und dritten Base von einem Phase-2-Intron. Dies ist wichtig, wenn es zu Exonduplikationen kommt. Ein Exon, das zwischen zwei Introns der gleichen Phase liegt („symmetrisches Exon“ genannt), kann problemlos dupliziert werden, ohne dass es zu einer Rasterverschiebung (Frameshift) kommt. Asymmetrische Exons, die zwischen zwei Introns unterschiedlicher Phase liegen, sind nicht duplizierbar.
Arten von Introns
Je nachdem, ob der Spleißvorgang autonom verläuft oder durch einen Riboproteinkomplex (das Spliceosom) geschieht, unterscheidet man zwischen selbstspleißenden und spleißosomalen Introns.
Selbstspleißende Introns
Bei den selbstspleißenden Introns, die 1981 von der Arbeitsgruppe um Thomas R. Cech entdeckt wurden,[8] unterscheidet man wiederum:
- Gruppe-I-Introns
- Gruppe-II-Introns
Näheres siehe unter Spleißen.
Spleißosomale Introns
Bei diesen Introns muss das Herausschneiden durch das Spliceosom erfolgen. Ob eine Sequenz beim Spleißen als Intron oder als Exon erkannt wird, hängt von der Sequenz ab.
GU-AG-Introns
Die häufigsten Introns sind die sogenannten GU-AG-Introns. Sie beginnen üblicherweise mit dem Donator GU (Guanin–Uracil) und enden mit dem Akzeptor AG (Adenin–Guanin).
2007 wurden bei 36 Genen des Menschen Introns gefunden, die ungewöhnlicherweise am rechten Ende auf UG (Uracil–Guanin) enden. Man nimmt bisher an, dass dadurch das Spleißen der mRNA verzögert wird, was diese wieder reifen lassen soll und erwartet von weiteren Forschungsarbeiten neue Therapieansätze gegen Erbkrankheiten und Krebs.[9]
Introns in der Science-Fiction
Introns werden in der Episode Genesis (7x19) der Science-Fiction-Serie Raumschiff Enterprise: Das nächste Jahrhundert dargestellt als genetische evolutionäre „Überbleibsel“, die die Erbinformation von Urformen von Spezies tragen. Durch einen Unfall entwickelt sich die Raumschiffmannschaft zu diesen Urformen zurück.[10][11]
Auch in der Serie Outer Limits – Die unbekannte Dimension sind in der Episode Genetische Botschaft (3x12) Introns ein Thema. Ein Wissenschaftler führt dort Gen-Experimente und einen Selbstversuch durch. Dies führt zu außergewöhnlichen Veränderungen an seinem Körper, insbesondere am Gehirn.
Weblinks
Spektrum.de: Vermeintlich überflüssige Introns verhindern Verhungern 17. Januar 2019
Einzelnachweise
- L. P. Villarreal: Viruses and the Evolution of Life. ASM-Press, Washington 2005.
- G. Witzany: Non-coding RNAs: persistent viral agents as modular tools for cellular needs. In: Ann NY Acad Sci 1178, 2009, S. 244–267.
- Informationen der Nobelstiftung zur Preisverleihung 1993 an Richard John Roberts (englisch)
- W. Gilbert: Why genes in pieces?. In: Nature. 271, Nr. 5645, 1978, S. 501. doi:10.1038/271501a0. PMID 622185.
- K. P. Kister, W. A. Eckert: Characterization of an authentic intermediate in the self-splicing process of ribosomal precursor RNA in macronuclei of Tetrahymena thermophila. In: Nucleic Acids Research. 15, Nr. 5, März 1987, S. 1905–20. doi:10.1093/nar/15.5.1905. PMID 3645543. PMC 340607 (freier Volltext).
- P. Valenzuela, A. Venegas, F. Weinberg, R. Bishop, W. J. Rutter: Structure of yeast phenylalanine-tRNA genes: an intervening DNA segment within the region coding for the tRNA. In: Proceedings of the National Academy of Sciences of the United States of America. 75, Nr. 1, Januar 1978, S. 190–4. doi:10.1073/pnas.75.1.190. PMID 343104. PMC 411211 (freier Volltext).
- A. Y. Liu, L. H. van der Ploeg, F. A. Rijsewijk, P. Borst: The transposition unit of variant surface glycoprotein gene 118 of Trypanosoma brucei. Presence of repeated elements at its border and absence of promoter-associated sequences. In: Journal of Molecular Biology. 167, Nr. 1, Juni 1983, S. 57–75. doi:10.1016/S0022-2836(83)80034-5. PMID 6306255.
- K. Kruger et al.: Self-splicing RNA: autoexcision and autocyclization of the ribosomal RNA intervening sequence of Tetrahymena. In: Cell November 1982, S. 147–157 (PDF; 1,6 MB).
- Genesis im Star-Trek-Wiki Memory Alpha
- Intron im Star-Trek-Wiki Memory Alpha