Cotranslationaler Proteintransport
Als cotranslationaler Proteintransport oder cotranslationale Translokation wird in der Zellbiologie ein Vorgang bezeichnet, bei dem schon während der Translation die sich bildende Polypeptidkette eines Proteins in bzw. durch eine Biomembran transportiert wird. Davon unterschieden wird ein posttranslationaler Proteintransport genannter Vorgang, mit dem erst nach der Translation das somit vollständig gebildete Protein transportiert wird.
Vorkommen
Bei Eukaryoten kommt cotranslationaler Proteintransport vor allem bei Transport durch oder in die Membran des endoplasmatischen Retikulums (ER) vor. Durch Vesikeltransport können sich die membrangebundenen Proteine in die Membranen anderer Organellen oder in die Cytoplasmamembran über den sekretorischen Weg verteilen. Die aus diesem Grund an der ER-Membran sitzenden Ribosomen geben dem rauen ER seinen Namen.
Bei Prokaryoten kommt cotranslationaler Proteintransport an der Cytoplasmamembran vor.
Mechanismus
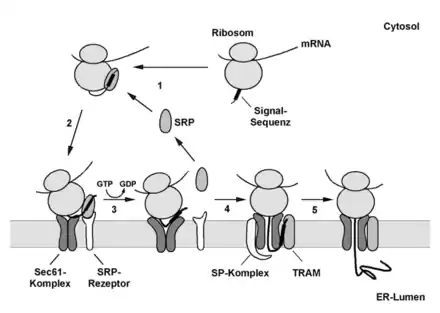
Der Transportprozess beginnt mit der spezifischen Erkennung der N-terminalen Signalsequenz. Sobald diese an der Oberfläche des Ribosoms erscheint, wird sie vom Signalerkennungspartikel (signal recognition particle, SRP) gebunden, das im Säuger aus sechs Proteinen und einer 7SL-RNA besteht. Das gebundene SRP interagiert dabei nicht nur mit der Signalsequenz, sondern auch mit dem Ribosom, wodurch die Synthesegeschwindigkeit der naszierenden Polypeptidkette herabgesetzt wird. SRP ist somit in der Lage, das Ribosom zur ER-Membran zielzuleiten, ohne dass vorher das zu transportierende Protein fertiggestellt wird. An der ER-Membran angelangt, interagiert der Komplex aus Ribosom, naszierender Peptidkette und SRP auf zweifache Weise mit der Membran. Zum einen wird das SRP von seinem membranständigen Rezeptor (SRP-Rezeptor) gebunden, zum anderen interagiert das Ribosom direkt mit dem tunnelbildenden Sec61-Komplex (bei Säugern bestehend aus Sec61A1, Sec61B und Sec61G). Nachdem SRP und SRP-Rezeptor aneinander gebunden haben, löst sich das SRP unter GTP-Hydrolyse vom Ribosom und übergibt die naszierende Polypeptidkette an den eigentlichen Translokationsapparat. Die genauen Prozesse sind noch Gegenstand der Forschung.
Im Säugersystem konnte in Rekonstitutionsexperimenten gezeigt werden, dass neben dem SRP-Rezeptor nur noch zwei weitere Komponenten in der Membran benötigt werden, um Proteine in vitro cotranslational zu transportieren. Es handelt sich dabei um das TRAM-Protein (translocating chain-associated membrane protein) und um den heterotrimeren Sec61-Komplex. Quervernetzungsexperimente sowie elektronenmikroskopische Untersuchungen zeigten, dass der Sec61-Komplex den Kanal bildet, durch den die Proteine hindurchtransportiert werden. Der Kanal wird dabei von drei bis vier heterotrimeren Sec61-Komplexen gebildet, die im elektronenmikroskopischen Bild als ringförmige Struktur erscheinen.
Die Insertion der naszierenden Kette in den Translokationskanal erfolgt in zwei Schritten. Zu Beginn des Translokationsprozesses ist die Bindung zwischen Sec61-Komplex und Ribosom relativ schwach. Die Ribosomen können durch eine Hochsalzbehandlung von der Membran abgewaschen werden. Erst wenn die naszierende Kette eine Länge von ca. 70 Aminosäuren erreicht hat und eine funktionelle Signalsequenz vorhanden ist, erhöht sich die Bindung zwischen Ribosom und Sec61-Komplex – selbst unter Hochsalzbedingungen lösen sich nicht Ribosomen von der Membran ab. Gleichzeitig öffnet sich der Translokationskanal zur luminalen Seite des ER. Durch elektronenmikroskopische Aufnahmen konnte gezeigt werden, dass das Ribosom so auf der Membranpore sitzt, dass der Sec61-Kanal eine Verlängerung des Ribosomenkanals darstellt. Die GTP-abhängige Synthese der naszierenden Kette am Ribosom reicht als alleinige Kraft aus, um die naszierende Polypeptidkette ins Lumen des ER zu transportieren.
- Die Signalsequenz der wachsenden Polypeptidkette wird vom SRP erkannt und gebunden. Gleichzeitig interagiert das SRP auch mit dem Ribosom, wodurch es zu einem Elongationsarrest kommt.
- Der Komplex aus Ribosom, naszierender Polypeptidkette und SRP bindet an die ER-Membran an den Sec61-Komplex, diese Bindung wird durch einen membranständigen SRP-Rezeptor vermittelt.
- Nach GTP-Hydrolyse löst sich SRP vom Ribosom, dieses wird auf den Sec61-Komplex übertragen. Es erfolgt ein zweiter Signalsequenzerkennungsschritt durch den Sec61-Komplex.
- Der Translokationskanal öffnet sich zum ER-Lumen. Der N-Terminus der naszierenden Kette liegt in einer Haarnadelkonformation vor, wobei der eine Teil der Schleife von der Signalsequenz und der andere Teil von den C-terminal nachfolgenden Abschnitten der Polypeptidkette gebildet wird. Die Signalsequenz interagiert dabei mit Sec61a, TRAM und Membranlipiden. Durch die weitere Elongation der Polypeptidkette wird der C-terminale Bereich der Schleife durch die Membran geschoben. Die Signalsequenz wird dabei vom restlichen Protein durch den Signalpeptidasekomplex (SP-Komplex) abgespalten.
- Anschließend wird die restliche naszierende Polypeptidkette direkt durch den von Ribosom und Sec61-Komplex gebildeten Kanal ins Lumen des ER transportiert.
Literatur
- T. U. Schwartz: Origins and evolution of cotranslational transport to the ER. In: Adv Exp Med Biol. Band 607, 2007, S. 52–60. PMID 17977458; doi:10.1007/978-0-387-74021-8_4
- C. Rabu, V. Schmid, B. Schwappach, S. High: Biogenesis of tail-anchored proteins: the beginning for the end? In: J Cell Sci. Band 122(Pt 20), 2009, S. 3605–3612. PMID 19812306; PDF (freier Volltextzugriff, englisch)
- R. Zimmermann, S. Eyrisch, M. Ahmad, V. Helms: Protein translocation across the ER membrane. In: Biochim Biophys Acta. Band 1808, Nr. 3, 2011, S. 912–924. PMID 20599535; doi:10.1016/j.bbamem.2010.06.015
- I. Saraogi, S. O. Shan: Molecular mechanism of co-translational protein targeting by the signal recognition particle. In: Traffic. Band 12, Nr. 5, 2011, S. 535–542. PMID 2129150; doi:10.1111/j.1600-0854.2011.01171.x
- S. Shao, R. S. Hegde: Membrane protein insertion at the endoplasmic reticulum. In: Annu Rev Cell Dev Biol. Band 27, 2011, S. 25–56. PMID 21801011; doi:10.1146/annurev-cellbio-092910-154125
- R. S. Hegde, R. J. Keenan: Tail-anchored membrane protein insertion into the endoplasmic reticulum. In: Nat Rev Mol Cell Biol. Band 12, Nr. 12, 2011, S. 787–798. PMID 22086371; doi:10.1038/nrm3226