Parvularcula bermudensis
Parvularcula bermudensis ist ein marines Bakterium, das 2003 in der westlichen Sargassosee entdeckt wurde. Bei der Art handelt es sich um ein gramnegatives, strikt aerobes, bewegliches Bakterium. Es benötigt Natriumchlorid (Kochsalz) für das Wachstum und ist somit halophil. Auf Marine-Agar bildet es kleine, gelb-braune, harte Kolonien. Das Genom des Stammes P. bermudensis HTCC2503 wurde 2010 vollständig sequenziert.
| Parvularcula bermudensis | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Parvularcula bermudensis | ||||||||||||
| Cho & Giovannoni, 2003 |
Parvularcula bermudensis gehört zu der Klasse der Alphaproteobacteria, unterscheidet sich jedoch von den bekannten Ordnungen, so dass es eine eigene Ordnung „Parvularculales“ innerhalb der Alphaproteobacteria bildet. Innerhalb der Ordnung stellt die Familie „Parvularculaceae“ die einzige Familie dar, mit der einzigen Gattung Parvularcula, somit handelt es sich bei der Familie und der Ordnung um ein monotypisches Taxon.
Merkmale
Erscheinungsbild
Die Zellen von Parvularcula bermudensis sind kurze Stäbchen, auch kokkoide Formen treten auf. Sie sind gramnegativ. Die Zellen sind 0,6–1,8 μm lang und 0,4–1,3 μm breit. Die Art ist durch eine monopolare Geißel schwach motil, kann sich also selbständig bewegen. Endosporen werden nicht gebildet.[1]
Auf festen Nährböden wachsen die Zellen zu sehr kleinen Kolonien heran, ihr Durchmesser liegt zwischen 0,3 und 0,8 mm. Diese sind gelb bis braun gefärbt, erscheinen opak und sind hart. In der Aufsicht sind die Kolonien rund geformt mit einer klaren Begrenzung, von der Seite betrachtet konvex. Die Kolonien erscheinen in der Oberfläche des Nährmediums eingesunken.[1]
Wachstum und Stoffwechsel
Der Stoffwechsel von Parvularcula bermudensis beruht auf der Atmung, die Art ist strikt aerob, benötigt also Sauerstoff zum Wachsen. Der Oxidase-Test verläuft positiv, Katalase lässt sich nicht nachweisen. Weiterhin ist der Stoffwechsel als chemoorganotroph und heterotroph zu kennzeichnen, P. bermudensis benutzt organische Verbindungen als Energiequelle und ebenso zum Aufbau zelleigener Stoffe. Der pH-Wert für bestes Wachstum ist 8,0. Wachstum erfolgt bei pH-Werten zwischen 6,0 und 9,0. Die optimale Temperatur für das Wachstum liegt bei 30 °C. Wachstum erfolgt innerhalb von 10 bis 37 °C, wobei es bei 10 °C etwa 40 Tage dauert, bis Kolonien erkennbar sind. P. bermudensis ist halophil und wächst in Nährmedien, die 0,75 bis 25 % Natriumchlorid (Kochsalz) enthalten. Optimal für das Wachstum ist ein Gehalt von 3,0 % Kochsalz im Nährmedium. Zur Kultivierung sind einfache Nährmedien nicht geeignet.[1] Stattdessen kann Marine-Agar verwendet werden, ein Nährmedium, das neben Pepton und Hefeextrakt noch Mineralsalze enthält, die in ihrer Zusammensetzung der von Meerwasser entsprechen.[2]
Biochemische Merkmale, wie beispielsweise die vorhandenen Enzyme können in einer „Bunten Reihe“ zur Identifizierung von P. bermudensis verwendet werden. Neben dem negativen Katalase- und dem positiven Oxidase-Test können folgende Merkmale herangezogen werden: Sie kann Nitrat zu Nitrit reduzieren, bei dieser Denitrifikation wird jedoch kein Gas (molekularer Stickstoff) gebildet. Der Urease-Test fällt positiv aus, die Art besitzt das Enzym Urease und ist somit in der Lage, Harnstoff abzubauen. Auch Gelatine wird durch Hydrolyse verwertet. Sie ist jedoch nicht zur Äskulinhydrolyse fähig. Sie verfügt nicht über das Enzym Arginindihydrolase (ADH) und kann daher die Aminosäure Arginin nicht abbauen. Der Indol-Test verläuft negativ.[1]
Im Rahmen des chemoorgano-heterotrophen Stoffwechsels kann P. bermudensis zahlreiche organische Verbindungen als Kohlenstoffquelle nutzen, dazu gehören Kohlenhydrate (Pentosen, Hexosen und Oligosaccharide), Zuckeralkohole und Aminosäuren. So wird unter aeroben Bedingungen beispielsweise Glucose genutzt, dabei wird keine Säure gebildet, wie es für eine Gärung typisch wäre. Weitere verwertbare Substrate sind z. B. D-Arabinose, L-Rhamnose, D-Mannose, Saccharose, D-Cellobiose, D-Maltose, D-Melezitose, D-Mannitol, D-Sorbitol und myo-Inositol, weiterhin die Aminosäuren Glutaminsäure, Lysin, Serin, Leucin und Isoleucin.[1]
Kohlenhydrate, die nicht genutzt werden können, sind beispielsweise D-Ribose, D-Xylose, D-Galactose, D-Fructose, L-Sorbose, β-Lactose, D-Trehalose, D-Melibiose und D-Raffinose. Zu den weiteren organischen Verbindungen, die P. bermudensis nicht verwerten kann, gehören u. a. Glycerin (Glycerol), Adonitol, Citrat, Gluconat, Lactat, D-Malat, Pyruvat und Succinat.[1]
Chemotaxonomische Merkmale
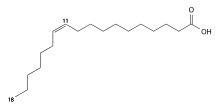
Parvularcula bermudensis produziert Pigmente, die zu der Gruppe der Carotinoide gehören. Bacteriochlorophyll a (ein Photosynthese-Pigment) kommt nicht vor. Die in den Membranlipiden vorkommenden Fettsäuren sind Moleküle mit einer geraden Zahl von Kohlenstoffatomen (C12 bis C18) und einer Doppelbindung (einfach ungesättigte Fettsäuren) oder keiner Doppelbindung (gesättigte Fettsäuren). Die mit 73 % hauptsächlich vorkommende Fettsäure trägt das Kürzel C18:1ω-7c und wird als cis-Omega-7-Octadecensäure oder cis-Vaccensäure bezeichnet.[1]
Genetik
Das Genom des Bakterienstammes P. bermudensis HTCC2503 wurde 2010 vollständig sequenziert und 2011 veröffentlicht. Dabei handelt es sich um den Stamm, der 2003 in der westlichen Sargassosee entdeckt wurde.[3] Das Genom weist eine Größe von 2903 Kilobasenpaaren (kb) auf,[4] das ist in etwa 60 % der Genomgröße von Escherichia coli und liegt als zirkuläres Bakterienchromosom vor.[3] Es sind 2685 Proteine annotiert.[5] Das Genom enthält u. a. Gene für die Biosynthese der Carotinoide und für eine Beta-Lactamase, ein Enzym, mit dem das Bakterium das Antibiotikum Penicillin und verwandte Substanzen zerstören kann.[3] Der GC-Gehalt (der Anteil der Nukleinsäuren Guanin und Cytosin) in der Bakterien-DNA liegt bei 61 Mol-Prozent.[4] Zuvor wurden für phylogenetische Untersuchungen die Nukleotide der 16S-rRNA bestimmt, ein für Prokaryoten typischer Vertreter der ribosomalen RNA.[6]
Pathogenität
Parvularcula bermudensis ist nicht pathogen („krankheitserregend“), sie wird durch die Biostoffverordnung in Verbindung mit der TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466 der Risikogruppe 1 zugeordnet.[7]
Systematik
Äußere Systematik
Äußere Systematik von Parvularcula bermudensis nach Cho und Giovannoni 2003[1]
|
Eine vorläufige Untersuchung der 16S-rRNA des isolierten Stammes HTCC2503 von Parvularcula bermudensis zeigte zunächst, dass der Bakterienstamm zur Klasse der Alphaproteobacteria gehört. Anschließend wurde die 16S-rRNA-Sequenz mit bekannten Sequenzen von typischen Vertretern der zum Zeitpunkt der Untersuchung bekannten sechs Ordnungen der Alphaproteobacteria verglichen. Die größte Ähnlichkeit ergab sich mit Aminobacter aminovorans in der Ordnung Rhizobiales und mit Silicibacter lacuscaerulensis in der Ordnung Rhodobacterales. Allerdings waren die phylogenetischen Unterschiede so deutlich, dass die untersuchten Bakterienstämme (neben dem Stamm HTCC2503 noch ein Umweltisolat mit der Bezeichnung „nicht kultivierter Klon H9“) eine monophyletische Gruppe bilden, die als eigene Ordnung neben den Rhizobiales und Rhodobacterales anzusehen ist.[1]
P. bermudensis ist eine von mehreren Arten aus der Gattung Parvularcula.[8] Aufgrund der phylogenetischen Untersuchungen wurde eine neue (zum Zeitpunkt der Entdeckung die siebte) Ordnung „Parvularculales“ innerhalb der Alphaproteobacteria etabliert.[1] Innerhalb der Ordnung stellt die Familie „Parvularculaceae“ die einzige Familie dar, mit der einzigen Gattung Parvularcula, somit handelt es sich bei der Familie und der Ordnung um ein monotypisches Taxon.[9] Gemäß der Systematik der Bakterien werden die Bezeichnungen in Anführungszeichen gesetzt, um darzustellen, dass diese Taxa noch nicht valide publiziert worden sind oder nicht durch den International Code of Nomenclature of Bacteria (ICNB) erfasst werden.
Von der Gattung sind folgende Arten bekannt (Stand 2014):[8]
- Parvularcula bermudensis Cho & Giovannoni 2003 (die Typusart)
- Parvularcula dongshanensis Yu et al. 2013
- Parvularcula lutaonensis Arun et al. 2009
Innere Systematik
Von Parvularcula bermudensis sind keine Synonyme bekannt.[10] Die Entdeckung der Art basiert auf der Untersuchung von zwei Bakterienstämmen. Die Stämme werden als HTCC2503 und HTCC2517 bezeichnet und weisen die gleichen phänotypischen und genotypischen Merkmale auf. Der Stamm P. bermudensis HTCC2503 ist der Typusstamm der Art.[1] Er wird auch unter der Bezeichnung ATCC BAA-594 geführt. Es sind mehrere Bakterienstämme von P. bermudensis in verschiedenen Sammlungen von Mikroorganismen hinterlegt.[11]
Etymologie
Der Gattungsname Parvularcula ist aus dem lateinischen Wort parvulus („sehr klein“) und arcula („Schmuckkasten“, „Juwelenkästchen“) zusammengesetzt. Der Artname P. bermudensis bezieht sich auf den Fundort des Typusstammes in der Sargassosee, in der Nähe der Bermudainseln.[1]
Entdeckungsgeschichte
Die Art wurde 2003 durch Jang-Cheon Cho und Stephen J. Giovannoni erstbeschrieben. Sie entnahmen im August 2001 in der Nähe der Bermudainseln in einer Tiefe von 10 m Proben von Meerwasser des Atlantischen Ozeans. Eine Vielzahl mariner Mikroorganismen lässt sich nicht einfach durch Kultivierung auf üblichen festen Nährböden isolieren. Für eine Identifizierung oder taxonomische Einordnung müssen aber Reinkulturen zur Verfügung stehen.[1]
Daher wurde in den 1990er Jahren eine spezielle Kultivierungsmethode entwickelt, die nicht mit den sonst in der Mikrobiologie üblichen Nährmedien arbeitet, sondern mit flüssigen Medien, die Nährstoffe nur in sehr geringen Konzentrationen aufweisen (englisch extinction culturing). Zur Kultivierung von Bakterien aus dem Meer wird dazu steriles Meerwasser verwendet, das in Spuren (0,001 %) mit organischen Verbindungen angereichert ist. Die Meerwasserprobe mit den enthaltenen Bakterienzellen wird in dieses Medium gegeben und davon eine Verdünnungsreihe hergestellt. Die Bedingungen für die Inkubation werden dem natürlichen Habitat angepasst, z. B. durch eine dunkle Umgebung, eine konstante Temperatur von 16 °C und eine Inkubationsdauer von mehreren Wochen. Anschließend wird mit Hilfe der Durchflusszytometrie die Zellzahl bestimmt und nur die Proben, in denen Zellen gefunden werden, werden weiter untersucht. Zu Beginn des 21. Jahrhunderts wurde diese Methode als Hochdurchsatz-Kultivierung (englisch high-throughput culturing, HTC) unter Verwendung von Mikrotiterplatten optimiert. Durch die Mikrotiterplatten werden die benötigten Volumina reduziert und gleichzeitig die Anzahl der Kultivierungsgefäße erhöht, da eine solche Platte üblicherweise 48 oder 96 Kavitäten enthält. Auf diese Weise lassen sich mit wenig Materialaufwand viele Untersuchungen durchführen.[12] Die nach diesem Verfahren durch Cho und Giovannoni als positiv erkannten Proben wurden dann mit Hilfe des Marine-Agars kultiviert und die Reinkulturen weiter untersucht und als neue Spezies bestimmt.
Quellen
Literatur
- Jang-Cheon Cho, Stephen J. Giovannoni: Parvularcula bermudensis gen. nov., sp. nov., a marine bacterium that forms a deep branch in the alpha-Proteobacteria. In: International journal of systematic and evolutionary microbiology. Band 53, Nr. 4, 2003, S. 1031–1036, doi:10.1099/ijs.0.02566-0 (PDF, 312 kB [abgerufen am 16. Februar 2014]).
Einzelnachweise
- Jang-Cheon Cho, Stephen J. Giovannoni: Parvularcula bermudensis gen. nov., sp. nov., a marine bacterium that forms a deep branch in the α-Proteobacteria. In: International journal of systematic and evolutionary microbiology. Band 53, Nr. 4, Juli 2003, S. 1031–1036, ISSN 1466-5026. doi:10.1099/ijs.0.02566-0. PMID 12892122.
- Marine Agar 2216, Marine Broth 2216. In: Website Becton, Dickinson and Company (BD). Abgerufen am 18. Februar 2014. (englischsprachige Gebrauchsanweisung, 2. Auflage; PDF; 656 kB).
- H. M. Oh, I. Kang u. a.: Complete genome sequence of strain HTCC2503T of Parvularcula bermudensis, the type species of the order "Parvularculales" in the class Alphaproteobacteria. In: Journal of bacteriology. Band 193, Nr. 1, Januar 2011, S. 305–306, ISSN 1098-5530. doi:10.1128/JB.01205-10. PMID 21037002. PMC 3019957 (freier Volltext).
- Parvularcula bermudensis HTCC2503. In: Webseite Genomes Online Database (GOLD). Abgerufen am 18. Februar 2014.
- Parvularcula bermudensis. In: Webseite Genome des National Center for Biotechnology Information (NCBI). Abgerufen am 18. Februar 2014.
- Parvularcula bermudensis HTCC2503 strain HTCC2503 16S ribosomal RNA, complete sequence. In: Webseite Nucleotide von Parvularcula bermudensis des National Center for Biotechnology Information (NCBI). Abgerufen am 18. Februar 2014.
- TRBA (Technische Regeln für Biologische Arbeitsstoffe) 466: Einstufung von Prokaryonten (Bacteria und Archaea) in Risikogruppen. In: Webseite der Bundesanstalt für Arbeitsschutz und Arbeitsmedizin (BAuA). 25. April 2012, S. 162, abgerufen am 7. Januar 2014.
- Jean Euzéby, Aidan C. Parte: Genus Parvularcula. In: List of Prokaryotic names with Standing in Nomenclature (LPSN). Abgerufen am 17. Februar 2014.
- Jean Euzéby, Aidan C. Parte: Phylum „Proteobacteria“. In: List of Prokaryotic names with Standing in Nomenclature (LPSN). Abgerufen am 17. Februar 2014.
- Taxonomy Browser Parvularcula bermudensis. In: Webseite des National Center for Biotechnology Information (NCBI). Abgerufen am 17. Februar 2014.
- Strain Passport Parvularcula bermudensis. (Nicht mehr online verfügbar.) In: Website StrainInfo (gesammelte Informationen über Bakterienstämme in über 60 Biologischen Forschungseinrichtungen (biological resource centers, BRCs)). Archiviert vom Original am 27. Februar 2014; abgerufen am 19. Februar 2014. Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis.
- S. A. Connon, S. J. Giovannoni: High-throughput methods for culturing microorganisms in very-low-nutrient media yield diverse new marine isolates. In: Applied and environmental microbiology. Band 68, Nr. 8, August 2002, S. 3878–3885, ISSN 0099-2240. doi:10.1128/AEM.68.8.3878-3885.2002. PMID 12147485. PMC 124033 (freier Volltext).