tmRNA
tmRNA ist die Kurzform für transfer-messenger-RNA. Es handelt sich um eine kleine, stabile Ribonukleinsäure (kurz RNA). Der Name ist eine Kombination aus tRNA (transfer-RNA) und mRNA (messenger-RNA). Namensgebend hierbei ist die Fähigkeit, Eigenschaften beider RNA-Typen zu vereinen. Weitere, aber veraltete, geläufige Namen sind 10S RNA und 10Sa RNA.
Vorkommen
Gene für tmRNA wurden in jedem bakteriellen Genom gefunden, dies schließt auch Bakterien mit erheblich reduziertem Erbgut ein. Dagegen wurde tmRNA weder in Archaeen noch – mit Ausnahme mancher Organellen – in Eukaryoten identifiziert.
Gen
Das Gen, welches die tmRNA codiert, heißt ssrA. Es wird wie andere tRNAs aus einem Vorläufertranskript synthetisiert und auch in ähnlicher Weise durch Ribonukleasen weiter prozessiert, beispielsweise durch RNase P oder RNase E. Bakterien, die die CCA-Sequenz am 5'-Ende nachträglich (posttranskriptionell) einführen, machen dies auch für ihre prä-tmRNAs. Dies passiert beispielsweise in Bacillus subtilis.
Das für die Funktion der tmRNA essentielle Protein SmpB wird durch das Gen smpB codiert.
In den meisten Bakterienspezies ist die tmRNA nicht essentiell, kann aber zu Wachstumsdefekten führen, beispielsweise nach Stressantworten.[1] Für Neisseria gonorrhoeae, Shigella flexneri, Mycoplasma genitalium oder Haemophilus influenzae ist die Aktivität der tmRNA dagegen überlebenswichtig.
Aufbau
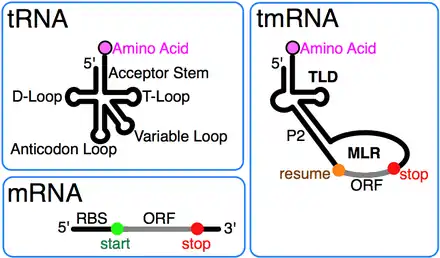
Der tRNA-Teil der tmRNA besitzt sowohl einen TΨC-Stamm als auch einen Akzeptor-Stamm, der mit der tRNA-typischen Sequenz CCA-5' endet. Es fehlen aber der Anticodonstamm und die D-Schleife. Ein spezieller offener Leserahmen (ORF) ähnelt einer mRNA und codiert für eine Proteinsequenz, die später an ein bestehendes Protein angehängt wird (siehe auch Abschnitt Funktion). In tmRNA wurden auch drei bis vier Pseudoknoten identifiziert, deren exakte Funktion noch unklar ist.
Die tRNA-Strukturelemente der tmRNA entsprechen denen einer tRNA, die Alanin erkennt (tRNAAla). So liegt im Akzeptorstamm das besondere G•C-Wobbel-Basenpaar vor, was zur Erkennung durch die Alanin-tRNA-Synthetase ausschlaggebend ist. Infolgedessen wird die tmRNA durch diese Alanin-tRNA-Synthetase mit Alanin beladen.
Für die Funktion einer normalen tRNA ist der Anticodonstamm essentiell. In der tmRNA wird diese durch ein besonderes Protein ersetzt: SmpB. Es muss an die tmRNA binden, damit diese seine Funktion erfüllen kann. Eine Struktur der tRNA-ähnlichen Domäne der tmRNA wurde in einem Komplex mit dem SmpB-Protein identifiziert.[2]
Arbeitsweise
Durch ihre Form ist die tmRNA wie eine tRNA in der Lage, an ein Ribosom im Wartezustand (englisch stalled ribosome) anzudocken und so den Translationsprozess wieder aufzunehmen. Hierbei wird der eigene mRNA-Teil der tmRNA als neues Leseraster benutzt. Der Translationsprozess wird nun mit diesem fortgesetzt, was man auch als trans-Translation bezeichnet. Dadurch wird die bisher hergestellte Peptidkette um eine spezielle Markierung in Form eines Peptidkettenrestes erweitert. Dieser Rest dient als Signal zum späteren Abbau. Ein Stopcodon auf dem mRNA-Teil bewirkt am Ende die Termination der Translation und somit die Freigabe des Ribosoms.
Funktionen
Qualitätskontrolle
Bei der Translation von mRNA an Ribosomen zu Proteinen kann es vorkommen, dass der mRNA ein Stopcodon fehlt. Dies passiert beispielsweise deshalb, weil die Transkription der mRNA vor Erreichen des Stopcodon vorzeitig abgebrochen wurde. Auch eine Leserasterverschiebung kann dazu führen, dass im Leseraster kein Stopcodon mehr vorkommt. Alternativ kann während der Translation das in der mRNA vorkommende Stopcodon überlesen werden. In all diesen Beispielen gelangt das Ribosom schließlich an das 3'-Ende der mRNA und verharrt in diesem Wartezustand; es kann weder „zurück“ in die 5'- noch „vorwärts“ in 3'-Richtung.
Da dieser Komplex aus Ribosom und mRNA in zellulärer Umgebung stabil ist, werden diese Ribosomen dem Translationspool entzogen und verweilen nutzlos im Cytosol. Solch eine fehlerhafte Translation geschieht bei Escherichia coli pro Zellteilung bis zu 13.000 mal, durchschnittlich jedes Ribosom in der Zelle ist während eines Zellzyklus davon betroffen.
Darüber hinaus erfüllen die dabei hergestellten und möglicherweise freigesetzten Proteine und Peptide keine Aufgabe, da sie nicht ihre volle Länge erreicht haben. Außerdem können sie potentiell schädlich für die Zelle sein.
Um dem zu begegnen, erfüllt die tmRNA folgende wichtige Funktionen:
- Sie befreit feststeckende Ribosomen von der mRNA und fügt diese wieder dem Translationspool zu.
- Sie markiert die bei der Translation entstandenen unvollständigen Peptidketten für den Abbau.
Regulation der Genexpression
Neben einer Aufgabe in der Qualitätskontrolle spielt die trans-Translation auch eine wichtige Rolle in der Regulation der Genexpression mancher Substrate.[1]
In E. coli wird die trans-Translation für die Regulation von LacI genutzt. LacI ist der homotetramere Repressor des lac-Operons, der – wenn ausreichend Glucose zur Verfügung steht – an die Promotorstellen des lac-Operons bindet. Damit wird ein Ablesen der Gene lacZ, lacY und lacA verhindert, die für den Abbau der Lactose nötig wären. Der LacI-Repressor autoinhibiert seine eigene Synthese; er bindet dabei an zwei lac-Operatorstellen, O1 und O3. O1 liegt beim lacZYA-Promotor, O3 am Ende der codierenden Region für lacI selbst. Durch Binden von LacI an O1 und O3 kommt es für die dazwischenliegenden DNA zu einer Ausbildung einer Schleife. Wenn nun eine RNA-Polymerase das lacI-Gen abliest (Transkription), gelangt sie vor Erreichen des Stopcodons an diese Schleife und fällt ab. Das transkribierte LacI ist damit unvollständig, es fehlt u. a. das Stopcodon.
Ribosomen, die diese unvollständige LacI-mRNA ablesen, würden schließlich am 3’-Ende festsitzen. Nur durch den Prozess der tmRNA in der trans-Translation wird sichergestellt, dass diese Ribosomen befreit und das unvollständige LacI (LacI*) dem Abbau zugeführt wird. Letzteres ist die entscheidende Aufgabe in der Regulation durch die trans-Translation, denn es wurde gezeigt, dass LacI* immer noch ein aktiver Repressor ist; die Regulation der Genexpression würde damit ohne den Prozess der trans-Translation scheitern.[3]
Ein anderes Beispiel für die Rolle der trans-Translation wird im Zuge einer Stressantwort vorgeschlagen.[4] Unter Stressbedingungen sorgt das Toxin RelE für einen globalen, zellulären Translationsstopp, indem mRNAs geschnitten werden. Dies soll dafür sorgen, dass kostbare Ressourcen sofort für essentielle zelluläre Prozesse umgeleitet werden – die Translation ist ein kostspieliger Vorgang. Wenn die stressinduzierende Ursache überwunden wurde, wird RelE inaktiviert. Durch den Prozess der trans-Translation werden die Ribosomen befreit, so dass die Zelle wieder normal arbeiten kann.
Literatur
- K. C. Keiler: Biology of trans-translation. In: Annu Rev Microbiol., 62, 2008, S. 133–151. PMID 18557701. doi:10.1146/annurev.micro.62.081307.162948
- J. Wower, I. K. Wower, C. Zwieb: Making the jump: new insights into the mechanism of trans-translation. In: J Biol., 7(5), 2008, S. 17. PMID 18598387, PMC 2447533 (freier Volltext).
- O. V. Shpanchenko u. a.: Structural aspects of trans-translation. In: IUBMB Life, 62(2), 2010, S. 120–124. PMID 20073035. doi:10.1002/iub.296
- C. S. Hayes, K. C. Keiler: Beyond ribosome rescue: tmRNA and co-translational processes. In: FEBS Lett., 584(2), 2010, S. 413–419. PMID 19914241
Einzelnachweise
- K. C. Keiler: Biology of trans-translation. In: Annu Rev Microbiol., 62, 2008, S. 133–151. PMID 18557701. doi:10.1146/annurev.micro.62.081307.162948.
- Y. Bessho u. a.: Structural basis for functional mimicry of long-variable-arm tRNA by transfer-messenger RNA. In: Proc. Natl. Acad. Sci. USA. Band 104, Nr. 20, 2007, S. 8293–8298, doi:10.1073/pnas.0700402104, PMID 17488812.
- T. Abo u. a.: SsrA-mediated tagging and proteolysis of LacI and its role in the regulation of lac operon. In: EMBO J., 19(14), 2000, S. 3762–3769. PMID 10899129. PMC 313975 (freier Volltext).
- S. K. Christensen, K. Gerdes: RelE toxins from bacteria and Archaea cleave mRNAs on translating ribosomes, which are rescued by tmRNA. In: Mol Microbiol., 48(5), 2003, S. 1389–1400. PMID 12787364.