RNA-Virus
Als RNA-Virus (Plural RNA-Viren, synonym RNS-Virus, Ribovirus) bezeichnet man Viren, deren Erbmaterial (Genom) aus RNA (Abkürzung für englisch ribonucleic acid, „Ribonukleinsäure“) besteht. Der Begriff RNA-Viren ist keine taxonomische Sammelbezeichnung und enthält keine verwandtschaftlichen Bezüge.
Eine genaue (nicht-taxonomische) Klassifikation der RNA-Viren wird in den Baltimore-Gruppen 3 (doppelsträngiges RNA-Genom), 4 (einzelsträngiges RNA-Genom positiver Polarität) und 5 (einzelsträngiges RNA-Genom negativer Polarität) und der (noch unvollständigen) Taxonomie der Viren vorgenommen. Taxonomisch werden die RNA-Viren derzeit (Stand Januar 2022) in den Realms („Bereiche“) Riboviria (fast alle RNA-Viren inklusive der Retroviren und Pararetroviren) und Ribozyviria (Deltavirus und Verwandte), sowie einigen nicht näher klassifizierten Familien wie den Avsunviroidae und Pospiviroidae erfasst.
Riboviria
| Riboviria | ||||||
|---|---|---|---|---|---|---|
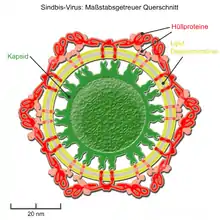
Maßstabsgerechter Querschnitt des | ||||||
| Systematik | ||||||
| ||||||
| Taxonomische Merkmale | ||||||
| ||||||
| Wissenschaftlicher Name | ||||||
| Riboviria | ||||||
| Links | ||||||
|
Riboviria ist ein vom International Committee on Taxonomy of Viruses (ICTV) 2018/2019 neu geschaffenes Taxon der höchsten Rangstufe Realm (englisch realm), analog zur Domäne bei zellulären Organismen (Lebewesen).[2] Diese Rangstufe löst immer mehr die alte Baltimore-Klassifikation ab.
Dabei umfassen die Riboviria zunächst alle echten RNA-Viren (Baltimore-Gruppen 3, 4 und 5). Aufgrund großer struktureller Unterschiede wurden jedoch Satellitenviren der Baltimore-Gruppe 5 (wie die Gattung Deltavirus mit dem Hepatitis-D-Virus) 2019/2020 herausgenommen.[3] Neu hinzugekommen sind jedoch die revers transkribierenden Viren:
Bei Retroviren (Baltimore-Gruppe 6) wird die RNA während der Replikation in der vom Virus befallenen Wirtszelle mittels eines Enzyms, der Reversen Transkriptase, in DNA umgeschrieben. Diese umfassen neben der Familie Retroviridae (Retroviren im engeren Sinn) noch einige weitere kleinere Familien wie die Belpauviridae, Metaviridae und Pseudoviridae.
Als Pararetroviren werden Viren gelegentlich bezeichnet, die ihr DNA-Genom über einen RNA-Zwischenschritt replizieren. Sie benötigen dafür ebenfalls eine Reverse Transkriptase (Baltimore-Gruppe 7). Dazu gehört die Familie Caulimoviridae, die vom ICTV wegen Homologien zusammen mit den obigen Familien von Retroviren in die gemeinsame Ordnung Ortervirales gestellt werden.
Eigenschaften
Zu den RNA-Viren gehören die meisten Pflanzenviren, viele Tierviren und einige Bakteriophagen. Die RNA-Viren können behüllt oder unbehüllt, die RNA einzelsträngig (ssRNA) oder doppelsträngig (dsRNA), positiv oder negativ strangorientiert, mit segmentiertem oder unsegmentiertem Genom vorliegen.
Die Erreger der überwiegenden Mehrheit der neu auftretenden viralen Infektionskrankheiten der letzten Jahrzehnte (Variationen der Influenzaviren, SARS, SARS-CoV-2) und Ebolavirus, aber auch die bereits jahrtausendealten Tollwut-Erreger sind RNA-Viren.
Variabilität
RNA-Viren sind aufgrund der höheren Fehlerrate der RNA-Polymerasen wesentlich variabler als DNA-Viren,[4] da ihre RNA-Polymerase meist keine proof-reading-Exonuklease-Funktion aufweist.[5][6][7] Eine Ausnahme bilden die Nidovirales, die eine Korrekturlesefunktion mit der Exoribonuklease ExoN aufweisen, wodurch die Genomgröße etwas weniger begrenzt wird.[8] Durch die hohe Mutationsrate produzieren RNA-Viren zwar mehr defekte, nicht-infektiöse virale Partikel, was aufgrund der Funktionsminderung als Fitnesskosten bezeichnet wird. Sie können sich jedoch im Zuge einer Immunevasion auch schneller an neue Wirte oder Zwischenwirte anpassen sowie durch Fluchtmutation der Immunantwort entgehen.[9] Dennoch gibt es konservierte Bereiche der viralen Genome, bei denen ein hoher Selektionsdruck auf die Funktion der konservierten Sequenz wirkt. Beispielsweise gibt es beim Hepatitis-C-Virus in der Nähe des core protein einen konservierten Bereich,[10] dessen RNA eine IRES enthält.[11] Durch die im Vergleich zu DNA-Viren geringere genetische Konservierung bzw. durch die hohe genetische Variabilität müssen Impfstoffe häufiger an aktuell kursierende Virenstämme angepasst werden.[9] Ebenso ist dadurch eine zeitliche Bestimmung der Evolution der RNA-Viren im Sinne einer molekularen Uhr schwieriger.[12][13]
Wirtsresistenz
Im Zuge der Koevolution von RNA-Viren und ihren Wirten sind in den Wirten verschiedene Mechanismen zur Abwehr der RNA-Viren entstanden. Zu den Resistenzfaktoren des Menschen gegen RNA-Viren gehören unter anderem die RNA-Interferenz, einige PAMP-Rezeptoren, die Proteinkinase R. Daneben erfolgt die Immunantwort. Jedoch haben auch RNA-Viren zusätzliche Mechanismen zur Umgehung der Resistenz entwickelt.[14]
Systematik
Die RNA-Viren werden in die Baltimore-Gruppen 3, 4 und 5 klassifiziert (die aber keine taxonomische Gruppen, d. h.Verwandtschaftsgruppen, darstellen).
Gruppe III: dsRNA-Viren
Es gibt zwölf Familien und einige nicht zugeordnete Genera:[6][15]
- Familie Amalgaviridae
- Familie Birnaviridae
- Familie Chrysoviridae
- Genus Alphachrysovirus
- Genus Betachrysovirus[15] mit Species Colletotrichum fructicola chrysovirus 1
- Familie Cystoviridae
- Familie Endornaviridae
- Familie Megabirnaviridae
- Genus Megabirnavirus
- Familie Partitiviridae
- Genus Alphapartitivirus
- Genus Betapartitivirus
- Genus Gammapartitivirus
- Genus Deltapartitivirus
- Genus Cryspovirus
- Familie Picobirnaviridae
- Genus Picobirnavirus
- Familie Quadriviridae
- Genus Quadrivirus
- Familie Reoviridae
- Familie Quadriviridae
- Genus Quadrivirus
- Familie Totiviridae
- Familie nicht bestimmt
Gruppe IV: positive-strängige ssRNA-Viren
Die Gruppe IV umfasst drei Ordnungen, über 34 Familien und einige nicht zugeordnete Virusarten und Genera.[19]
- Ordnung Nidovirales
- Unterordnung Abnidovirineae
- Familie Abyssoviridae
- Unterfamilie Tiamatvirinae
- Genus Alphaabyssovirus – Subgenus: Aplyccavirus
- Unterfamilie Tiamatvirinae
- Familie Arteriviridae
- Familie Abyssoviridae
- Unterordnung Cornidovirineae
- Familie Coronaviridae
- Unterordnung Mesnidovirineae
- Familie Medioniviridae
- Unterfamilie Medionivirinae
- Genus Turrinivirus – Subgenus: Beturrivirus
- Unterfamilie Tunicanivirinae
- Genus Bolenivirus – Subgenus: Balbicanovirus
- Unterfamilie Medionivirinae
- Familie Mesoniviridae
- Unterfamilie Hexponivirinae
- Genus Alphamesonivirus – Subgenera: Casualivirus, Enselivirus, Hanalivirus, Kadilivirus, Karsalivirus, Menolivirus, Namcalivirus, Ofalivirus
- Unterfamilie Hexponivirinae
- Familie Medioniviridae
- Unterordnung Monidovirineae
- Familie Mononiviridae
- Unterfamilie Mononivirinae
- Genus Alphamononivirus – Subgenus: Dumedivirus
- Unterfamilie Mononivirinae
- Familie Mononiviridae
- Unterordnung Ronidovirineae
- Familie Euroniviridae
- Unterfamilie Ceronivirinae
- Genus Charybnivirus – Subgenera: Cradenivirus, Wenilivirus
- Unterfamilie Crustonivirinae
- Genus Paguronivirus – Subgenus: Behecravirus
- Unterfamilie Ceronivirinae
- Familie Roniviridae
- Unterfamilie Okanivirinae
- Genus Okavirus – Subgenus: Tipravirus
- Unterfamilie Okanivirinae
- Familie Euroniviridae
- Unterordnung Tornidovirineae
- Familie Tobaniviridae
- Unterfamilie Piscanivirinae
- Unterfamilie Remotovirinae
- Genus Bostovirus – Subgenus: Bosnitovirus
- Unterfamilie Serpentovirinae
- Genus Infratovirus – Subgenus: Xintolivirus
- Genus Pregotovirus – Subgenus: Roypretovirus
- Genus Sectovirus – Subgenus: Sanematovirus
- Genus Tiruvirus – Subgenus: Tilitovirus
- Unterfamilie Torovirinae (früher Fam. Coronaviridae)
- Familie Tobaniviridae
- Unterordnung Abnidovirineae
- Ordnung Picornavirales
- Familie Dicistroviridae
- Familie Iflaviridae
- Familie Marnaviridae
- Familie Picornaviridae
- Familie Polycipiviridae
- Genus Chipolycivirus
- Genus Hupolycivirus
- Genus Sopolycivirus
- Familie Secoviridae
- Unterfamilie Comovirinae
- Genus Comovirus
- Genus Fabavirus
- Genus Nepovirus, mit Species Chicory yellow mottle virus, Grapevine bulgarian latent virus, Grapevine fanleaf virus, Myrobalan latent ringspot virus, Tomatenschwarzringvirus – en. Tomato black ring virus,; Arabis-Mosaic-Virus – en. Arabis mosaic virus, sowie Beet ringspot virus
- Unterfamilie nicht bestiimt
- Genus Cheravirus
- Genus Sadwavirus
- Genus Sequivirus
- Genus Torradovirus
- Genus Waikavirus
- Unterfamilie Comovirinae
- Ordnung Tolivirales
- Familie Carmotetraviridae
- Genus Alphacarmotetravirus, mit Species Providence virus
- Familie Luteoviridae
- Familie Tombusviridae
- Familie Carmotetraviridae
- Ordnung Tymovirales
- Familie Alphaflexiviridae
- Genus Allexivirus
- Genus Botrexvirus
- Genus Lolavirus
- Genus Mandarivirus
- Genus Platypuvirus
- Genus Potexvirus
- Genus Sclerodarnavirus
- Familie Betaflexiviridae
- Unterfamilie Quinvirinae
- Genus Carlavirus
- Genus Foveavirus
- Genus Robigovirus
- Unterfamilie Trivirinae
- Genus Capillovirus
- Genus Chordovirus
- Genus Citrivirus
- Genus Divavirus
- Genus Prunevirus
- Genus Tepovirus
- Genus Trichovirus
- Genus Vitivirus
- Unterfamilie Quinvirinae
- Familie Gammaflexiviridae
- Genus Mycoflexivirus
- Familie Deltaflexiviridae
- Genus Deltaflexivirus
- Familie Tymoviridae
- Genus Maculavirus
- Genus Marafivirus
- Genus Tymovirus, mit Species Wild cucumber mosaic virus sowie Gelbe-Rüben-Mosaikvirus – en. Turnip yellow mosaic virus (TYMV)
- Familie Alphaflexiviridae
- Ordnung nicht bestimmt:
- Familie Alphatetraviridae
- Genus Betatetravirus
- Genus Omegatetravirus
- Familie Alvernaviridae
- Genus Dinornavirus
- Familie Alvernaviridae
- Genus Amalgavirus
- Genus Zybavirus
- Familie Astroviridae
- Genus Avastrovirus[20]
- Genus Mamastrovirus[21]
- Genus ‚Bastrovirus‘[22]
- Familie Barnaviridae
- Genus Barnavirus[23]
- Familie Benyviridae
- Genus Benyvirus, mit Species Beet necrotic yellow vein virus (satelliten-ähnliche RNA)
- Familie Botourmiaviridae (alias Ourmiaviridae[24])
- Genus Botoulivirus
- Genus Magoulivirus
- Genus Ourmiavirus
- Genus Scleroulivirus
- Familie Bromoviridae
- Familie Caliciviridae
- Familie Carmotetraviridae
- Genus Alphacarmotetravirus
- Familie Closteroviridae
- Familie Flaviviridae
- Familie „Fusariviridae“[25][26][27]
- Familie Hepeviridae
- Familie Hypoviridae
- Genus Hypovirus
- Familie Kitaviridae[28]
- Genus Blunervirus, mit Species Blueberry necrotic ring blotch virus (BNRBV)
- Genus Cilevirus (CiLV)
- Genus Higrevirus, mit Species Hibiscus green spot virus 2 (HGSV-2)
- Familie Leviviridae
- Familie Luteoviridae
- Genus Enamovirus
- Genus Luteovirus, mit den Barley-Yellow-Dwarf-Viren – en. Barley yellow dwarf virus MAV (BYDV-MAV), Barley yellow dwarf virus PAV (BYDV-PAV) sowie Barley yellow dwarf virus PAS (BYDV-PAS)
- Genus Polerovirus, mit Species Cereal yellow dwarf virus RPV (CYDV-RPV) sowie Cereal yellow dwarf virus RPS (CYDV-RPS)
- Familie Matonaviridae
- Genus Rubivirus (früher zu Togaviridae), mit einziger Species Rötelnvirus, en. Rubella virus (RUBV)
- Familie Narnaviridae
- Genus Mitovirus
- Genus Narnavirus
- Familie Nodaviridae
- Familie Permutotetraviridae
- Genus Alphapermutotetravirus
- Familie Potyviridae
- Familie Sarthroviridae (laut Vorschlag sind dies Satelliten-Viren)
- Genus Macronovirus, mit einziger Species Macrobrachium satellite virus 1[29], zu dieser Extra small virus (XSV)[30][31]
- Familie Solemoviridae
- Genus Polemovirus
- Genus Sobemovirus, mit Species Cymbidium ringspot virus
- Familie Solinviviridae
- Genus Invictavirus, mit Species Solenopsis invicta virus 3
- Genus Nyfulvavirus, mit Species Solenopsis invicta virus 3
- Familie Togaviridae
- Familie Tombusviridae
- Familie Virgaviridae[32]
- Familie nicht bestimmt:
- Genus Jingmenvirus mit Species Jingmen tick virus (JMTV,?Fam. Flaviviridae)[33]
- Genus Ideaovirus, mit Species Privet idaeovirus
- Genus Sinaivirus mit Species Lake Sinai Virus 1 und 2[34]
- Genus ‚Negevirus‘, mit Species ‚Blackford virus‘, ‚Bofa virus‘, ‚Buckhurst virus‘, ‚Marsac virus‘, ‚sowie Muthill virus‘[35]
- Familie Alphatetraviridae
- Nicht zugeordnete Gruppe:[36][37][38]
- ‚Orsay virus‘[39]
- ‚Le Blanc virus‘
- ‚Santeuil virus‘
- Genus nicht bestimmt und nicht in ICTV Master Species List (Ausgabe #34)
Satellitenviren
Klassifizierung nach Krupovic et al. (2016).[47]
- Familie Sarthroviridae,[48] mit Genus Macronovirus[49]
- Familie nicht bestimmt
- Genus Albetovirus
- Genus Aumaivirus
- Genus Papanivirus
- Genus Virtovirus
Gruppe V: negativ-strängige ssRNA-Viren
In der Gruppe V befinden sich 8 Ordnungen und mehr als 21 Familien.[50] Daneben existieren einige nicht zugeordnete Familien, Genera und Virusarten.[6] Seit November 2018 hat das ICTV diese Viren (mit Ausnahme des Deltavirus) verschiedenen Phyla, Subphyla und Klassen zugeordnet.[51]
- Phylum (Stamm): Negarnaviricota, Subphylum (Unterstamm): Haploviricotina, Klasse: Chunqiuviricetes
- Phylum (Stamm): Negarnaviricota, Subphylum (Unterstamm): Haploviricotina, Klasse: Milneviricetes
- Ordnung Serpentovirales
- Familie Aspiviridae (veraltet Ophioviridae)
- Genus Ophiovirus
- Familie Aspiviridae (veraltet Ophioviridae)
- Ordnung Serpentovirales
- Phylum (Stamm): Negarnaviricota, Subphylum (Unterstamm): Haploviricotina, Klasse: Monjiviricetes
- Ordnung Jingchuvirales
- Familie Chuviridae
- Genus Mivirus, mit Species Crustacean Mivirus
- Familie Chuviridae
- Ordnung Mononegavirales (nicht segmentierte negativsträngige RNA-Viren)[52]
- Familie Artoviridae
- Genus Peropuvirus (früher zu Nyamiviridae), mit Species Pillworm peropuvirus
- Familie Bornaviridae
- Genus Carbovirus, mit Species Southwest carbovirus (Southwest carpet python virus, SWCPV) und Typusspecies Queensland Carbovirus (Jungle carpet python virus, JCPV)
- Genus Cultervirus, mit Typusspecies Sharpbelly cultervirus
- Genus Orthobornavirus[53] – en. Borna Disease Virus, das Virus der Bornaschen Krankheit, mit Species Mammalian 1 orthobornavirus (Typus) u. a.
- Familie Filoviridae
- Familie Lispiviridae
- Genus Genus Arlivirus (inklusive der früheren Gattungen Wastrivirus und Chengivirus/Chengtivirus, mit Species Wuchang arlivirus (Chengtivirus, Tick virus 6, TcTV-6), Tacheng arlivirus (Tacheng chengtivirus 6, TcTV-6) sowie Typusspecies Lishi arlivirus (Lishi spider virus 2, LsSV-2)[54]
- Familie Mymonaviridae
- Genus Sclerotimonavirus mit Typusspecies Sclerotinia sclerotimonavirus (Sclerotinia sclerotiorum negative-stranded RNA virus 1, SsNSRV-1)
- Familie Nyamiviridae
- Genus Berhavirus, mit Species Echinoderm berhavirus
- Genus Crustavirus (ehemals Crabvirus), mit Species Wenzhou crustavirus (WzCV-1)
- Genus Nyavirus, mit Species Sierra nevada nyavirus
- Genus Orinovirus
- Genus Socyvirus
- Genus Tapwovirus
- Familie Paramyxoviridae
- Familie Pneumoviridae
- Familie Rhabdoviridae
- Familie Sunviridae
- Genus Sunshinevirus (SunCV)
- Familie Xinmoviridae
- Genus Anphevirus (XcMV)
- keiner Familie zugeordnet
- Familie Artoviridae
- Ordnung Jingchuvirales
- Phylum (Stamm): Negarnaviricota, Subphylum (Unterstamm): Haploviricotina, Klasse: Yunchangviricetes
- Ordnung Goujianvirales
- Familie Yueviridae
- Genus Yuyuevirus
- Familie Yueviridae
- Ordnung Goujianvirales
- Phylum (Stamm): Negarnaviricota, Subphylum (Unterstamm): Haploviricotina, Klasse: Ellioviricetes
- Ordnung Bunyavirales
- Familie Arenaviridae
- Familie Cruliviridae
- Genus Lincruvirus
- Familie Fimoviridae
- Genus Emaravirus, mit Species European mountain ash ringspot-associated virus[56]
- Familie Hantaviridae
- Familie Leishbuviridae
- Familie Mypoviridae
- Genus Hubavirus
- Familie Nairoviridae
- Genus Orthonairovirus
- Genus Shaspivirus
- Genus Striwavirus
- Familie Peribunyaviridae
- Familie Phasmaviridae
- Genus Orthophasmavirus
- Genus Feravirus (früher zu Feraviridae)
- Genus Inshuvirus
- Genus Jonvirus (früher als Orthojonvirus zu Jonviridae)
- Genus Sawastrivirus (früher Wastrivirus), mit Typusspecies Sanxia sawastrivirus (Sanxia wastrivirus Water strider virus 4, SxWSV-4)[54]
- Genus Wuhivirus
- Familie Phenuiviridae
- Genus Bandavirus (früher Banyangvirus), mit Species Dabie bandavirus – syn. SFTS-Virus
- Genus Beidivirus
- Genus Goukovirus
- Genus Horwuvirus
- Genus Hudivirus
- Genus Mobuvirus
- Genus Phasivirus
- Genus Phlebovirus
- Genus Pidchovirus
- Genus Tenuivirus
- Genus Wubeivirus
- Familie Tospoviridae
- Familie Wupedeviridae
- Genus Wumivirus
- Familie „Lincruviridae“ (Vorschlag)[57]
- Genus „Portunivirus“ (Vorschlag) mit Typusspezies Crab portunivirus[57]
- Ordnung Bunyavirales
- Phylum (Stamm): Negarnaviricota, Subphylum (Unterstamm): Polyploviricotina, Klasse: Insthoviricetes
- Ordnung Articulavirales
- Familie Amnoonviridae
- Genus Tilapinevirus
- Familie Orthomyxoviridae
- Familie Amnoonviridae
- Ordnung Articulavirales
- Phylum (Stamm): nicht bestimmt, Subphylum (Unterstamm): nicht bestimmt, Klasse: nicht bestimmt
- Ordnung nicht bestimmt
- Familie nicht bestimmt
- Genus Deltavirus, mit einziger Species Hepatitis-D-Virus (HDV)
- Familie nicht bestimmt
- Ordnung nicht bestimmt
Literatur
- D. M. Knipe, Peter M. Howley, D. E. Griffin, (Hrsg.): Fields Virology. 5. Auflage, Lippincott Williams & Wilkins, Philadelphia 2007, ISBN 978-0-7817-6060-7.
Einzelnachweise
- ICTV Master Species List 2018b.v2. MSL #34, März 2019
- ICTV Master Species List 2018b v1. MSL #34, Feb. 2019
- ICTV: ICTV Taxonomy history: Hepatitis delta virus. EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- R. Sanjuan, M. R. Nebot, N. Chirico, L. M. Mansky, R. Belshaw: Viral Mutation Rates. In: Journal of Virology. Band 84, Nr. 19, 2010, ISSN 0022-538X, S. 9733–9748. doi:10.1128/JVI.00694-10.
- J. W. Drake, J. J. Holland: Mutation rates among RNA viruses. In: Proceedings of the National Academy of Sciences of the United States of America. 1999, Band 96, Nr. 24, S. 13910–13913, PMID 10570172, PMC 24164 (freier Volltext).
- Donald W. Klein, Lansing M. Prescott, John Harley: Microbiology. Wm. C. Brown, Dubuque, Iowa 1993, ISBN 0-697-01372-3.
- MA Martinez et al.: Quasispecies Dynamics of RNA Viruses. In: G. Witzany (Hrsg.): Viruses: Essential Agents of Life. Springer, 2012, ISBN 978-94-007-4898-9, S. 21–42.
- C Lauber, JJ Goeman, C Parquet Mdel, P Thi Nga, EJ Snijder, K Morita, AE Gorbalenya: The footprint of genome architecture in the largest genome expansion in RNA viruses. In: PLOS Pathogens. Band 9, Nr. 7, Jul 2013, S. e1003500. doi:10.1371/journal.ppat.1003500.
- D. A. Steinhauer, J. J. Holland: Rapid evolution of RNA viruses. In: Annual Review of Microbiology. 1987, Band 41, S. 409–33, PMID 3318675.
- J. Bukh, R. H. Purcell, R. H. Miller: Sequence analysis of the core gene of 14 hepatitis C virus genotypes. In: Proceedings of the National Academy of Sciences. Band 91, Nummer 17, August 1994, S. 8239–8243, PMID 8058787, PMC 44581 (freier Volltext).
- A. Tuplin, D. J. Evans, P. Simmonds: Detailed mapping of RNA secondary structures in core and NS5B-encoding region sequences of hepatitis C virus by RNase cleavage and novel bioinformatic prediction methods. In: The Journal of general virology. Band 85, Nr. 10, Oktober 2004, S. 3037–3047, doi:10.1099/vir.0.80141-0, PMID 15448367.
- E. C. Holmes: What does virus evolution tell us about virus origins? In: Journal of Virology. 2011, Band 85, Nr. 11, S. 5247–51. doi:10.1128/JVI.02203-10, PMID 21450811, PMC 3094976 (freier Volltext).
- M. R. Patel, M. Emerman, H. S. Malik: Paleovirology - ghosts and gifts of viruses past. In: Current Opinion in Virology. 2011, Band 1, Nr. 4, S. 304–309, doi:10.1016/j.coviro.2011.06.007, PMID 22003379, PMC 3190193 (freier Volltext).
- A. M. Dickson, J. Wilusz: Strategies for viral RNA stability: live long and prosper. In: Trends in Genetics. 2011, Band 27, Nr. 7, S. 286–93. doi:10.1016/j.tig.2011.04.003. PMID 21640425, PMC 3123725 (freier Volltext).
- Circulifer tenellus virus 1. Auf: Virus-Host DB.
- Henxia Xia et al.: A dsRNA virus with filamentous viral particled. In: Nature Communications. Band 8, Nr. 168, 2017, doi:10.1038/s41467-017-00237-9
- Spissistilus festinus virus 1. Auf: Virus-Host DB.
- SIB: Positive Strand RNA Viruses. Auf: ViralZone.
- SIB: Avastrovirus. Auf: ViralZone.
- SIB: Mamastrovirus. Auf: ViralZone.
- Karoline dos Anjos, Tatsuya Nagata, Fernando Lucas de Melo: Complete Genome Sequence of a Novel Bastrovirus Isolated from Raw Sewage. In: Genome Announcements. Oktober 2017, Band 5, Nr. 40, S. e01010-17, doi:10.1128/genomeA.01010-17, PMC 5629051 (freier Volltext).
- SIB: Barnavirus. Auf: ViralZone.
- Eugene V. Koonin, Valerian V. Dolja: Virus World as an Evolutionary Network of Viruses and Capsidless Selfish Elements. In: Microbiology and Molecular Biology Reviews. 20. Mai 2014, doi:10.1128/MMBR.00049-13, Figur 3.
- Fusariviridae (FAMILY). Auf: UniProt Taxonom.
- FUSARIVIRIDAE, auf: PLANOSPHERE
- Fusariviridae, auf: NCBI Genomes
- D. F. Quito-Avila, P. M. Brannen, W. O. Cline, P. F. Harmon, R. R. Martin: Genetic characterization of Blueberry necrotic ring blotch virus, a novel RNA virus with unique genetic features. In: Journal of General Virology. Juni 2013, Band 94, Teil 6, S. 1426–1434, doi:10.1099/vir.0.050393-0, PMID 23486668
- SIB: Macronovirus. Auf: ViralZone.
- D. Qian et al.: Extra small virus-like particles (XSV) and nodavirus associated with whitish muscle disease in the giant freshwater prawn, Macrobrachium rosenbergii. In: Journal of Fish Diseases. Band 26, Nr. 9, September 2003, S. 521-527, PMID 14575370, doi:10.1046/j.1365-2761.2003.00486.x.
- J.Widada, S. Widada, J. R. Bonami: Characteristics of the monocistronic genome of extra small virus, a virus-like particle associated with Macrobrachium rosenbergii nodavirus: possible candidate for a new species of satellite virus. In: Journal of General Virology. Band 85, Nr. 3, März 2004, S. 643–646, PMID 14993649, doi:10.1099/vir.0.79777-0
- M. J. Adams, J. F. Antoniw, J. Kreuze: Virgaviridae: a new Familie of rod-shaped plant viruses. In: Archives of Virology.. Band 154, Nr. 12, 2009, S. 1967–72. doi:10.1007/s00705-009-0506-6. PMID 19862474.
- Xin-Cheng Qin et al.: A tick-borne segmented RNA virus contains genome segments derived from unsegmented viral ancestors. In: PNAS Band 111, Nr. 18, 6. Mai 2014, S. 6744–6749, doi:10.1073/pnas.1324194111
- SIB: Double Stranded RNA Viruses. Auf: ViralZone.
- Claire L. Webster, Ben Longdon, Samuel H. Lewis, Darren J. Obbard: Twenty-Five New Viruses Associated with the Drosophilidae (Diptera). In: Evolutionary bioinformatics online. 2016, Band 12, Supplement 2, S. 13–25, doi:10.4137/EBO.S39454, PMC PMC4915790 (freier Volltext), PMID 27375356
- Carl J. Franz, Guoyan Zhao, Marie-Anne Félix, David Wang: Complete Genome Sequence of Le Blanc Virus, a Third Caenorhabditis Nematode-Infecting Virus. In: Journal of Virology. 2012, doi:10.1128/JVI.02025-12, PMID 23043172, American Society for Microbiology Journals
- Hongbing Jiang, Carl J. Franz, Guang Wu, Hilary Renshaw, Guoyan Zhao, Andrew E. Firth, David Wang: Orsay virus utilizes ribosomal frameshifting to express a novel protein that is incorporated into virions. In: Virology. 2 Februar 2014, Band 450-451, Nr. 100, S. 213–221, doi:10.1016/j.virol.2013.12.016, PMC 3969245 (freier Volltext), PMID 24503084.
- Carl J. Franz, Hilary Renshaw, Lise Frezal, Yanfang Jiang, Marie-Anne Félix, David Wang: Orsay, Santeuil and Le Blanc viruses primarily infect intestinal cells in Caenorhabditis nematodes. In: Virology. Band 448, 5 Januar 2014, S. 255–264, doi:10.1016/j.virol.2013.09.024.
- Orsay virus. Auf: Virus-Host DB.
- J. F. van den Heuvel, H. R. Hummelen, Martin Verbeek, Annette Dullemans: Characteristics of Acyrthosiphon pisum Virus, a Newly Identified Virus Infecting the Pea Aphid. In: Journal of Invertebrate Pathology. November/ Dezember 1997, Band 70, Nr. 3, S. 169–176, doi:10.1006/jipa.1997.4691, PMID 9367722
- A. J. Gibbs, M. Torronen, A. M. Mackenzie, J. T. Wood, J. S. Armstrong, H. Kondo, T. Tamada, P. L. Keese: The enigmatic genome of Chara australis virus. In: Journal of General Virology. November 2011, Band 92, Teil 11, S. 2679–2690, doi:10.1099/vir.0.033852-0, PMID 21733884.
- Nesidiocoris tenuis virus. Auf: Virus-Host DB.
- Marion Heller-Dohmen et al.: The nucleotide sequence and genome organization of Plasmopara halstedii virus. In: Virology Journal. 2011, Band 8, S. 123, doi:10.1186/1743-422X-8-123, PMC 3069955 (freier Volltext), PMID 21410989.
- Rosellinia necatrix fusarivirus 1. Auf: Virus-Host DB.
- Kelp fly virus. Auf: Virus-Host DB.
- Eric Dubois et al.: Effect of pollen traps on the relapse of chronic bee paralysis virus in honeybee (Apis mellifera) colonies. In: Apidologie. April 2018, Band 49, Nr. 2, S. 235–242, doi:10.1007/s13592-017-0547-x.
- M. Krupovic, J. H. Kuhn, M. G. Fischer: A classification system for virophages and satellite viruses. In: Archives of Virology. Band 161, Nr. 1, 2016, S. 233–247, doi:10.1007/s00705-015-2622-9.
- ICTV: Sarthroviridae. Virus Taxonomy: 2019 Release EC 51, Berlin, Germany, July 2019 (MSL #35).
- SIB: Macronovirus. Auf: ExPASy: ViralZone.
- Alan Cann: Principles of Molecular Virology. Academic Press, 2011, ISBN 978-0-12-384939-7.
- SIB: Negative Strand RNA Viruses. Auf: ViralZone.
- Gaya K. Amarasinghe et al.: Taxonomy of the order Mononegavirales: update 2018. In: Archives of Virology. Band 163, Nr. 8, August 2018, S. 2283–2294.
- SIB: Orthobornavirus. Auf: ViralZone.
- Claudio L. Afonso et al.: Taxonomy of the order Mononegavirales: update 2016. In: Archives of Virology. Band 161, Nr. 8, 1. August 2016, S. 2351–2360.
- J. O. Bool et al.: Identification and partial characterization of Taastrup virus: a newly identified member species of the Mononegavirales. In: Virology. 5. Februar 2004, Band 319, Nr. 1, S. 49–59, PMID 14967487, doi:10.1016/j.virol.2003.10.017.
- ICTV Emaravirus. Auf: talk.ictvonline.org
- Jamie Bojko: Animal dsRNA and ssRNA- viruses. Vorschlag an das ICTV vom 15. Oktober 2019.