Hepatitis-D-Virus
Hepatitis-D-Virus (offiziell Deltavirus italiense[1]; engl. früher auch Hepatitis delta virus, HDV, früher auch δ-Agens genannt) ist eine Spezies (Art) in der Gattung Deltavirus. Es ist ein nur beim Menschen natürlich vorkommendes Virusoid, also ein von den Genprodukten eines anderen Virus (dem Hepatitis-B-Virus, HBV) abhängiges Virus. Es kann in einer Zelle nur dann neue infektiöse Partikel bilden, wenn diese Zelle gleichzeitig mit HBV infiziert ist und die Hüllproteine (HBs-Antigene) des HBV produziert werden. Das Hepatitis-D-Virus ist der Erreger einer chronischen Leberentzündung, der Hepatitis D, die gleichzeitig mit einer frischen Hepatitis B auftreten kann (Simultaninfektion) oder bei einer chronischen Hepatitis B als zusätzliche Infektion hinzukommt (Superinfektion). Sein in der Tierwelt einzigartiges RNA-Genom zeigt eine strukturelle und funktionelle Verwandtschaft mit einigen viralen Erregern bei Pflanzen. Aufgrund seiner besonderen Genomstruktur und Replikationsweise gilt das HDV als molekulares Relikt der chemischen Evolution und stützt die Annahmen der RNA-Welt-Hypothese. Die frühere Zuordnung zum Realm Riboviria wurde vom International Committee on Taxonomy of Viruses (ICTV) im März 2020 zurückgezogen,[2] ein neuer Realm Ribozyviria[3] wurde fůr die Deltaviren und ähnliche Viren errichtet.[1]
| Hepatitis-D-Virus | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Deltavirus italiense[1] | ||||||||||||||
| Kurzbezeichnung | ||||||||||||||
| HDV | ||||||||||||||
| Links | ||||||||||||||
|
Entdeckung
Der Turiner Gastroenterologe Mario Rizzetto und seine Arbeitsgruppe berichteten 1977 über den Nachweis eines bis dahin unbekannten Proteins in Leberbioptaten einiger Patienten, die während eines größeren Ausbruchs von HBV im Mittelmeerraum in der Mitte der 1970er-Jahre chronisch mit dem Hepatitis-B-Virus (HBV) infiziert worden waren.[4] Rizzetto hatte sich vorher mit dem Nachweis von Proteinen des HBV durch Immunfluoreszenz in Zellkernen infizierter Leberzellen beschäftigt. Das neue Protein hielt er zunächst für ein bislang nicht erkanntes Antigen des HBV, das er ebenfalls in den Zellkernen infizierter Leberzellen nachweisen konnte. Da es eng mit dem bereits bekannten Core-Antigen des HBV (HBc-Antigen) assoziiert war, nannte er das neue Protein „Delta-Antigen“. Patienten mit dem Delta-Antigen entwickelten auch spezifische Antikörper gegen dieses Protein.
Das Delta-Antigen konnte zwar in HBV-infizierten Zellen nachgewiesen werden, jedoch nicht in gereinigten Virionen des HBV. Somit war es kein Strukturprotein des HBV und fand sich nur während der Virusvermehrung.[5] In Zusammenarbeit mit Robert H. Purcell und John L. Gerin wurden Schimpansen mit Delta-Antigen-haltigen Patientenproben infiziert. Experimente mit Schimpansen waren bei der Erforschung von HBV-Infektionen seit den frühen 70er-Jahren durchgeführt worden. Im Blutserum eines infizierten Tieres konnten Purcell, Gerin und Rizzetto schließlich Viruspartikel nachweisen, die Delta-Antigen enthielten, jedoch deutlich kleiner waren als HBV-Virionen (35 bis 37 nm im Vergleich zu etwa 50 nm). Die Partikel waren mit einem RNA-Molekül assoziiert, das sich von den viralen mRNA-Molekülen des HBV unterschied und für alle bis dahin bekannten, bei tierischen Viren vorkommenden RNA-Genome sehr klein war (molekulare Masse nur 5 × 105). Damit war der Nachweis erbracht, dass es sich um ein neu entdecktes Virus handelte und das Delta-Antigen ein Bestandteil dieses neuen Virus war.[6][7]
Auf der Basis von gereinigtem Delta-Antigen wurden 1981 ein Radioimmunassay und ein ELISA-Test entwickelt, die spezifische anti-HDV-Antikörper im Blutserum von Patienten erkannten.[8] Die Antikörper wurden weltweit bei sehr vielen Patienten gefunden, die mit HBV infiziert waren; besonders häufig gelang der Nachweis bei HBV-Patienten aus dem Mittelmeerraum, Hämophilen und Suchtkranken von intravenösen Drogen. Bei Blutproben von anti-HBc-negativen Patienten, also Personen, die niemals mit HBV infiziert waren oder sind, wurden die anti-HDV-Antikörper nicht gefunden. Dies ließ den Schluss zu, dass HDV nur assoziiert mit HBV vorkommt und es ähnlich wie HBV parenteral durch Blut übertragen wird.
Die RNA des HDV wurde von A. Kos am Primatenzentrum in Rijswijk (Niederlande) 1986 genauer charakterisiert und als einzelsträngiger, zirkulär geschlossener Ring erkannt.[9] Die erste kurze Sequenzierung der RNA zeigte die hohe Ähnlichkeit des HDV zu Viroiden, den bei Pflanzen vorkommenden, infektiösen RNA-Molekülen. Der hydrodynamische Durchmesser der HDV-Virionen wurde 1986 von Ferruccio Bonino und Wolfram H. Gerlich mittels Gelfiltration auf 36 nm bestimmt; sie wiesen auch erstmals die Existenz zweier verschieden großen Formen des Delta-Antigen nach und dass die Virushülle des HDV fast ausschließlich aus dem kleinsten der drei HBV-Hüllproteine des HBV (sHBs-Antigen) besteht.[10]
Verbreitung und Wirtsspektrum
Das HDV ist weltweit verbreitet, eine besonders hohe Prävalenz liegt in Süditalien und dem gesamten Mittelmeerraum vor. Regional hohe Prävalenzen findet man ebenso in Rumänien, der Mongolei und einigen Ländern Zentralafrikas und Südamerikas (Venezuela, Kolumbien, östliches Brasilien). Die Prävalenz von HDV verhält sich überwiegend gleichförmig zur Häufigkeit chronischer HBV-Infektionen. Abweichend hiervon ist die HDV-Prävalenz in China bei etwa 9 % chronisch HBV-Infizierter vergleichsweise gering. In entwickelten Ländern ist die Prävalenz von HDV in der Gesamtpopulation gering, jedoch erhöht bei Personengruppen, die einem hohen Risiko einer parenteralen Übertragung ausgesetzt sind, darunter vor allem i.v.-Drogenabhängige. Bei HDV können aufgrund der Genomsequenz drei Genotypen unterschieden werden, wobei der Genotyp 1 weltweit nachgewiesen wurde, Genotyp 2 ist in Asien (v. a. Taiwan und Japan) dominant und Genotyp 3 wurde ursprünglich nur in Südamerika nachgewiesen.
Ein anderer natürlicher Wirt als der Mensch wurde für das HDV bislang nicht beschrieben; einziger Ort der Virusreplikation sind Leberzellen. Unter experimentellen Bedingungen gelang die Infektion von Schimpansen bei Anwesenheit von HBV und bei Waldmurmeltieren (Woodchucks), wenn diese mit dem HBV-ähnlichen Woodchuck-Hepatitis-Virus (WHV), Gattung Orthohepadnavirus, infiziert waren. In letzterem Fall entstanden HDV-Virionen, in deren Virushülle das WHV-S-Antigen vorlag. Stabil HDV-infizierte Zellkulturen konnten bisher nicht etabliert werden. Nach Einschleusung (Transfektion) experimentell hergestellter, komplementärer HDV-RNA (HDV-cDNA) in Zellkulturen ist sich replizierende HDV-RNA und Delta-Antigen nachweisbar. Da diese Replikation in sehr verschiedenen Zelllinien durchgeführt werden kann, ist anzunehmen, dass der natürliche strenge Tropismus für Leberzellen auf spezifische Rezeptoren der Zelloberfläche zurückzuführen ist. Solange in den transfizierten Zellen nicht gleichzeitig auch HBs-Antigen synthetisiert wird, kommt es jedoch nicht zu einer Verpackung der HDV-Kapside und damit nicht zur Produktion infektiöser HDV-Virionen.
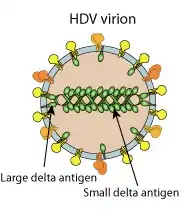
Virusstruktur
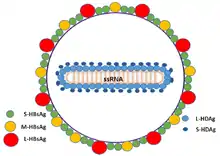
Virusgenom
Das Genom des HDV besteht aus einer 1.670 bis 1.683 Nukleotiden großen, einzelsträngigen RNA mit negativer Polarität (-ssRNA). Der GC-Gehalt ist mit etwa 60 % vergleichsweise hoch. Das HDV ist das einzige humanpathogene Virus, dessen genomische RNA zu einem Ring geschlossen (zirkulär) vorliegt. Dieser wird nur unter denaturierenden Bedingungen im Elektronenmikroskop sichtbar. Unter nicht-denaturierenden, physiologischen Bedingungen kommt es innerhalb des RNA-Stranges in einem Bereich von etwa 70 % zur Ausbildung von intramolekularen Basenpaarungen und die RNA erscheint dann als unverzweigter, stäbchenförmiger Strang. Das HDV-Genom codiert nur für ein einzelnes Protein, das HDV-Antigen, dessen Offener Leserahmen (ORF) auf der komplementären, positivsträngigen RNA liegt. Beide während der RNA-Replikation entstehenden RNA-Stränge (genomische -ssRNA und antigenomische +ssRNA) besitzen eine Ribozym-Aktivität.
Morphologie
Die 36 nm im Durchmesser großen, behüllten Virionen besitzen im Inneren ein 18 nm großes, sphärisches Nukleokapsid, das aus der genomischen RNA und dem sie umgebenden HDV-Antigen gebildet wird. Die Dichte der Virionen ist bei der Dichtegradientenzentrifugation mit Cäsiumchlorid 1,25 g/ml und damit der Dichte anderer behüllter Viren vergleichbar. HDV besitzt eine im Vergleich zu anderen behüllten Viren recht hohe Umweltstabilität und kann auch bei Temperaturen bis 60 °C über 30 Stunden nicht inaktiviert werden.
Klassifikation und Bedeutung in der Virusevolution
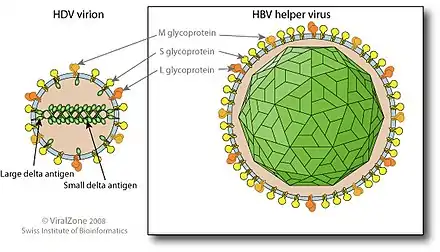
Eine Klassifikation des Hepatitis-D-Virus (Deltavirus italiense) innerhalb der Virustaxonomie stößt auf die Schwierigkeit, dass es zu anderen Viren keine Ähnlichkeit hinsichtlich der Genomsequenz und dem Replikationsmechanismus besitzt. Es ist derzeit eine der acht Spezies in der Gattung Deltavirus, zugeordnet zu der Virusfamilie Kolmioviridae. Eine Verwandtschaft besteht auf der Ebene der Genomsequenz eines Viroid-ähnlichen Abschnittes des HDV nur mit einigen Satellitenviren von Pflanzenviren, die ebenfalls als einzelsträngige, zirkuläre RNA vorkommen. Im Gegensatz zu HDV besitzen diese jedoch keine Hülle. Zu diesen nahe verwandten Viroid-Satelliten gehören das Velvet-Tobacco-Mottle-Virus (vVTMoV), das Subterranean-Clover-Mottle-Virus (vSCMoV), das Lucerne-Transient-Streak-Virus (vLTSV) und das Solanum-nodiflorum-Mottle-Virus (vSNMV). Ein gemeinsamer, evolutionärer Vorgänger des HDV und der pflanzlichen Viroide kann damit angenommen werden.[11]
In ICTV Taxonomy 2020 wurde ein neuer Bereich Ribozyviria[3] fůr die Gattung Deltavirus und ähnliche Gattungen (Daazvirus, Dagazvirus, Daletvirus, Dalvirus, Deevirus, Dobrovirus, Thurisazvirus) der Virusfamilie Kolmioviridae errichtet.[1]
Die genannten Viroid-Satelliten und das HDV werden aufgrund ihres besonderen RNA-Genoms, das sowohl genotypische als auch phänotypische Eigenschaften besitzt, als archaisches Relikt aus den frühesten Stufen einer zellfreien, molekularen Evolution angesehen. Als mögliche „Lebende Fossilien“ kann sich ihre RNA ohne Proteine (RNA-Polymerasen) oder DNA-Zwischenstufen vermehren, da das RNA-Genom die Fähigkeiten zur autonomen Selbstvermehrung, zur enzymatischen Spaltung von sich selbst (Autolyse durch Ribozym-Aktivität) und zum Aneinanderheften eigener RNA-Fragmente (RNA-Ligation) besitzt.[12] Damit stünden diese viralen Systeme am Übergang der chemischen zur biologischen Evolution und repräsentieren Eigenschaften, die sich aus der Hypothese des Hyperzyklus und der Entstehung der evolutionären Quasispezies ableiten lassen. Diese archaischen viralen RNA-Genome spiegeln möglicherweise eine nur aus RNA bestehende Vorstufe in der Entstehung des Lebens wider, die in der sogenannten RNA-Welt-Hypothese angenommen wird.
Meldepflicht
In Deutschland ist jeder direkte oder indirekte Nachweis vom Hepatitis-D-Virus namentlich meldepflichtig nach § 7 des Infektionsschutzgesetzes.
Literatur
- Michael M. C. Lai: Hepatitis Delta Virus. In: Allan Granoff, Robert G. Webster: Encyclopedia of Virology. San Diego 1999, (Band 1) S. 664–669, ISBN 0-12-227030-4.
- David M. Knipe, Peter M. Howley (Hrsg.): Fields’ Virology. 5. Auflage, 2 Bände. Philadelphia 2007, ISBN 0-7817-6060-7.
- A. Smedile und M. Rizzetto: HDV: thirty years later. Dig. Liver Dis. (2011) 43 Suppl 1, S. 15–18, PMID 21195366.
Einzelnachweise
- ICTV: ICTV Taxonomy history: Deltavirus italiense, EC 52, Online meeting, October 2020; Email ratification March 2021 (MSL #36)
- ICTV: ICTV Taxonomy history: Hepatitis delta virus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- Ribozyviria Proposal, akzeptiert von ICTV EC 52 Online meeting, Oktober 2020, und ratifiziert im März 2021
- M. Rizzetto, M. G. Canese et al.: Immunofluorescence detection of new antigen-antibody system (delta/anti-delta) associated to hepatitis B virus in liver and in serum of HBsAg carriers. Gut (1977) 18(12), S. 997–1003, PMID 75123, PMC 1411847 (freier Volltext)
- M. G. Canese, M. Rizzetto et al.: An ultrastructural and immunohistochemical study on the delta antigen associated with the hepatitis B virus. J. Pathol. (1979) 128(4), S. 169–175, PMID 392063
- Rizzetto M, Hoyer B, Canese MG, Shih JW, Purcell RH, Gerin JL: δ Agent: association of δ antigen with hepatitis B surface antigen and RNA in serum of δ-infected chimpanzees. Proc Natl Acad Sci U S A. (1980) 77(10), S. 6124–6128, PMID 6934539, PMC 350226 (freier Volltext).
- M. Rizzetto, M. G. J. Canese et al.: Transmission of the hepatitis B virus-associated delta antigen to chimpanzees. Infect. Dis. (1980) 141(5), S. 590–602, PMID 6989929.
- O. Crivelli, M. Rizzetto et al.: Enzyme-linked immunosorbent assay for detection of antibody to the hepatitis B surface antigen-associated delta antigen. J. Clin. Microbiol. (1981) 14(2), S. 173–177, PMID 7024305, PMC 271929 (freier Volltext).
- A. Kos, R. Dijkema et al.: The hepatitis delta (delta) virus possesses a circular RNA. Nature (1986) 323(6088), S. 558–560, PMID 2429192
- F. Bonino, K. H. Heermann, M. Rizzetto und W. H. Gerlich: Hepatitis delta virus: protein composition of delta antigen and its hepatitis B virus-derived envelope. J. Virol. (1986) 58(3), S. 945–950, PMID 3701932, PMC 253003 (freier Volltext).
- S. F. Elena, J. Dopazo et al.: Phylogeny of viroids, viroidlike satellite RNAs, and the viroidlike domain of hepatitis delta virus RNA. Proc. Natl. Acad. Sci. U S A. (1991) 88(13), S. 5631–5634 PMID 1712103, PMC 51931 (freier Volltext).
- T. O. Diener: Circular RNAs: relics of precellular evolution? Proc. Natl. Acad. Sci. U S A. (1989) 86(23), S. 9370–9374, PMID 2480600, PMC 298497 (freier Volltext).
Weblinks
- HDV auf Viralzone
- Webseite des Nationalen Referenzzentrums für Hepatitis-B- und -D-Viren (Institut für Medizinische Virologie Gießen)