IRES (Biologie)
Als interne ribosomale Eintrittsstelle (englisch internal ribosomal entry site), abgekürzt IRES, wird in der Zellbiologie ein spezifisch gefalteter Abschnitt innerhalb eines RNA-Einzelstrangs bezeichnet, der die Bindung an Ribosomen vermittelt.[1] Die Sekundärstruktur einer IRES ermöglicht es, bei der Synthese von Proteinen in Eukaryoten die Translation unabhängig von der 5′-Cap-Struktur zu initiieren, beispielsweise auch von der Mitte einer messenger-RNA (mRNA) aus. IRES wurden bisher in der RNA von Viren gefunden sowie in der mRNA für einige Gene von Zellen beschrieben.
Geschichte
1988 wurde erstmals eine internal ribosomal entry site (IRES) im RNA-Genom des Poliovirus und des Encephalomyocarditis-Virus in den Laboren von Nahum Sonenberg[2] und Eckard Wimmer beschrieben.[3] Der Vorgang des dadurch vermittelten Translationsbeginns wurde als internal initiation of translation bezeichnet.
Grundlagen
Üblicherweise tragen eukaryotische zelluläre mRNAs ein speziell angebundenes Nukleotid an ihrem 5'-Ende, die sogenannte 5'-Cap-Struktur, welche zusammen mit weiteren zellulären Faktoren die Bindung an Ribosomen vermittelt. Bei der Proteinbiosynthese wird damit gewöhnlich die Translation eingeleitet. Von diesem komplexen Steuerungssystem ausgenommen sind manche viralen Genome, die mittels einer IRES unmittelbar an die 40S-Untereinheit von Ribosomen binden können und so den Aufbau virentypischer Proteine durch den zellulären Syntheseapparat veranlassen. Daneben wurden in Eukaryoten auch für einige ihrer Gene – beispielsweise für das Onkogen c-Myc, die Ornithin-Decarboxylase, den Fibroblasten-Wachstumsfaktor 2 oder den endothelialen Wachstumsfaktor – in den transkribierten mRNAs besondere Sekundärstrukturen beschrieben, die auf ähnliche Weise als interne ribosomale Eintrittsstelle bei der Proteinbiosynthese funktionieren.[4] Über Cap-unabhängige Translationselemente ist darüber hinaus ein weiterer alternativer Mechanismus zur Einleitung der Translation möglich.
Manche der IRES werden in den Zusammenhang mit Krankheiten gebracht; so scheint der Funktionsverlust eines IRES-Elements auf der mRNA des Connexin 32-Gens eine Hauptursache der Charcot-Marie-Tooth-Krankheit zu sein.[5]
Die molekularen Mechanismen von viralen IRES sind umfangreicher charakterisiert als die der eukaryotischen.[6] Das IRES des Hepatitis-C-Virus bindet direkt an die P-Stelle der 40S-ribosomale Untereinheit, daher sind mit dieser IRES keine eukaryotischen Initiationsfaktoren wie eIF1, 1A, 4A, 4B und 4E nötig. Die Picornavirus-IRES bindet über eIF4 an die 40S-Untereinheit.[7] Manche viralen und eukaryotischen IRES benötigen weitere zelluläre Proteine, die als IRES-trans-acting factor (ITAF) bezeichnet werden.
Virale IRES
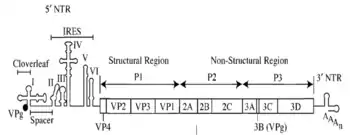
Einige RNA-Viren besitzen eine IRES, womit die Produktion von viralen Proteinen an Ribosomen der Zelle unabhängig von Initiationsfaktoren gestartet werden kann. Dies gilt beispielsweise für das Hepatitis-C-Virus und für die Picornaviren, z. B. das Poliovirus. Entdeckt wurde die IRES-Struktur beim Poliovirus, das in der Zellkultur die Synthese von zellulären Proteinen zugunsten der viralen Produkte blockiert. Da hier der zelluläre Initiationsfaktor eIF4G durch virale Enzyme gespalten wird, können nur noch solche RNA-Stränge translatiert werden, die wie die Virus-RNA eine IRES enthalten. Davon abweichende Varianten einer IRES kommen bei Dicistroviridae vor.[8]
Beispiele viraler und zellulärer IRES
| Virus | IRES |
| Poliovirus | Picornavirus IRES |
| Rhinovirus | Picornavirus IRES |
| Encephalomyocarditis virus | Picornavirus IRES |
| Foot-and-mouth disease virus | Aphthovirus IRES |
| Hepatitis-A-Virus | Hepatitis A IRES |
| Hepatitis-C-Virus | Hepatitis C IRES |
| Classical swine fever virus | Pestivirus IRES |
| Bovine viral diarrhea virus | Pestivirus IRES |
| Friend Murines Leukämievirus | |
| Moloney Murines Leukämievirus (MMLV) | |
| Rous-Sarkom-Virus | |
| Humanes Immundefizienzvirus | |
| Plautia stali intestine virus | Cripavirus internal ribosome entry site (IRES) |
| Rhopalosiphum padi virus | Cripavirus internal ribosome entry site (IRES) |
| Cricket paralysis virus | Cripavirus internal ribosome entry site (IRES) |
| Triatoma virus | Cripavirus internal ribosome entry site (IRES) |
| Kaposi's sarcoma-associated herpesvirus | Kaposi's sarcoma-associated herpesvirus IRES |
| Marek's disease virus MDV | 5'Leader IRES and intercistronic IRES in the 1.8-kb family of immediate early transcripts (IRES)1 |
| Proteintyp | Proteine |
| Wachstumsfaktoren | Fibroblast growth factor (FGF-1 IRES and FGF-2 IRES), Platelet-derived growth factor B (PDGF/c-sis IRES), Vascular endothelial growth factor (VEGF IRES), Insulin-like growth factor 2 (IGF-II IRES) |
| Transkriptionsfaktoren | Antennapedia, Ultrabithorax, MYT-2, NF-κB repressing factor NRF, AML1/RUNX1, Gtx homeodomain protein |
| Translationsfaktoren | Eukaryotic initiation factor 4G (elF4G)a, Eukaryotic initiation factor 4Gl (elF4Gl)a, Death associated protein 5 (DAP5) |
| Onkogenes | c-myc, L-myc, Pim-1, Protein kinase p58PITSLRE, p53 |
| Transporter/Rezeptoren | Cationic amino acid transporter Cat-1, Nuclear form of Notch 2, Voltage-gated potassium channel |
| Aktivatoren der Apoptose | Apoptotic protease activating factor (Apaf-1) |
| Inhibitoren der Apoptose | X-linked inhibitor of apoptosis (XIAP), HIAP2, Bcl-xL, Bcl-2 |
| Proteine in neuronalen Dendriten | Activity-regulated cytoskeletal protein (ARC), α-subunit of calcium calmodulin dependent kinase II dendrin, Microtubule-associated protein 2 (MAP2), neurogranin (RC3), Amyloid precursor protein |
| Andere | Immunoglobulin heavy chain binding protein (BiP), Heat shock protein 70, β-subunit of mitochondrial H+-ATP synthase, Ornithine decarboxylase, connexins 32 and 43, HIF-1α, APC |
Anwendungen
IRES-Elemente werden auch im Zuge eines Vektordesigns eingesetzt, beispielsweise für die Coexpression eines Reportergens zur Kontrolle der Transfektions- oder Transduktionseffizienz. Hierbei wird das zu klonierende Gen meistens vor die IRES gesetzt (in 5'-Richtung), das Reportergen dahinter. Die Expression des Reportergens zeigt die Synthese der mRNA in voller Länge an.[9]
Beschränkungen und Alternativen
Virale IRES-Sequenzen werden in der technischen Molekularbiologie häufig eingesetzt, um polycistronische mRNA nachzuahmen. Hierzu werden mehrere Gene auf einem Plasmid hintereinander kloniert und zwischen die Gene jeweils eine IRES-Sequenz geschaltet. Bei diesem Konstrukt wird nur noch ein einziger Promoter und Terminator benötigt. Allerdings haben die eingeführten IRES-Sequenzen den Nachteil, dass sich die Expressionseffizienz für jedes weitere nachfolgende Gen verringert.[10]
Eine technische Alternative zu den IRES stellen die viralen 2A-Peptide dar. Beim Einsatz von 2A-Peptid-Sequenzen wird die Genregulation eines bakteriellen Operons imitiert und eine künstlich geschaffene Reihung mehrerer Gene in einem gemeinsamen offenen Leserahmen über nur einen Promotor reguliert. Durch die je zwischengeschalteten selbst-spaltenden 2A-Peptide – Oligopeptide aus rund zwanzig Aminosäuren – werden die gemeinsam exprimierten Genprodukte in einzelne Proteine zerlegt.[11] Der Vorteil gegenüber einer Verwendung von IRES-Sequenzen ist hierbei die äquimolare Expression der Proteine über den gemeinsamen Promotor. Allerdings verbleiben nach Spaltung der 2A-Peptide kurze Peptidanteile an den Termini der exprimierten Proteine, die deren Funktion stören können.[12]
Einzelnachweise
- S. R. Thompson: Tricks an IRES uses to enslave ribosomes. In: Trends in microbiology. Band 20, Nummer 11, November 2012, S. 558–566, ISSN 1878-4380, doi:10.1016/j.tim.2012.08.002, PMID 22944245, PMC 3479354 (freier Volltext).
- Pelletier J, Sonenberg N: Internal initiation of translation of eukaryotic mRNA directed by a sequence derived from poliovirus RNA. In: Nature. 334, Nr. 6180, 1988, S. 320–5. doi:10.1038/334320a0. PMID 2839775.
- Jang SK, Kräusslich HG, Nicklin MJ, Duke GM, Palmenberg AC, Wimmer E: A segment of the 5' nontranslated region of encephalomyocarditis virus RNA directs internal entry of ribosomes during in vitro translation. In: J. Virol.. 62, Nr. 8, August 1988, S. 2636–43. PMID 2839690. PMC 253694 (freier Volltext).
- A. A. Komar, B. Mazumder, W. C. Merrick: A new framework for understanding IRES-mediated translation. In: Gene. Band 502, Nummer 2, Juli 2012, S. 75–86, ISSN 1879-0038. doi:10.1016/j.gene.2012.04.039. PMID 22555019. PMC 3361623 (freier Volltext).
- A. Huddler, R. Werner: Analysis of a Charcot-Marie-Tooth Disease Mutation Reveals an Essential Internal Ribosome Entry Site Element in the Connexin-32 Gene.
- López-Lastra M, Rivas A, Barría MI: Protein synthesis in eukaryotes: the growing biological relevance of cap-independent translation initiation. In: Biol. Res.. 38, Nr. 2–3, 2005, S. 121–46. PMID 16238092.
- Hellen CU, Sarnow P: Internal ribosome entry sites in eukaryotic mRNA molecules. In: Genes Dev.. 15, Nr. 13, 2001, S. 1593–612. doi:10.1101/gad.891101. PMID 11445534.
- E. Jan: Divergent IRES elements in invertebrates. In: Virus research. Band 119, Nummer 1, Juli 2006, S. 16–28, ISSN 0168-1702. doi:10.1016/j.virusres.2005.10.011. PMID 16307820.
- Kozak M: A second look at cellular mRNA sequences said to function as internal ribosome entry sites. In: Nucleic Acids Res.. 33, Nr. 20, 2005, S. 6593–602. doi:10.1093/nar/gki958. PMID 16314320. PMC 1298923 (freier Volltext).
- Donna Michnick, Louise C. Wasley, Monique V. Davies, Randal J. Kaufman: Improved vectors for stable expression of foreign genes in mammalian cells by use of the untranslated leader sequence from EMC virus. In: Nucleic Acids Research. Band 19, Nr. 16, 25. August 1991, ISSN 0305-1048, S. 4485–4490, doi:10.1093/nar/19.16.4485.
- Qingyou Xia, Ping Zhao, Riyuan Wang, Feng Wang, Yuancheng Wang: 2A self-cleaving peptide-based multi-gene expression system in the silkworm Bombyx mori. In: Scientific Reports. Band 5, 5. November 2015, S. 16273, doi:10.1038/srep16273, PMID 26537835, PMC 4633692 (freier Volltext).
- A. L. Szymczak-Workman, K. M. Vignali, D. A. A. Vignali: Design and Construction of 2A Peptide-Linked Multicistronic Vectors. In: Cold Spring Harbor Protocols. Band 2012, Nr. 2, Februar 2012, ISSN 1559-6095, doi:10.1101/pdb.ip067876.