Urvorfahr
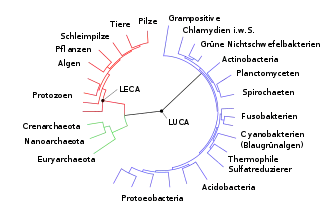
Der Urvorfahr (englisch Last Universal Ancestor, LUA, oder Last Universal Common/Cellular Ancestor, LUCA)[1][2] ist die – hypothetische – letzte gemeinsame Stammform aller heutigen (rezenten) zellulären Organismenarten. Er wird von den sogenannten monophyletischen Abstammungstheorien postuliert, die davon ausgehen, dass sämtliche Lebewesen der Erde, Pflanzen, Tiere, Pilze und einzellige Formen, sich auf eine einzige „primitive“ Urform zurückführen lassen, während alle übrigen auf der frühen Erde wahrscheinlich existierenden zeitgenössischen, ähnlich primitiven Formen keine Nachkommen in der rezenten Lebewelt und auch nicht in der jüngeren Erd- und Evolutionsgeschichte hinterlassen haben. Es wird in diesen Hypothesen davon ausgegangen, dass diese Urform vor mindestens 3,5 Milliarden Jahren, also im späten Hadaikum oder frühen Archaikum, gelebt hat. Für den Entstehungsort des Lebens und Lebensraum des LUCA wurden etliche Szenarien vorgeschlagen, in der letzten Zeit wurden aber insbesondere alkalische „Weiße Raucher“ als Hydrothermalquellen nicht zu hoher Temperatur (wie es sie beispielsweise in der „Lost City“ gibt) favorisiert.[3][4]
Hintergrund
Für den Urvorfahren wird entsprechend dem Sparsamkeitsprinzip (Ockhams Rasiermesser) üblicherweise angenommen, dass dieser diejenigen Merkmale der heute lebenden Organismen hatte, die diesen gemeinsam sind: eine Zellstruktur, DNA, den (weitestgehend übereinstimmenden) genetischen Code wie auch mRNA, tRNA und eine ribosomal vermittelte Translation.[5] Sogar DNA-Reparaturmechanismen weisen über alle zellulären Organismen und einige Viren Ähnlichkeiten (Homologien) auf: die Familie der RecA-Rekombinasen mit RecA in Bakterien, Rad51 und DMC1 in Eukaryoten, RadA in Archaeen und UvsX im Bakteriophagen T4, einem Bakterienvirus.[6] Ein weiteres Indiz für diese Annahme ist die Erkenntnis, dass Archaeen (wie Halobacterium salinarum) die gleichen Mechanismen zur Erhaltung der Zellgröße benutzen wie Bakterien und Eukaryonten, d. h. dass die Zellteilungsstrategie in allen drei Domänen des Lebens offenbar gleich ist.[7]
Da die Organisation der DNA bei Bakterien fundamental von der bei Archaeen und komplexen Zellen (Eukaryoten) abweicht, wird neuerdings die These vertreten, dass der zelluläre LUCA noch der RNA-Welt angehört hat. Die Speicherung der Erbinformation in der DNA wird dann als eine Fähigkeit angesehen, die zunächst von Retroviren ‚erfunden‘ wurde und die dann zelluläre Organismen mehrmals durch Übertragung von solchen Viren erworben haben. Daraus resultieren die Bakterien einerseits, die Archaeen und Eukaryoten andererseits. Der grundsätzliche Aufbau der Ribosomen als Eiweißfabriken und der genetische Code stimmen dagegen bei allen zellulären Organismen so gut überein, dass bereits der LUCA darüber verfügt haben muss.[8]
Darüber hinaus ist es möglich, dass der Urvorfahr thermophil war.[9] Das wird allerdings zunehmend angezweifelt, weil diese Hypothese auf vergleichenden Sequenzanalysen der rRNA (ribosomalen RNA) basierte, die bei thermophilen Organismen fragwürdige Ergebnisse liefern.[10] Es scheint, dass seine Zellwandbestandteile Murein enthielten, eine häufige Zellwandkomponente moderner Bakterien.[11][12]
Anmerkungen zu möglichen Fehlschlüssen:
- Es handelt sich beim Last Universal Ancestor nicht um ein bestimmtes Individuum, sondern um ein Taxon im Rang einer Art und somit um eine Population.
- Diese Urpopulation repräsentiert nicht zwangsläufig auch die ersten existierenden (zellulären) Organismen.
- Folglich handelte es sich auch nicht zwingend um die „primitivst“-möglichen Lebensformen.
Die Entstehung und Entwicklung des genetischen Codes und der Ribosomen hätte dann vor dem LUCA bereits in der RNA-Welt stattgefunden. Vorher standen als Biokatalysatoren nur Ribozyme zur Verfügung, danach auch echte (Protein-)Enzyme (Zwei Phasen der RNA-Welt). Moderne Ribosomen bestehen aus beidem, ribosomaler RNA (rRNA) und (Hilfs-)Proteinen. Wenn man unter dem Begriff Progenot einen Organismus versteht, der noch im Begriff ist, die Beziehung zwischen Genotyp und Phänotyp zu entwickeln, dann wäre dieser Organismus in der ersten Phase anzusiedeln, also vor dem LUCA, der vermutlich in der zweiten Phase anzusiedeln ist.[13] Ähnlich wird manchmal der Begriff Protozelle oder Protobiont verstanden: In einem sich replizierenden Vesikel wird RNA repliziert (siehe auch Mikrosphäre).[14]
Eine statistische Untersuchung aus dem Jahr 2010 hat ergeben, dass das Leben sehr wahrscheinlich von einem einzigen gemeinsamen Vorfahren abstammt.[15] Ein einziger gemeinsamer Vorfahr ist danach 102860-mal wahrscheinlicher als mehrere.[16] All seine Zeitgenossen sind seither ausgestorben; lediglich das genetische Erbe hätte bis zum heutigen Tag überlebt.
Im Gegensatz dazu propagierte Carl Woese, dass unser genetisches Prä-LUCA-Erbe von einer Vielzahl an Organismen entstammte, als nur von einer Spezies (Biofilmtheorie, Horizontaler [lateraler] Gentransfer). Die Fähigkeit primitiver Lebewesen zum horizontalen Gentransfer könnte nämlich bedeuten, dass anstelle eines einzigen universellen Ahns eine Gemeinschaft (Genpool) primitiver Einzeller tritt.[17][18][4] Da diese sich untereinander im stetigen Genaustausch befanden ('Common ancestral community'), könnten sie aus heutiger Sicht quasi als eine Einheit (Species) erscheinen. Dieser Genaustausch könnte u. a. auch durch Viren vermittelt worden sein.
Der Aufbau von Dendrogrammen (Baumdiagrammen) auf der Grundlage genetischer Distanz zwischen allen existenten Zelltypen zeigt eine relativ frühe Aufspaltung zwischen Archaeen, die hoch resistent gegenüber extremen Lebensbedingungen (extremophil) sind, und den übrigen Lebensformen. Dies hat zu einigen Vermutungen darüber geführt, dass sich der Urvorfahr in solch extremen Ökosystemen entwickelt haben könnte, etwa im Tiefseerücken.
Zwei oder drei Domänen
Neue Untersuchungstechniken haben in den Jahren seit etwa 2015 das Bild stark verändert. Die bisherigen Stammbäume beruhten auf dem Vergleich homologer DNA-Sequenzen vor allem aus der kleineren Untereinheit der ribosomalen RNA. Die für die Amplifikation verwendeten Methoden, vor allem die verwendeten Primer, erwiesen sich nun für eine Reihe ungewöhnlicher Prokaryoten als ungeeignet. Diese Organismen waren der Aufmerksamkeit der Biologen bis dahin entgangen, weil sie sich mit den in Jahrzehnten entwickelten Standard-Techniken der Mikrobiologie nicht kultivieren ließen, und großenteils bis heute nicht lassen. Viele von ihnen besitzen ein sehr kleines Genom, ihnen fehlen für Lebensvorgänge wesentliche Synthese- und Stoffwechselwege,[19] weshalb man annimmt, sie können womöglich nur als Symbionten oder Parasiten, gemeinsam mit anderen Organismen, überleben. Da die formale Beschreibung von Prokaryotentaxa an Kultivierung gebunden ist, verharren die neu gefundenen Linien auf dem Status eines Candidatus. Tatsächlich wurden aber zahlreiche Taxa, die aufgrund ihrer Divergenz im Rang von Stämmen (Phyla) eingeordnet werden müssen, gefunden, bei denen nicht ein kultivierbares Mitglied enthalten ist. Ihre Existenz verrät sich durch Analysemethoden, bei denen ohne Umweg über Kultivierung direkt Genome in der Umwelt analysiert und dann einzelnen Linien/Arten zugeordnet werden (genannt Metagenomik) oder bei denen das Genom einer einzelnen, isolierten Zelle amplifiziert und sequenziert wird.[20]
Eine solche Gruppe, die zu den Archaeen gehörenden „Lokiarchaeota“[21][22] erwies sich unerwarteterweise nach genetischen Analysen als nahe verwandt mit den Eukaryoten. Danach wurden weitere Schwestergruppen der „Lokiarchaeota“ gefunden („Thorarchaeota“, „Odinarchaeota“, „Heimdallarchaeota“ etc.); alle zusammen werden zur „Asgard“-Supergruppe zusammengefasst.[23][24]
Ein mit Hilfe der neuen Techniken 2016 aufgestellter neuer Stammbaum des Lebens, der auf mehr als 3000 vollständig analysierten Genomen beruht,[25] fand nicht nur eine neue Großgruppe der Bakterien, die (mit einer Ausnahme) ausschließlich bisher unkultivierbare Bakterienstämme enthält. Ein Ergebnis war außerdem, dass der Ursprung der Eukaryoten innerhalb der Archaeen liegt, und nicht, wie bisher angenommen,[26] unabhängig von diesen ist. Danach würden nur zwei (statt bisher drei) Domänen des Lebens existieren, denn die Eukaryoten wären zu einer Linie innerhalb der Archaeen reduziert.[27] Allerdings war dies in Form der Eozyten-Hypothese schon früher vorgeschlagen worden,[28] galt aber bisher als im Widerspruch zu den Befunden. Jetzt käme als ein solcher Ausgangspunkt der Eukaryoten die Asgard-Supergruppe der Archaeen in Frage.
Eine weitere Unterstützung dieser Zweiteilung der Lebewesen kommt aus der Erkenntnis, dass die DNA-Replikationssysteme der Bakterien auf der einen und der Archaeen sowie Eukaryoten auf der anderen Seite sich derart stark voneinander unterscheiden, dass – im Gegensatz zu den Ribosomen und dem Genetischen Code – für diese kein gemeinsamer Ursprung (Homologie) angenommen werden kann. Möglicherweise hatte der zelluläre LUCA daher noch (wie viele Viren) ein RNA-Genom, war also ein (hypothetischer) Ribozyt.[8][29]
Ähnliches gilt für an DNA-Reparaturmechanismen betrachtete Proteine. Während es für die Proteine aus der Familie der RecA-Rekombinasen wie oben erwähnt Homologien über alle zellulären Organismen (und einige Viren) gibt, finden sich für weitere Reparaturproteine Homologien unter Eukaryoten und Archaeen (Rad54, Mre11, und Rad50).[6][30][31]
In Zusammenhang mit diesen Entdeckungen wurden auch andere bisherige Ansichten zum systematischen Wert grundlegender Unterschiede in Frage gestellt, etwa zum Aufbau der Lipide der Zellmembran[32] oder zum Ursprung der grampositiven Bakterien.[33] Da jedoch die ATP-Synthase allen Lebewesen in ihrer komplexen Grundstruktur gemein ist (mit Ausnahme von wenigen rein gärenden Mikroben sowie primitiven Vielzellern wie einigen Myxozoen[34][35] und Korsetttierchen[36][37]), sollte der LUCA bereits über eine Basisversion davon verfügt haben. Da die ATP-Synthase ein membrangebundenes Protein ist, sollte der LUCA bereits über Membranen (in irgendeiner Form) verfügt haben, so dass in ihm ein zumindest primitiv-zellulärer Organismus zu vermuten ist, etwa mit semipermeabler Membranhülle (statt wie bei heutigen Zellen mit impermeabler Membranhülle und aktiven Transportmechanismen).[38] Die Analyse der Proteinfamilien der verschiedenen Untereinheiten membranständiger F- und A-/V-Typ-ATPasen erlaubt Rückschlüsse auf die detaillierte Entstehungs- und Entwicklungsgeschichte dieser 'motorischen' Enzyme. Dies stützt weiter die Annahme, dass LUCA bereits ein zellulärer Organismus mit einer zumindest primitiven Form von Membranen war, und dass die Eukaryoten sich aus einem Zweig der Archaeen entwickelt haben.[39][40][4]
Eine weitere Unterstützung der Annahme eines gemeinsamen Ursprungs aller zellulären Organismen könnte sich aus der vergleichenden stöchiometrischen Analyse fundamentaler Stoffwechselwege (englisch metabolic pathways) von Modellorganismen ergeben. Am MIT hatten Jean-Benoît Lalanne, Gene-Wei Li und Kollegen in einer Veröffentlichung im März 2018 über eine entsprechende Untersuchung an verschiedenen Bakterien und an der Bäckerhefe eine bemerkenswerte Übereinstimmung gefunden. Die evolutionären Kräfte, die hinter dieser hochkonservierten Stöchiometrie stehen, blieben aber zunächst im Dunkeln.[41][42]
Die bisher erzielten Ergebnisse sind aufgrund ihrer Neuartigkeit noch vorläufig, sie stehen teilweise im Widerspruch zu den anhand der traditionellen Methoden erzielten Erkenntnissen und müssen daher noch umfassend geprüft werden. Sie deuten aber auf die Möglichkeit hin, dass eventuell einige Prokaryoten mit sehr kleinem Genom möglicherweise nicht auf die Reduktion komplexer organisierter Vorfahren zurückgehen, sondern dass es sich um reliktäre Linien handeln könnte, die in der modernen Welt nur als Symbionten überleben konnten.
Urviren
Im Gegensatz zu den zellulären Organismen (Biota inklusive Mensch) gibt es offenbar verschiedene Urviren (virale LCAs).[8] Obwohl auch äußerst verschiedene Viren homologe Hüll-Proteine besitzen, gibt es zwanzig oder mehr grundsätzlich verschiedene solche Proteine, die nicht miteinander verwandt sind, was gegen einen gemeinsamen Vorfahren aller Viren spricht.[43] Viren scheinen in einer Periode starken horizontalen Gentransfers durch Reduktion aus urtümlichen Zellen entstanden, die ein segmentiertes RNA-Genom besaßen (Ribozyten, RNA-Welt), nicht von einem „modernen“ Gegenstück.[44] Im Einklang mit der RNA-Welt- und Ribozyten-Hypothese müssen die ursprünglichen Viren RNA-Viren gewesen sein, bevor diese die DNA zur Speicherung der Erbinformation entdeckt hätten.[8]
Urkaryoten, Archezoen und LECA
Der letzte gemeinsame Vorfahr der Eukaryoten wird in Analogie zum Last Universal Common Ancestor gelegentlich als Last Eukaryotic Common Ancestor, abgekürzt LECA, bezeichnet.[45]
Alle bisher untersuchten Eukaryoten besitzen, neben einem vom Cytoplasma durch eine Kernmembran abgegrenzten Zellkern, Mitochondrien oder Organellen (Hydrogenosom, Mitosom etc.), die offenbar von Mitochondrien abstammen oder mit diesen einen gemeinsamen Vorfahren haben. Zumindest ließen sich überall in der Zellkern-DNA Gene mitochondrialen Ursprungs nachweisen als Resultat eines lateralen Gentransfers von einem früheren Mitochondrium zum Kern.[46][47] Daher sollte LECA bereits einen Zellkern und mitochondrienartige Organellen (mit eigener mtDNA) besessen haben.
Falls der Erwerb des Zellkerns dem der Mitochondrien vorausging, muss es vorher amitochondriale Organismen mit Zellkern gegeben haben, die im Laufe ihrer Evolution die Mitochondrien durch Endosymbiose erworben haben (siehe Endosymbiontentheorie). Carl Woese und George Fox führten 1977 für diese hypothetischen Formen von „primitiven“ amöboiden prädatorischen Einzellern den Begriff Urkaryoten (engl. urkaryotes) ein[48] und Thomas Cavalier-Smith führte für rezente, primitive amitochondriale Einzeller, die er für Abkömmlinge dieser Urkaryoten hielt, die Gruppenbezeichnung Archezoa ein.[49] Allerdings mehrten sich nachfolgend Hinweise darauf, dass es sich bei den Archezoen um sekundär amitochondriale echte Eukaryoten handelt,[50] das heißt um Abkömmlinge von LECA.
Als Alternative zur Urkaryoten-Hypothese wird ein Erwerb von Mitochondrien durch Archaeen diskutiert (Wasserstoff-Hypothese[51]) vor oder gleichzeitig mit dem Zellkern, der eventuell viralen Ursprungs (unter den NCLDVs) ist.[8]
Archaeen und Bakterien – LACA und LBCA
In Analogie wird der letzte gemeinsame Vorfahr der Archaeen als LACA bezeichnet;[52][53] der der Bakterien (siehe Urbakterium) als LCBA,[54] manchmal auch LBCA.[53]
LUCELLA und weitere Begriffe
Der letzte gemeinsame Vorfahre aller zellulären Organismen (Bakterien einerseits, Archaeen inkl. Eukaryoten andrerseits) wird gelegentlich als LUCELLA bezeichnet[55] – nach der RNA-Welt-Hypothese könnte dies ein Ribozyt (Zelle mit RNA-Genom) gewesen sein.
Weitere Begriffe dieser Art setzen oft bestimmte Annahmen voraus, so z. B. LAECA (last archaeo-eukaryotic common ancestor) eine parallele Entwicklung von Archaeen und Eukaryoten. Nach der Eozyten-Hypothese entwickelten sich die Eukaryoten aber aus einem Zweig der Archaeen heraus (dabei endosymbiotische Aufnahme eines α-Proteobakteriums als Mitochondrium), der LAECA ist dann identisch mit dem LACA.
Literatur
- Nicolas Glansdorff, Ying Xu, Bernard Labedan: The Last Universal Common Ancestor : emergence, constitution and genetic legacy of an elusive forerunner. Biology Direct 2008, 3:29.
Weblinks
- Urahn allen Lebens war hitzescheu, scinexx vom 2. Januar 2009. Springer-Verlag, Heidelberg. Quelle: University of Montreal, NPO
- SWR2 Wissen – Manuskriptdienst: Wie das Leben auf die Erde kam. (PDF; 67 kB), Radio Akademie: Evolution – Fluss des Lebens (3), 16. Mai 2009 – Autor: Uwe Springfeld, Redaktion: Detlef Clas, Regie: Günter Maurer.
- William F. Martin (Bild): Ursprung des Lebens : «Luca» liebte es heiss – Ein kurioser Einzeller ist der gemeinsame Vorfahr allen Lebens auf der Erde. Nun haben Forscher rekonstruiert, wie er selbst gelebt hat, dpa/NZZ 25. Juli 2016.
- Joseph F. Sutherland: On The Origin Of Tha Bacteria And The Archaea, auf B.C vom 16. August 2014
- Mart Krupovic, Valerian V. Dolja, Eugene V. Koonin: The LUCA and its complex virome, in: Nature Rev Microbiol, Nr. 18, 14. Juli 2020, S. 661–670, doi:10.1038/s41579-020-0408-x
- Frank Schubert: Urahn aller Lebewesen ernährte sich von Wasserstoff, spektrum.de vom 13. Dezember 2021
- New study sheds light on origins of life on Earth, EurekAlert! vom 14. Januar 2022
Einzelnachweise
- Patrick Forterre: Looking for LUCA. Memento im Webarchiv vom 20. Juli 2021.
- Buchvorstellung von Susanne Liedtke: Matt Ridley, Die Geschichte von Luga – „Alphabet des Lebens“. In: bild der wissenschaft.
- Frank Schubert: Urahn aller Lebewesen ernährte sich von Wasserstoff, auf: spektrum.de vom 13. Dezember 2021
- Nick Lane: Der Funke des Lebens - Energie und Evolution, Konrad Theiss Verlag, (C) 2017 by WBG, ISBN 978-3-8062-3484-8. Englischer Originaltitel: Nick Lane: The Vital Question - Energy, Evolution, and the Origins of Complex Life, 2015-07-20, ISBN 978-0-393-08881-6, PDF (Memento vom 10. September 2017 im Internet Archive). Anm.: In der deutschen Ausgabe heißt es 'alkaline hydrothermale Schlote' statt korrekt 'alkalische'. Siehe insbes. Abb./Fig. 20 und 16. (der wundersame sich selbst auflösende Stammbaum).
- Xiaoxia Liu, Jingxian Zhang, Feng Ni, Xu Dong, Bucong Han, Daxiong Han, Zhiliang Ji, Yufen Zhao: Genome wide exploration of the origin and evolution of amino acids. In: BMC Evolutionary Biology. Band 10, 15. März 2010, ISSN 1471-2148, S. 77, doi:10.1186/1471-2148-10-77 (englisch).
- Haseltine CA, Kowalczykowski SC: An archaeal Rad54 protein remodels DNA and stimulates DNA strand exchange by RadA. In: Nucleic Acids Research. 37, Nr. 8, Mai 2009, S. 2757–2770. doi:10.1093/nar/gkp068. PMID 19282450. PMC 2677860 (freier Volltext).
- Archaea, Bacteria, and Eukarya Have More in Common Than Previously Thought, auf SciTechDaily vom 2. Januar 2018
- Patrick Forterre: Evolution – Die wahre Natur der Viren. In: Spektrum der Wissenschaft. Heft 8/2017, S. 37 (Online-Artikel veröffentlicht am 19. Juli 2017); Anmerkung: Der Autor spricht dabei von „mehreren viralen LUCAs“, scheint die Bezeichnung „LUCA“ folglich als Synonym für LCA/MRCA zu verwenden
- Dawn J. Brooks, Eric A. Gaucher: A thermophilic last universal ancestor inferred from its estimated amino acid composition. S. 200–207 in: David A. Liberles (Hrsg.): Ancestral Sequence Reconstruction. Oxford University Press, Oxford (UK) 2007, ISBN 978-0-19-929918-8, Kapitel-doi:10.1093/acprof:oso/9780199299188.001.0001 (alternativer Volltextzugriff: Foundation for Applied Molecular Evolution)
- Nicolas Galtier, Nicolas Tourasse, Manolo Gouy: A Nonhyperthermophilic Common Ancestor to Extant Life Forms. In: Science. Band 283, Nr. 5399, 8. Januar 1999, S. 220–221, doi:10.1126/science.283.5399.220 (englisch).
- Arthur L. Koch: The Exocytoskeleton. In: Journal of Molecular Microbiology and Biotechnology. Band 11, Nr. 3–5, S. 115–125, doi:10.1159/000094048 (englisch).
- Arthur L. Koch: The Exoskeleton of Bacterial Cells (the Sacculus): Still a Highly Attractive Target for Antibacterial Agents That Will Last For a Long Time. In: Critical Reviews in Microbiology. Band 26, Nr. 1, Januar 2000, ISSN 1040-841X, S. 1–35, doi:10.1080/10408410091154165 (englisch).
- Johann Peter Gogarten, David Deamer: Is LUCA a thermophilic progenote?. In: Nature Microbiology. 1, 25. November 2016, S. 16229. doi:10.1038/nmicrobiol.2016.229.
- I. A. Chen, P. Walde: From self-assembled vesicles to protocells. In: Cold Spring Harbor perspectives in biology. Band 2, Nummer 7, Juli 2010, S. a002170, doi:10.1101/cshperspect.a002170, PMID 20519344, PMC 2890201 (freier Volltext) (Review).
- Douglas L. Theobald, A formal test of the theory of universal common ancestry. nature.com. Abgerufen am 15. Januar 2011., Nature, Band 465, 2010, S. 219–222
- All Present-day Life Arose From A Single Ancestor. sciencenews.org. Abgerufen am 15. Januar 2011.
- New Mexico Museum of Natural History and Science: Tree of Life – Introduction, dort insbesondere die interaktive Info zu ‘Who were they’
- F. L. Sousa, T. Thiergart, G. Landan, S. Nelson-Sathi, I. A. Pereira, J. F. Allen, N. Lane, W. F. Martin: Early bioenergetic evolution. In: Philosophical transactions of the Royal Society of London. Series B, Biological sciences. Band 368, Nummer 1622, Juli 2013, S. 20130088, doi:10.1098/rstb.2013.0088, PMID 23754820, PMC 3685469 (freier Volltext) (Review), Figure 6: The amazing disappearing tree
- Christopher T. Brown, Laura A. Hug, Brian C. Thomas, Itai Sharon, Cindy J. Castelle, Andrea Singh, Michael J. Wilkins, Kelly C. Wrighton, Kenneth H. Williams, Jillian F. Banfield (2015): Unusual biology across a group comprising more than 15% of domain Bacteria. Nature 523, S. 208–211. doi:10.1038/nature14486
- Lindsey Solden, Karen Lloyd Kelly Wrighton (2016): The bright side of microbial dark matter: lessons learned from the uncultivated majority. Current Opinion in Microbiology 31, S. 217–226. doi:10.1016/j.mib.2016.04.020
- Anja Spang, Jimmy H. Saw, Steffen L. Jørgensen, Katarzyna Zaremba-Niedzwiedzka, Joran Martijn, Anders E. Lind, Roel van Eijk, Christa Schleper, Lionel Guy, Thijs J. G. Ettema (2015): Complex archaea that bridge the gap between prokaryotes and eukaryotes. Nature 521: 173–179. doi:10.1038/nature14447
- Loki's Castle and the New Tree of Life: Two Domains and the CPR Hidden Folk. University of Hawai'i Reed Lab, Floyd A. Reed, 24. Juni 2016.
- K. W. Seitz et al.: Genomic reconstruction of a novel, deeply branched sediment archaeal phylum with pathways for acetogenesis and sulfur reduction, Research Gate, Januar 2016
- Katarzyna Zaremba-Niedzwiedzka et al.: Asgard archaea illuminate the origin of eukaryotic cellular complexity. In: Nature. 541, 2017, S. 353–358. doi:10.1038/nature21031.
- Laura A. Hug, Brett J. Baker, Karthik Anantharaman, Christopher T. Brown, Alexander J. Probst, Cindy J. Castelle, Cristina N. Butterfield, Alex W. Hernsdorf, Yuki Amano, Kotaro Ise, Yohey Suzuki, Natasha Dudek, David A. Relman, Kari M. Finstad, Ronald Amundson, Brian C. Thomas, Jillian F. Banfield (2016): A new view of the tree of life. Nature Microbiology 1, Article number: 16048. doi:10.1038/nmicrobiol.2016.48
- Carl R. Woese, Otto Kandler, Mark L. Wheelis (1990): Towards a natural system of organisms: Proposal for the domains Archaea, Bacteria, and Eucarya. Proceedings of the National Academy of Sciences USA 87: S. 4576-4579.
- Kasie Raymann, Céline Brochier-Armanet, Simonetta Gribaldo (2015): The two-domain tree of life is linked to a new root for the Archaea. Proceedings of the National Academy of Sciences USA 112 (21): S. 6670–6675. doi:10.1073/pnas.1420858112
- James A. Lake, E. Henderson, M. Oakes, M. W. Clark: Eocytes: A new ribosome structure indicates a kingdom with a close relationship to eukaryotes. Proceedings of the National Academy of Sciences USA 81, 1984, S. 3786–3790.
- Yarus M: Primordial genetics: phenotype of the ribocyte. In: Annu. Rev. Genet.. 36, 2002, S. 125–51. doi:10.1146/annurev.genet.36.031902.105056. PMID 12429689.
- Huang Q, Liu L, Liu J, Ni J, She Q, Shen Y: Efficient 5'-3' DNA end resection by HerA and NurA is essential for cell viability in the crenarchaeon Sulfolobus islandicus. In: BMC Molecular Biology. 16, 2015, S. 2. doi:10.1186/s12867-015-0030-z. PMID 25880130. PMC 4351679 (freier Volltext).
- Lin Z, Kong H, Nei M, Ma H: Origins and evolution of the recA/RAD51 gene family: evidence for ancient gene duplication and endosymbiotic gene transfer. In: Proceedings of the National Academy of Sciences of the United States of America. 103, Nr. 27, Juli 2006, S. 10328–10333. bibcode:2006PNAS..10310328L. doi:10.1073/pnas.0604232103. PMID 16798872. PMC 1502457 (freier Volltext).
- Laura Villanueva, Stefan Schouten, Jaap S. Sinninghe Damsté (2016): Phylogenomic analysis of lipid biosynthetic genes of Archaea shed light on the ‘lipid divide’. Environmental Microbiology 19: 54–69. doi:10.1111/1462-2920.13361
- Sergio A. Muñoz-Gómez & Andrew J. Roger (2016): Phylogenomics: Leaving negative ancestors behind. eLife 2016; 5:e20061. doi:10.7554/eLife.14589
- Jan Osterkamp: Erstes Tier ohne Atmung und Mitochondrien, auf: Spektrum.de vom 25. Februar 2020
- Tel Aviv University researchers discover unique non-oxygen breathing animal, auf: EurekAlert! vom 25. Februar 2020
- Andy Coghaln: Zoologger: The mud creature that lives without oxygen, auf: NewScientist vom 7. April 2010
- Roberto Danovaro et al.: The first metazoa living in permanently anoxic conditions, in: BMC Biology, Band 8, Nr. 30, 6. April 2010, doi:10.1186/1741-7007-8-30
- Armen Y Mulkidjanian, Michael Y Galperin, Kira S Makarova, Yuri I Wolf and Eugene V Koonin: Evolutionary primacy of sodium bioenergetics. In: Biology Direct. 3, Nr. 13, 2008. doi:10.1186/1745-6150-3-13.
- Armen Y Mulkidjanian, Kira S Makarova, Michael Y Galperin, Eugene V Koonin: Inventing the dynamo machine: the evolution of the F-type and V-type ATPases. In: Nature Reviews Microbiology. 5, Nr. 11, 2007, S. 892-899. doi:10.1038/nrmicro1767. Dieser Artikel bei Uni Osnabrück: Perspectives (PDF) (Seite nicht mehr abrufbar, Suche in Webarchiven) Info: Der Link wurde automatisch als defekt markiert. Bitte prüfe den Link gemäß Anleitung und entferne dann diesen Hinweis.
- Armen Y Mulkidjanian, Michael Y Galperin, Eugene V Koonin: Co-evolution of primordial membranes and membrane proteins. In: Trends Biochem Sci.. 4, Nr. 34, 2009, S. 206–215. doi:10.1016/j.tibs.2009.01.005. PMC 2752816 (freier Volltext). (Seite nicht mehr abrufbar, Suche in Webarchiven) Info: Der Link wurde automatisch als defekt markiert. Bitte prüfe den Link gemäß Anleitung und entferne dann diesen Hinweis.
- Justin Chen: New Discovery Suggests All Life May Share This Common Design Principle, auf SciTechDaily vom 30. März 2018
- Jean-Benoît Lalanne, James C. Taggart, Monica S. Guo, Lydia Herzel, Ariel Schieler, Gene-Wei Li: Evolutionary Convergence of Pathway-Specific Enzyme Expression Stoichiometry, Cell, Band 173, Nr. 3, 19. April 2018 (online 29. März 2018), S. 749-761.e38, doi:10.1016/j.cell.2018.03.007
- M. Krupovic et al.:Multiple origins of viral capsid proteins from cellular ancestors, PNAS 114 (12) vom 3. Februar 2017, doi:10.1073/pnas.1621061114, Abschnitte Significance und Abstract
- A. Nasir et al.:A phylogenomic data-driven exploration of viral origins and evolution, Science Advances AAAS, Vol. 1 (8), 25. September 2015, doi:10.1126/sciadv.1500527
- Eugene V. Koonin: The origin and early evolution of eukaryotes in the light of phylogenomics, in: BioMed Central: Genome Biology 201011:209 vom 5. Mai 2010, doi:10.1186/gb-2010-11-5-209
- Brigitte Boxma, Rob M. de Graaf, Georg W. M. van der Staay, Theo A. van Alen, Guenola Ricard, Toni Gabaldon, Angela H. A. M. van Hoek, Seung Yeo Moon-van der Staay, Werner J. H. Koopman, Jaap J. van Hellemond, Aloysius G. M. Tielens, Thorsten Friedrich, Marten Veenhuis, Martijn A. Huynen, Johannes H. P. Hackstein: An anaerobic mitochondrion that produces hydrogen. In: Nature. Band 434, Nr. 7029, 3. Februar 2005, S. 74–79, doi:10.1038/nature03343.
- A. Akhmanove et al.: A hydrogenosome with a genome. In: Nature. Band 396, Nr. 6711, 10. Dezember 1998, S. 527–528, doi:10.1038/25023.
- C. R. Woese, G. E. Fox: Phylogenetic structure of the prokaryotic domain: the primary kingdoms. In: Proceedings of the National Academy of Sciences. Band 74, Nummer 11, November 1977, S. 5088–5090, PMID 270744, PMC 432104 (freier Volltext).
- Tom Cavalier-Smith: Archaebacteria and Archezoa. In: Nature. 339, Nr. 6220, Mai 1989, S. 100–101. doi:10.1038/339100a0. PMID 2497352.
- Patrick J. Keeling: A kingdom’s progress: Archezoa and the origin of eukaryotes. In: BioEssays. Band 20, Nummer 1, Januar 1998, S. 87–95, doi:10.1002/(SICI)1521-1878(199801)20:1<87::AID-BIES12>3.0.CO;2-4 (alternativer Volltextzugriff: SemanticScholar).
- W. Martin, M. Müller: The hydrogen hypothesis for the first eukaryote. In: Nature. 392, Nr. 6671, 1998, S. 37–41. doi:10.1038/32096. PMID 9510246.
- Marta Kwapisz, Frédéric Beckouët, Pierre Thuriaux: Early evolution of eukaryotic DNA-dependent RNA polymerases. In: Trends in Genetics. 24, 2008, S. 211, doi:10.1016/j.tig.2008.02.002, 3. Abbildung.
- Joana C. Xavier, Rebecca E. Gerhards, Jessica L. E. Wimmer, Julia Brueckner, Fernando D. K. Tria, William F. Martin: The metabolic network of the last bacterial common ancestor, Band 4, Nr. 413, 26. März 2021. Dazu:
Nadja Podbregar: Urahn aller Bakterien rekonstruiert – Ur-Bakterium war anaerob, stäbchenförmig und ähnelte heutigen Clostridien, auf: scinexx vom 7. April 2021. - S. Hou, K. S. Makarova, et al.: Complete genome sequence of the extremely acidophilic methanotroph isolate V4, Methylacidiphilum infernorum, a representative of the bacterial phylum Verrucomicrobia. In: Biology direct. Band 3, Juli 2008, S. 26, doi:10.1186/1745-6150-3-26, PMID 18593465, PMC 2474590 (freier Volltext).
- Supriya Patil, Kiran Kondabagil: Coevolutionary and Phylogenetic Analysis of Mimiviral Replication Machinery Suggest the Cellular Origin of Mimiviruses, in: Molecular Biology and Evolution, Band 38, Nr. 5, Mai 2021, S. 2014–2029, doi:10.1093/molbev/msab003, Epub 11. Februar 2021.