Lentiviren
Lentiviren sind behüllte Einzel(+)-Strang-RNA-Viren, (ss(+)RNA) und bilden die Gattung (Genus) Lentivirus innerhalb der Familie der Retroviren (Retroviridae). Die Bezeichnung Lentiviren (langsame Viren) leitet sich von lateinisch lentus ‚langsam‘ ab, da viele dieser Viren langsam fortschreitende, chronisch degenerative Krankheiten auslösen. Andere wiederum verursachen keine Erkrankung ihres Wirts. Lentiviren sind sehr Spezies-spezifisch und wurden bisher nur in verschiedenen Säugetierarten gefunden. Lentiviren verbleiben lebenslang im Wirt, da sie Abwehrmechanismen des Immunsystems umgehen können. Sie können im Gegensatz zu den anderen Retroviren auch nicht teilungsaktive, eukaryotische Zellen infizieren. Bekanntester Vertreter der Lentiviren ist das Humane Immundefizienz-Virus (HIV).
| Lentiviren | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
HI-Virus, Grafik | ||||||||||||||||||
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| Lentivirus | ||||||||||||||||||
| Links | ||||||||||||||||||
|
Geschichte
Bereits 1904 wurde mit der Ansteckenden Blutarmut der Einhufer (Equine Infektiöse Anämie) die erste von Lentiviren ausgelöste Krankheit beschrieben.[2] Das die Krankheit auslösende Virus der equinen infektiösen Anämie (EIAV) wurde jedoch erst sehr viel später entdeckt.
In den 1930ern und 1940ern wurde in Island erstmals die Maedi-Visna-Erkrankung bei Schafen beobachtet, das auslösende Virus wurde in den 1950ern als das erste Lentivirus, das Maedi-Visna-Virus, abgekürzt MVV, beschrieben. In diesem Zusammenhang wurde von dem Beschreiber Björn Sigurdsson auch der Begriff Langsame Virusinfektionen geprägt, der zunächst für verschiedene langsam fortschreitende Infektionskrankheiten, darunter auch Scrapie, verwendet wurde[3] und der zu der Bezeichnung „Lentiviren“ führte.[4]
1981 wurde die Immunschwächeerkrankung Aids beschrieben und zwei Jahre später wurde am Institut Pasteur in Paris das die Krankheit auslösende HI-Virus entdeckt, das inzwischen das am besten untersuchte Virus überhaupt ist. Aids ist jedoch immer noch nicht heilbar und hat sich zu einer Pandemie entwickelt.
Einteilung
Die Gattung Lentivirus wird in fünf verschiedene Untergruppen eingeteilt, die nach den Wirten benannt sind, die sie infizieren: bovine, equine, ovine/caprine, feline Lentiviren und die Lentiviren der Primaten. Dazu gehören zum Beispiel die folgenden Arten:
- Bovine Lentiviren:
- Bovines Immundefizienz-Virus (BIV) der Rinder
- Jembrana-Disease-Virus der Rinder
- Equine Lentiviren:
- Virus der equinen infektiösen Anämie (EIAV) der Equiden (Pferde, Esel)
- Feline Lentiviren:
- Ovine/caprine Lentiviren:
- Maedi-Visna-Virus (MVV) der Schafe
- Caprines Arthritis-Enzephalitis-Virus (CAEV) der Ziegen
- Primaten-Lentiviren:
- Humanes Immundefizienz-Virus (HIV) mit den beiden Arten HIV-1 und HIV-2
- Simianes Immundefizienz-Virus (SIV) bei verschiedenen Affen, mit verschiedenen Unterformen wie etwa SIVcpz (Schimpansen).[5]
- Bovine Lentiviren:
EIAV und Jembrana gehören zu den Lentiviren, sie können jedoch im Gegensatz zu den meisten anderen Lentiviren auch eine akute Erkrankung hervorrufen.
Unterschiede zu anderen Retroviren und evolutionäre Entwicklung
Lentiviren unterscheiden sich von anderen Retroviren durch ein charakteristisches Merkmal: Sie können im Gegensatz zu beispielsweise Alpha- oder Gammaretroviren die Regulation ihrer eigenen Gene beeinflussen. Aus diesem Grund werden sie zu den komplexen Retroviren gerechnet. Sie sind außerordentlich variabel in ihrer Nukleotidsequenz. Durch die Abwesenheit eines Fehlerkorrekturmechanismus und die Einsträngigkeit des RNA-Genoms weisen sie die mit am schnellsten evolvierenden Genome auf.[6]
Lentiviren produzieren durch alternatives Spleißen sehr viele verschiedene mRNAs, im Fall von HIV sind es mehr als 20, während einfache Retroviren meist nur zwei verschiedene mRNA-Varianten exprimieren, eine gespleißte und eine ungespleißte. Sie können dadurch mindestens zehn verschiedene Proteine produzieren. Das lentivirale Genom weist eine sehr starke Kondensation auf. Die offenen Leserahmen von tat, rev und env überschneiden sich bei HIV.[6]
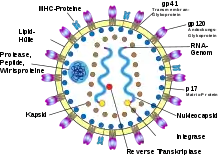
Aufbau
Lentiviren gehören zu den komplexen Retroviren. Sie sind behüllt und haben einen Durchmesser von 80–100 nm. Sie haben wie alle Retroviren in ihrem Genom drei „Hauptgene“, gag, pol und env, die für die viralen Proteine codieren und die in allen Lentiviren in der Reihenfolge 5'-gag-pol-env-3' angeordnet sind. Außerdem haben sie verschiedene zusätzliche Gene, die auch als akzessorische Gene (englisch accessory oder auch auxiliary) bezeichnet werden und die sich von Lentivirus zu Lentivirus unterscheiden können. Diese Gene beziehungsweise ihre Produkte sind an der Regulation, Synthese und Prozessierung der viralen RNA beteiligt und wirken teilweise den Abwehrmechanismen der Wirtszellen entgegen. HIV-1 enthält beispielsweise die akzessorischen Gene vif, vpr, vpu, tat, rev und nef. Die Long-Terminal-Repeat-Region (LTR-Region) der Lentiviren ist etwa 600 Nukleotide (nt) lang, davon entfallen auf die U3-Region etwa 450 nt, die R-Region 100 und auf die U5-Region 70 nt. Durch die akzessorischen Gene ist das Genom der Lentiviren im Schnitt etwas größer als das der einfachen Retroviren.
Zu den Transaktivatoren, die die Transkriptionseffizienz des LTR-Promotors erhöhen, gehören Tat, Tax und Tas. Rev und Rex sind Transaktivatoren, die posttranskriptionell wirken.
nef
nef negative factor ist ein myristyliertes intrazelluläres Protein, das dafür sorgt, dass der CD4-Rezeptor runterreguliert wird und nicht mehr an der Zelloberfläche erscheint.
rev
rev (regulator of expression of virion proteins) codiert für ein 13–19 kDa großes Protein, das in Tetrameren oder größeren Aggregaten vorliegt. Es reichert sich im Zellkern an und bindet an RRE, das rev-responsive element in der viralen mRNA, wodurch diese RNAs bevorzugt aus dem Kern exportiert und prozessiert werden, was eine gesteigerte Expressionseffizienz zur Folge hat.
tas
Tas Transaktivator of spumaviruses, auch Bel1 genannt, ist ein nucleäres Protein, das in humanen Spumaviren an das BRE-Element um U3-Bereich von humanen Spumaviren bindet.
tat
tat (transactivator of transcription) codiert für die Tat-Proteine, die im Verlauf des Replikationszyklus als erste Virusproteine hergestellt werden. Sie haben die Funktion, die Transkriptionseffizienz des HI-Virus zu verstärken. Tat bindet dazu an die transactivation-responsive region (TAR), die am 5'-Ende der viralen Transkripte zu finden ist und bewirkt eine verbesserte Elongation. Tat ist somit ein RNA-bindendes-Protein, die TAR-Region ist das erste RNA-Enhancer-Element, das beschrieben wurde.[7] Tat-Proteine werden durch eine Acetyltransferase aktiviert, welche dazu Acetylreste auf Lysylreste des Proteins überträgt.[8]
vif
vif (viral infectivity factor) codiert für das Vif-Protein, das im Viruspartikel an die verpackte RNA bindet. Es hat eine Größe von 22–30 kDa und befähigt die Partikel, den zellulären APOBEC3-Abwehrmechanismus der Wirtszelle zu umgehen.
vpr
vpr codiert für das Virale Protein R (Vpr). Vpr ist ein kleines basisches Protein von 96 Aminosäuren, das in HIV-1, HIV-2 und SIV konserviert ist und in die Viruspartikel verpackt wird, indem es an das Gag-Polyprotein bindet. Im Verlauf der Infektion hat Vpr verschiedene Funktionen wie beispielsweise einen Effekt auf die Korrektheit der reversen Transkription, den Import des Präintegrationskomplexes in den Zellkern, den Verlauf des Zellzyklus, die Regulation der Apoptose sowie auf die Transaktivierung der HIV-LTRs und verschiedener Wirtsgene.[9]
vpu
vpu (viral protein u) wurde im Genom von HIV-1 und von SIVcpz gefunden. Es ist 80–82 Basenpaare lang und hat die Funktion, in manchen humanen Zelltypen die Freisetzung von Viruspartikeln zu fördern. Vpu von HIV-1 der Gruppe M (major) bewerkstelligt dies, indem es einen zellulären Virusabwehrmechanismus, der durch das Protein Tetherin vermittelt wird, unterdrückt.[10]
vpx
vpx codiert für das Virale Protein X (Vpx). Es besteht aus 104 bis 119 Aminosäuren und hat eine molare Masse zwischen 12 und 16 kDa. Das Protein wird in die Viruspartikel verpackt.
Dieses Gen findet sich lediglich im Genom des humanen Lentivirus HIV-2 sowie in den Primaten-SIVs der afrikanischen Grünen Meerkatzen (SIVagm), der Makaken (SIVmac), der Weißlid-Mangaben (SIVsm) und der Mandrills (SIVdrill). Im Genom von HIV-1 oder des Schimpansen-SIV (SIVcpz) hingegen ist vpx nicht vorhanden. vpx ähnelt jedoch dem vpr von HIV-1 und SIVcpz und erfüllt ähnliche Funktionen. vpx ist bei einem Rekombinationsereignis in Grünen Meerkatzen aus vpr entstanden.[11]
Replikationszyklus
Der Replikationszyklus von Lentiviren läuft wie der aller Retroviren ab, mit der Besonderheit, dass Lentiviren die Fähigkeit haben, die Kernhülle zu überwinden und somit auch ruhende, sich nicht in Teilung befindende Zellen zu infizieren. Der Eintritt in den Zellkern erfolgt nach der Bildung des Präintegrationskomplexes (PIC) im Zytoplasma. Da die Kernporen kleiner sind als der PIC, muss es sich um einen aktiven Transportprozess handeln. An diesem Prozess sind sowohl zelluläre als auch virale Proteine beteiligt.[12]
Biotechnologische Verwendung
Lentiviren werden in der Gentechnik als virale Vektoren eingesetzt, um gezielt Gene in Zielzellen zu schleusen und dort zu exprimieren.
Einzelnachweise
- ICTV: ICTV Taxonomy history: Commelina yellow mottle virus, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- H. Vallée, H. Carré: Sur la nature infectieuse de l'anémie du cheval. In: Comptes rendus des seánces de l'Académie des Sciences. Série D: Sciences naturelles. Band 139, 1904, S. 331–333 (französisch).
- Slow Viruses from World of Genetics.
- Björn Sigurdsson: Rida, a chronic encephalitis of sheep. With general remarks on infections which develop slowly and some of their special characteristics. In: Br Vet J. Band 110, 1954, S. 341–354 (englisch). Zitiert nach Massimo Palmarini: A Veterinary Twist on Pathogen Biology. In: PLOS Pathogens. 2007, doi:10.1371/journal.ppat.0030012 (englisch).
- Dezhong Xu, Huimin Sun, Haixia Su, Lei Zhang; Jingxia Zhang, Bo Wang, Rui Xu: SARS coronavirus without reservoir originated from an unnatural evolution, experienced the reverse evolution, and finally disappeared in the world, in: Chinese Medical Journal, Band 127, Nr. 13, 5. Juli 2014, S. 2537–2542, doi:10.3760/cma.j.issn.0366-6999.20131328
- G. Myers, G. N. Pavlakis: Evolutionary potential of complex retroviruses. In: J. Levy (Hrsg.): The Retroviridae. Vol. 1. Plenum Press, New York 1992, Seiten 51–105.
- John Brady and Fatah Kashanchi: Tat gets the „green“ light on transcription initiation. In: Retrovirology. Band 2, 2005, S. 69. PMID 16280076.
- Wilma Dormeyer: Die biochemische Analyse der Proteinacetylierung am Beispiel des HIV-1 Tat Proteins. Dissertation, 2003, Ruhr-Universität Bochum (PDF; 2,2 MB)
- E. Le Rouzic, S. Benichou: The Vpr protein from HIV-1: distinct roles along the viral life cycle. In: Retrovirology. 2005, 2:11.
- S. J. Neil, T. Zang, P. D. Bieniasz: Tetherin inhibits retrovirus release and is antagonized by HIV-1 Vpu. Nature. 2008 Jan 16, PMID 18200009.
- T. M. Fletcher, 3rd, B. Brichacek, N. Sharova, M. A. Newman, G. Stivahtis, P. M. Sharp, M. Emerman, B. H. Hahn, M. Stevenson: Nuclear import and cell cycle arrest functions of the HIV-1 Vpr protein are encoded by two separate genes in HIV-2/SIV(SM). EMBO J. 1996 November 15, 15(22): 6155–6165, PMID 8947037.
- Y. Suzuki, R. Craigie: The road to chromatin – nuclear entry of retroviruses. Nat Rev Microbiol. 2007 Mar, 5(3):187-96. Review, PMID 17304248.