Polycomb-Körper
Polycomb-Körper (englisch polycomb bodies, PcG bodies) sind Proteinkomplexe im Zellkern, die unter anderem in Pflanzen, Fliegen (wie Drosophila) und Säugetieren nachgewiesen wurden. Sie sind vermutlich an Teile des Chromatins gebunden.[1][2] Nach einer Immunfärbung entstehen mikroskopisch sichtbare Strukturen innerhalb des Zellkerns. Ihre Größe und Anzahl variiert innerhalb verschiedener Zelltypen, und ihre Verteilung ist abhängig vom Entwicklungszustand der Zelle. Sie enthalten eine hohe Konzentration an Polycomb-Proteinen. Diese Proteine der Polycomb-Gruppe sind wichtige Faktoren, die die Stilllegung von Entwicklungsgenen auch über mehrere Zellteilungen hinweg aufrechterhalten. Sie sind für die normale Entwicklung mehrzelliger Organismen unerlässlich. In Säugetieren sind sie an grundlegenden Prozessen wie dem zellulärem Gedächtnis, Zellproliferation, genomischer Prägung, X-Inaktivierung und der Krebsentwicklung beteiligt. In Pflanzen sind sie u. a. an der Zelldifferenzierung und der Vernalisation – der Prozess, durch den das Blühen durch lang anhaltende Kälte beschleunigt wird – beteiligt.[3][4] Die Regulation der Zellentwicklung durch Polycomb-Proteine wurde intensiv anhand des Modellorganismus Drosophila melanogaster erforscht. Die Polycomb-Proteine modifizieren die Histone des Chromatins in der Weise, dass Gene unzugänglich für die Proteine der Transkriptionsmaschinerie der Zelle und damit praktisch stillgelegt werden. Dies wird Gen-Silencing genannt. Die so stillgelegten DNA-Abschnitte sammeln sich in den Polycomb-Körpern. Diese Stilllegung kann später auch wieder aufgehoben werden.
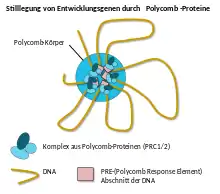
Zum Aufbau der Polycomb-Körper gibt es mehrere Modelle, die sie entweder als proteinbasierte Strukturen ansehen oder als Körperchen, die aus Chromatin bestehen. Die Polycomb-Körper bewegen sich innerhalb des Zellkerns und bringen so unterschiedliche Bereiche des Chromatins in Verbindung. Polycomb-Proteine werden innerhalb der Polycomb-Körper ständig ausgetauscht.
Einführung
Innerhalb eukaryotischer Zellen enthält der Zellkern das Erbgut in Form der DNA, sichtbar als Chromatin. Zu einem bestimmten Zeitpunkt wird jeweils jedoch nur ein Teil der Gene benötigt. Dies ist abhängig vom Entwicklungszyklus der Zelle und vom Zelltyp. Hierbei muss die Zelle eine Auswahl treffen, welche Gene aktiv sind und welche nicht. Die DNA der nicht benötigten Gene wird kompakt zusammengefaltet und bildet das Heterochromatin. Die DNA wickelt sich hier kompakt zu Nukleosomen auf, wird mit weiteren Proteinen gebunden und stark komprimiert. So sind die Gene unzugänglich für die Enzyme, die diese ablesen. Bei aktiven Genen ist das Chromatin nicht so kompakt ausgebildet; es bilden sich DNA-Schleifen aus, von denen die Gene abgelesen werden. Insbesondere sind hier die Regulationsbereiche für Gene wie Promotoren und Enhancer für die Regulationsproteine zugänglich. Diese dreidimensionale Organisation von Genen ist wichtig zur Regulierung der Genexpression, das heißt, wie die Information der Gene in Proteine umgesetzt werden kann.
Zentraler Bestandteil von Nukleosomen sind die Histon-Proteine, deren Eigenschaften durch Bindung von weiteren Proteinen verändert werden können. Die Funktion von Polycomb-Proteinen ist unter anderem, die Transkription von Genen zu unterdrücken. Dies geschieht unter anderem durch chemische Änderung der Histon-Proteine. Eine dieser Änderungen ist die Methylierung einiger Histone. Ein zentraler Teil der Stilllegung der DNA ist die Methylierung des Histons Nr. 3 (bezeichnet als H3K27me3) innerhalb eines Nukleosoms. Dies geschieht mit Hilfe von Komplexen aus Polycomb-Molekülen.
Während der Entwicklung eines Organismus müssen sich die Zellen unterschiedlich differenzieren. Dabei sind nacheinander unterschiedliche Gene aktiv. Dieses Genexpressionsmuster erfordert eine genaue Steuerung. Unterstützt wird dies durch epigenetische Mechanismen, indem ein zelluläres Gedächtnis aufgebaut wird, welches auch durch Replikation und Zellteilung hindurch stabil an deren Tochterzellen weitergegeben wird. Dies wurde bei der Fruchtfliege Drosophila intensiv erforscht. Dabei wurden epigenetische Regulatoren in Form von zwei Gruppen von Genen entdeckt: Die Gene der Polycomb-Gruppe (PcG) und der Trithorax-Gruppen (TrxG). Die Enzyme von PcG und TrxG vermitteln die Modifikation der Histone. Durch die Proteine der Polycomb-Gruppe kann ein Gen dauerhaft stummgeschaltet werden, während mit TrxG Gene aktiviert werden können.
Die PcG-Proteine sind nicht zufällig im Zellkern verteilt, sondern sammeln sich in mehreren Zentren zusammen, den Polycomb-Körpern. Sie unterscheiden sich in Größe und Anzahl in verschiedenen Zelltypen; in der Regel sind in undifferenzierten Zellen immer weniger und größere Körper vorhanden, während in differenzierten Zellen immer mehr und kleinere Körper auftreten.[5] Sie enthalten durch Polycomb-Proteine stillgelegte Gene.[6] Polycomb-Körper bewegen, treffen und teilen sich während der Entwicklung dynamisch.[6] Pro Zelle liegt die Anzahl dieser Körper zwischen 12 und 16 und der Durchmesser beträgt 0,3–1,0 µm.[7] Polycomb-Körper unterscheiden sich in Größe und Menge an Polycomb-Proteinen. Insbesondere PcG-Domänen, das heißt DNA-Abschnitte mit einer längeren linearen Größe, zeigen einen höheren Gehalt an Polycomb-Proteinen und erzeugen größere Polycomb-Körper.[8]
Polycomb-Körper wurden in Säuger- und Drosophila-Zellkernen durch Immunfärbung mit Antikörpern gegen PcG-Proteine beobachtet.[9] Durch mikroskopische Aufnahmen oder auch durch Live-Bildgebung von fluoreszenzmarkierten Proteinen wurde gezeigt, dass PcG-Proteine in einer relativ kleinen Anzahl von Polycomb-Körpern lokalisiert sind.[10]
Mit genomweiten Analysen mithilfe von Techniken wie der Chromosome Conformation Capture konnte die Verteilung von Polycomb-Proteinen und der Markierung der Histone bestimmt werden. Die Ergebnisse deuten auf eine hierarchische Organisation der regulierten Gene hin. Die erste Ebene besteht aus kurzen einzelnen Regionen der DNA, die durch PcG-Proteine gebunden sind. Hierzu gehören die PREs (Polycomb Response Elements). Diese DNA-Regionen fungieren als Antwortelemente, die notwendig sind, um PcG-Proteine zu rekrutieren und angrenzende Gene stillzulegen.[6] Die zweite Ebene dieser Organisation wird bestimmt durch das Clustering einzelner PREs in Polycomb-Domänen. Diese Regionen werden mit dem Histon H3K27me3 und teilweise durch Polycomb-Proteine markiert. Diese PREs können miteinander interagieren, um dreidimensionale Strukturen zu bilden.[6] Auf einer solchen hierarchischen Organisationsebene von PcG-Domänen befinden sich die Polycomb-Körper. Die Polycomb-Domänen können Kontakte über weit entfernte DNA-Regionen hinweg vermitteln. Dadurch bildet sich ein Netzwerk aus Polycomb-Proteinen und DNA aus.[6] Dabei entstehen Chromatin-Schleifen mit PcG-gebundenen regulatorischen Elementen und den Promotoren von PcG-Zielgenen, deren Transkription unterdrückt wird.[8] Bei PREs wurde gezeigt, dass sie weit entfernte Stellen auf dem gleichen oder einem anderen Chromosom kontaktieren können.[6] Diese Kontakte werden wahrscheinlich durch nicht-kodierende RNAs (ncRNA), Isolatoren oder über RNA-Interferenz (RNAi) vermittelt.[8][6]
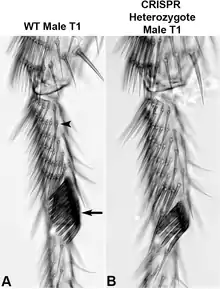
Im Jahr 1947 wurde das erste Gen der Polycomb-Gruppe, Polycomb, in Drosophila melanogaster von Pamela Lewis entdeckt.[1] Bei Drosophila haben männliche Fliegen an ihrem ersten Beinpaar eine borstige Struktur, den sogenannten Geschlechtskamm (sex comb). Dies dient zum Festhalten des Weibchens während der Begattung. Die Analyse einer Fliegenmutante, die zusätzliche Sexualkämme auf dem zweiten und dritten Beinpaar („polycomb“) besitzt, ermöglichte es den Forschern, das erste Polycomb-Element (Pc) zu identifizieren.[11]
Später wurden bei der Untersuchung homöotischer Gene die Funktion von Polycomb-Proteinen als negativer Regulator entdeckt. Diese Gene sind für die korrekte Körpersegmentierung während der Entwicklung notwendig. Insgesamt wurden 18 PcG-Gene in Drosophila entdeckt, die die korrekte Abfolge der Aktivierung des Hox-Genclusters regulieren.[11]
Bei Pflanzen ist die Existenz von Polycomb-Körpern schwierig nachzuweisen. Es gibt jedoch mehrere Indizien dafür, dass sich Polycomb-Komponenten oder ihre Ziel-Gene zu Clustern zusammenlagern.[5] Dort unterdrückt das Protein "LPH1", das funktionale pflanzliche Homolog von Polycomb, die Genexpression. Durch bildgebende Verfahren konnte im pflanzlichen Modellorganismus Arabidopsis (Ackerschmalwand) das Verteilungsmuster von LHP1 im Zellkern von einem einheitlichen Muster in meristematischen Zellen bis hin mehreren unterscheidbaren Foci in ausdifferenzierten Zellen variieren. Diese Beziehung zwischen der LHP1-Verteilung und dem Differenzierungsstatus der Zelle erinnert an die Verbindung zwischen Polycomp-Körpern und der Zelldifferenzierung bei Tieren.[5]
Beteiligte Proteine und DNA-Abschnitte
PcG-Proteine
Die Proteine der Polycomb-Gruppen sind wichtige Entwicklungsregulatoren, die die Expression von Hunderten von Genen steuern.[6] Ihre Aktivität ist notwendig, um ein "epigenetisches Gedächtnis" spezifischer Genexpressionsmuster zu erhalten. Als epigenetische Repressoren haben sie die wichtige Funktion, das Gedächtnis von Transkriptionsprogrammen während der Entwicklung und Differenzierung von Lebewesen zu erhalten. Die durch PcG-Proteine vermittelte Gen-Stilllegung tritt in Polycomb-Körpern auf. Hierbei vermitteln die in den Polycomb-Körpern befindlichen PcG-Komponenten die Chromatin-Kondensation ihrer Zielgene. Dies geschieht, indem sich mehrere Proteine der PcG-Gruppe zu Molekülkomplexen (PRC1 und PRC2) verbinden. Diese können die Histone der Nukleosomen so modifizieren, dass diese sich mit der daran aufgewickelten DNA zu einer kompakten Chromatinstruktur zusammenlagern. Dadurch werden die darin enthaltenen Gene sozusagen stillgelegt, da die DNA hier nicht mehr abgelesen werden kann (Gen-Silencing). Es hat sich gezeigt, dass PcG-Komplexe Chromatin auch in vitro verdichten und die DNA-Zugänglichkeit in vivo reduzieren.[6][8]
Meistens sind PcG-Proteine diffus im Zellkern verteilt. In einigen Zelltypen bilden sie jedoch auch Körper, die durch Fluoreszenzmikroskopie sichtbar sind, unabhängig davon, mit welcher Methode sie aufgenommen wurden. Das Verteilungsmuster der PcG-Proteine und der damit verbundenen Histonmarkierungen ist zelltypabhängig.[12][6]
PcG-Proteine können ihre Aktivität durch Interaktionen über eigentlich weit entfernte Abschnitte der DNA auch auf verschiedenen Chromosomen vermitteln. Dies ist die Grundlage, damit die Genexpression räumlich und zeitlich koordiniert werden kann. So trägt die dreidimensionale Organisation von PcG-Proteinen wesentlich zu deren Funktion bei. Mithilfe von PcG-Proteinen werden intra- und sogar interchromosomale Interaktionen über weite Entfernungen zwischen PcG-Zielen innerhalb des Zellkerns etabliert. Dadurch wird ein hohes Maß an Chromatin-Organisation im 3D-Kernraum erzeugt. Diese Kontakte von weit entfernten DNA-Abschnitten könnten durch nicht-kodierende RNAs, Isolatoren, DNA-Elementen und RNAi-Maschinen vermittelt werden.[8]
In Drosophila gibt es spezifische regulatorische DNA-Abschnitte, die Polycomb Group Response Elements (PREs) genannt werden. An diesen Stellen binden die PcG-Proteinkomplexe die DNA. Studien mit Drosophila lieferten hier experimentelle Beweise für eine sequentielle Bindung von PcG-Komplexen an diese PREs. Zum Beispiel legen PcG-Proteine die Hox-Gene durch Bindung an cis-regulierende DNA-Module still.[11][10]
Der Gegenspieler zu den PcG-Proteinen sind die Proteine der Trithorax(TrxG)-Gruppe. Sie arbeiten an mehreren hundert entwicklungsrelevanten Zielgenen antagonistisch zu den Proteinen der Polycomb-Gruppe, um aktive Transkriptionszustände aufrechtzuerhalten.[13] Im Allgemeinen arbeiten Proteine der Polycomb-Gruppe (PcG) mit Transkriptionsrepressoren zusammen, um die Genabschaltung zu erhalten, während Proteine der Trithoraxgruppe durch entsprechende Transkriptionsaktivatoren eine Genaktivierung ermöglicht.[8] Die Genstillegung durch Polycomb-Proteine ist über viele Zellgenerationen hinweg stabil und kann nur durch Keimbahndifferenzierungsprozesse überwunden werden.
Die repressiven Funktionen von PcG sind meist mit posttranslationalen Modifikationen von Histonen verbunden. Danach folgt eine Hemmung der Chromatin-Remodellierung und eine Verdichtung der Chromatins.[12] Die Enzymkomplexe von PcG und TrxG funktionieren, indem sie an die regulatorischen Regionen der Entwicklungsgene wie zum Beispiel PRE binden. Sie regulieren dadurch das komplizierte Gleichgewicht zwischen der Selbsterneuerung von Stamm- und Vorläuferzellen und der Durchführung der zellulären Differenzierung. Im Laufe der Differenzierung binden diese regulatorischen Regionen einen dieser beiden Proteinkomplexe und werden ausschließlich von den Proteinen PcG oder TrxG besetzt. Diese Bindung erfolgt zelllinienabhängig, sodass die Chromatinstruktur dieser Gene entweder im aktiven oder im stillen Zustand fixiert wird.
PRC1/PRC2
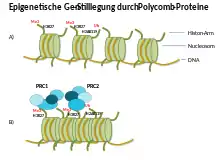
Die verschiedenen PcG-Proteine verbinden sich zu funktionell unterschiedlichen Komplexen, die zu zwei großen Familien gehören: den Polycomb repressive complex 1 und 2 (PRC1 und PRC2). Die Molekül-Komplexe jeder Familie zeigen eine katalytische Aktivität.[11] Die Aminosäuresequenz von PRC2 ist evolutionär konservierter als von PRC1. Jeder Komplex besteht aus mehreren Proteinen mit unterschiedlichen biochemischen Funktionen, von denen viele nicht gut verstanden sind. Anscheinend verändern PcG-Komplexe die Chromatin-Umgebung, bei dem sie durch ihre katalytische Aktivität Histone modifizieren und auch die Kondensierung des Chromatins veranlassen. Zudem regulieren PRC1 und PRC2 die Aktivität der RNA-Polymerase.[11][13]
PRC1 enthält die Proteine Ph (Polyhomeotic), Psc (Posterior Sex Combs), Sce (Sex Comb Extra)/Ring und Pc (Polycomb).[14] PRC2 enthält Esc, Su(z)12, p55/CAF und die Histonmethyltransferase E(z).[14] Bei Säugetieren hat sich der PRC1-Komplex jedoch im Laufe der Evolution erheblich erweitert, was zur Existenz mehrerer Orthologen pro PRC1-Komponente führte. Menschliche Zellen kodieren fünf HPC (CBX), sechs PSC, drei HPH und zwei SCE-Orthologe. Der typische PRC1-Komplex enthält je einen einzigen Vertreter aus jeder Genfamilie.[12] Alle PRC1-Protein-Komplexe enthalten einen Kern, der in den wichtigsten Tierlinien und in Pflanzen, nicht aber in Pilzen konserviert ist.[1]
In der Pflanze Arabidopsis ist das Protein "LIKE HETEROCHROMATIN PROTEIN1" (LHP1) ein funktionelles Homolog von Pc. Dort kolokalisiert LHP1 mit den epigenetischen Markierungen im gesamten Genom und interagiert mit PRC1- und PRC2-Mitgliedern sowie mit einer langen nicht-kodierenden RNA. Es wird zur Unterdrückung vieler Polycomb-Zielgene in Arabidopsis benötigt.[15]
Durch diese Protein-Komplexe werden posttranslationale Modifikationen von Histonproteinen vermittelt. Hierbei werden Modifikationen wie Acetylierung, Methylierung, Phosphorylierung und Ubiquitination durchgeführt, um einen kombinatorischen "Histoncode" zu erzeugen. Dies dient dazu, zelllinienspezifische Muster der Chromatinstruktur während der gesamten Entwicklung zu regulieren.
Der PRC2-Komplex besitzt eine Histonmethyltransferase-Aktivität. Er trimethyliert das Histon H3 auf Lysin Nr. 27 (daher die Abkürzung H3K27me3, "K" ist das Symbol für die Aminosäure Lysin). PRC2 ist erforderlich für das Auswählen der Genomregion (hier PRE), die stillgelegt werden soll. PRC1 wird für die Stabilisation dieser Stilllegung benötigt. PRC1 wirkt durch die Vermittlung der Ubiquitinierung des 119. Lysinrestes des Histons H2A; dies wird durch zwei der PRC1-Proteine erreicht, die als Ringfingerprotein 1A und 1B bezeichnet werden. Die Monoubiquitinierung von Lysin 119 auf H2A und die Di-/Trimethylierung von Lysin 27 auf H3 durch PRC1 bzw. PRC2 sollen die Gentranskription direkt blockieren.[11]
Diese posttranslationale Modifikation führt dazu, die lokale Chromatinstruktur in einen transkriptionell repressiven Zustand zu bringen. Ihre korrekte Etablierung ist entscheidend für die koordinierte Stilllegung von Genen während der gesamten Entwicklung von Säugetieren.
Polycomb Response Element (PRE)
Die Polycomb Response Elemente (PREs) wurden in Drosophila-Genen entdeckt und als DNA-Fragmente definiert, die die Aufrechterhaltung der stillgelegten Expression eines Transgens bewirken.[14] PREs rekrutieren den PRC2-Komplex, der das Histon 3 eines Nukelosoms dreifach methyliert. Dies ist eine Markierung, die von PcG-Proteinen des PRC1-Komplexes erkannt wird, um die Gen-Stilllegung zu bewirken.[6] PREs können insgesamt mehrere Aufgaben erfüllen: Sie rekrutieren Proteine der PcG- und TrxG-Familie. Sie nehmen abhängig vom Status von Promotoren und Enhancern einen aktiven oder einen stillgelegten Zustand an. Diesen Zustand können sie für längere Zeit aufrechterhalten. Der Zustand kann jedoch bei neuen Signalen wieder gewechselt werden.[13] Es bleibt der Zustand erhalten, der ursprünglich durch Transkriptionsfaktoren bestimmt wurde. Diese Aufrechterhaltung kann über viele Zellgenerationen hinweg bestehen, auch wenn die anfänglich bestimmenden Transkriptionsfaktoren fehlen. Hiermit haben PREs die Möglichkeit, ein stabiles epigenetisches Gedächtnis von stillgelegten als auch von aktiven Transkriptionszuständen zu liefern. Während die Eigenschaften von PREs und den DNA-Sequenzen, die sie definieren, bei Drosophila gut charakterisiert sind, haben sich ihre Gegenstücke bei Säugetieren als schwer fassbar erwiesen.[13] Dort wurden nur einige wenige PREs identifiziert; es hat sich jedoch gezeigt, dass 97 % der PRC2-Ziele CpG-Inseln oder ähnlichen CG-reichen Regionen entsprechen. Sie stellen bei Säugetieren PRE-ähnliche Sequenzen dar, die PcG- und TrxG-Komplexe rekrutieren können. Den PREs fehlt jedoch ein Konsensusmotiv.[5][1]
Es wurde gezeigt, dass PREs verschiedener PcG-Ziele auch über große Entfernungen hinweg miteinander interagieren können, zum Beispiel werden in Drosophila PcG-Proteine über PRE-Sequenzen zum Chromatin rekrutiert, die mehrere zehn Kilobasen von ihren Zielgenen entfernt sein können.[5]
Weitere Untersuchungen zeigten, dass PcG-Komplexe, die an verschiedene PREs gebunden sind weder notwendig noch ausreichend sind, eine Fernwirkung zwischen den PcG-Bindungsstellen zu vermitteln. Es wurde gezeigt, dass Isolatoren und nicht PREs Assoziationen zwischen PcG-Zielen vermitteln, um Polycomb-Körper zu bilden.[9] Versuche zeigten auch, dass ein zwischen einem PRE- und einem PcG-Zielgen platzierter Isolator die Wechselwirkung zwischen dem PRE und dem entfernten Promotor verhinderte. Dadurch wurde dessen Stilllegung blockiert. Deshalb kann davon ausgegangen werden, dass es eher die isolatorbindende Proteine und nicht die PcG-Komplexe sind, die für die höhere Organisation von PcG-Zielen im Zellkern verantwortlich sind. Mit Isolatoren kann die Chromatin-Konformation so geändert werden, dass sich Chromatinschleifen ausbilden. Mit diesen Schleifen ist der Isolator in der Lage, ein stromaufwärts gelegenes PRE mit einem stromabwärts gelegenen Gen in Kontakt zu bringen. Andererseits scheinen PcG-Proteine auch zur Funktion von Isolatorproteinen beizutragen.[12]
Aufbau
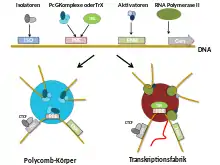
Polycomb-Körper gelten als proteinbasierte Strukturen, die durch Anhäufung von PcG-Proteinen gebildet werden. Jedoch zeigen andere Studien, dass sie eher als eine chromosomale Domäne angesehen werden denn als ein proteinbasiertes Kernkörperchen. Darauf deuten auch Experimente zur Kinetik hin, die die vollständige Rückgewinnung von PcG-Proteinen außerhalb der Polycomb-Körper zeigen. Frühere Studien haben die gleiche Anzahl von Polycomb-Körpern im Zellkern wie die Anzahl der auf Polytänchromosomen beobachteten Bänder festgestellt. Dies deutet darauf hin, dass Polycomb-Körper durch PcG-Proteine gebildet werden, die an ihr Zielchromatin binden. Einige Studien besagen, dass Polycomb-Körper als Wirtsorte für PcG-Zielgene dienen. Elektronenmikroskopisch konnte gezeigt werden, dass Polycomb-Körper Bereichen entsprechen, die aus kondensierten, ca. 100 nm dicken Chromatinfäden bestehen. Zusätzlich kommen noch Gene dazu, die PcG binden: Es wird erwartet, dass die PcG-Zielgene eine weitere Komponente des Polycomb-Körpers sind. Die Bedeutung für die strukturelle Basis diese Komponenten ist jedoch ziemlich umstritten.[12] Die Abschnitte der DNA, die an PcG-Proteine binden, bilden aus ihrem chromosomalen Kontext Schleifen und sind in den proteinbasierten Polycomb-Körper lokalisiert. Dort werden die dort vorhandenen Gene stillgelegt. Dies deutet darauf hin, dass die DNA-Schleifen die Polycomb-Körper als stilllegende Fabriken nutzen, anstatt strukturell zu ihrem Aufbau dienen.[12][6]
Für den Aufbau von Polycomb-Körpern gibt es drei Modelle, die die gerade erwähnten Punkte berücksichtigen:[12]
A) Ein Polycomb-Körper bildet sich wie ein typisches Kernkörperchen, basierend auf Proteinen. Er bildet sich durch eine Anhäufung von PcG-Proteinen und ist im Interchromatin-Kompartiment (siehe Chromosomenterritorium) angesiedelt.
B) Polycomb-Körper bilden eine Ansammlung von DNA-reichem Chromatin. An diese DNA gebunden sind die PcG-Proteine.
C) Der Polycomb-Körper wird durch eine Ansammlung von Polycomb-Proteinen gebildet, die an ihre Zielgene gebunden sind. Diese Gene bilden Schleifen aus ihrem Chromatin-Kontext heraus. Der Polycomb-Körper ist hier im Euchromatin lokalisiert.
Polycomb-Körper kolokalisieren mit dem Histon H3K27me3 und bilden kleine Kerndomänen von unterschiedlicher Intensität. Polycomb-Körper gehören nicht zu den am stärksten verdichteten Chromatinanteilen des euchromatischen Teils des Genoms. Diese Verteilung von Polycomb-Körpern steht im Einklang mit einer früheren Studie mit der Elektronenmikroskopie, die gezeigt hat, dass Polycomb-Moleküle im Perichromatin-Kompartiment des Säugerkerns konzentriert ist. Im Zellkern ist das dekondensierte Perichromatin am Rand des inaktiven Chromatins angeordnet. Darüber hinaus scheinen RNAs auch eine bedeutende Rolle bei der PcG-vermittelten Genabschaltung zu spielen. Beispielsweise erwiesen sich RNAi-Komponenten als wichtig für das Clustering von PREs oder der Funktion von Isolatoren. RNAs könnten die wichtige Botenstoffe und Regulatoren von Strukturkomponenten des PcG Körpers sein.[12][6]
Dynamik
Die strukturelle Beschaffenheit und Funktion von Polycomb-Körpern und die Zusammensetzung dieser Körper durch unterschiedliche Varianten der Polycomb-Proteine wurden durch verschiedene Experimente untersucht. Hierbei wurde auch die Kinetik von Polycomb-Proteinen erforscht. Im Allgemeinen wurde gezeigt, dass Polycomb-Proteine schnell in diesen Körpern ausgetauscht wurden.[12] Dies deutet darauf hin, dass Polycomb-Körper aus Polycomb-Proteinen bestehen. Weitere Analysen ergaben, dass die Anzahl der Polycomb-Körper und die Anzahl Bänder, die auf Polytänchromosomen sichtbar sind, gleich sind. Die beobachtete Kolokalisation von PcG-Zielgenen mit Polycomb-Körpern in diploiden Zellen bestätigt diese Ansicht. Die Dynamik von Polycomb-Proteinen außerhalb der Polycomb-Körpern scheint höher zu sein als innerhalb der Polycomb-Körper. Die Polycomb-Proteine bewegen sich in den Körpern langsamer und es gibt eine größere Variabilität ihrer Bewegung.[12][6]
Polycomb-Körper bewegen, treffen und teilen sich während der Entwicklung einer Zelle dynamisch. Ihre Bewegung hat lässt sich in zwei Kategorien einteilen: eine schnelle, aber stark eingeschränkte Bewegung und eine langsamere. Innerhalb des schnelleren Geschwindigkeitsbereiches bewegen sich Polycomb-Körper in Volumina, die etwas größer sind als die von kondensierten Chromatin-Domänen. Bei der langsamen Bewegung unterziehen sich Chromatin-Domänen und Polycomb-Körper koordiniert weiträumigen Bewegungen, die der Bewegung ganzer Chromosomenterritorien (das Gebiet innerhalb des Zellkern, welches ein einzelnes Chromosom belegt) entsprechen können.
Beide Bewegungsabläufe verlangsamen sich während der Entwicklung zunehmend, was darauf hindeutet, dass die Regulierung der Chromatindynamik eine wichtige Rolle bei der Aufrechterhaltung der Genabschaltung in differenzierten Zellen spielen kann. Durch Zeitraffer-Bildgebung von Chromosomen wurde gezeigt, dass einige Abschnitte des Chromatins eine brownsche Bewegung erfahren. Die Bewegung jedes Abschnitts beschränkte sich jedoch auf eine Teilregion des Zellkerns. Die Bewegung chromosomaler Abschnitte wurde als konsistent mit einem random walk beschrieben. Die schnelle Bewegung von Polycomb-Körpern und Chromatin-Domänen, die während der frühen Embryogenese beobachtet wurde, nimmt in späten Entwicklungsstadien stark ab, was auf einen möglichen Beitrag der Chromatindynamik zur Aufrechterhaltung eines stabilen Gen-Silencing hinweist. Auch die Temperatur beeinflusst die Bewegung von Chromatin-Domänen und Polycomb-Körpern. Die Bewegung von Polycomb-Körpern reagiert weniger empfindlich auf die Temperatur während der Embryogenese, während die Bewegung von Chromatin-Domänen während der gesamten Embryogenese von der Temperatur abhängt.[6]
Als Gegenspieler zu den Polycomb-Körpern können die Transkriptionsfabriken angesehen werden. Diese binden sich an aktives Chromatin und akkumulieren an einer Stelle Transkriptionsfaktoren, damit die Transkription effizient ablaufen kann. Es gibt mehrere Modelle, nach denen Polycomb-Zielgene zwischen Polycomb-Körpern pendeln, wenn sie unterdrückt werden, und zu Transkriptionsfabriken, wenn sie transkriptionell aktiv sind.[12]
Funktionen
Hierarchische Organisation von Entwicklungsgenen
Einzelne Chromosomen decken eine bestimmte Region innerhalb des Kerns ab, die Chromosomenterritorien. Mit zunehmender Auflösung bestehen Chromosomen aus topologisch assoziierten Domänen (TADs). Dies konnte durch die Chromosome Confirmation Capture-Technik bestimmt werden, da bestimmte Abschnitte von Chromosomen untereinander häufiger miteinander reagieren als mit weiter entfernten. Diese TADs sind scharf voneinander an Bindungsstellen von Isolatoren wie zum Beispiel dem CTCF-Protein abgegrenzt. Diese TADs unterscheiden sich anhand unterschiedlicher Arten von Histonmodifikationen und der Chromatinzugänglichkeit. TADs, in denen die Transkription aktiv durchgeführt wird, enthalten entsprechende Histon-Markierungen. Durch diese Gruppierung des Chromatins können ganze Gen-Komplexe aktiv oder inaktiv geschaltet werden. So werden zum Beispiel nach der Differenzierung von embryonalen Stammzellen die gesamte TAD-Struktur und die Lage der TAD-Grenzen nicht verändert. Es treten nur kleine Umlagerungen auf, die mit einer Umverteilung der Markierungen der Histone korrelieren, und so Teile des Chromatin aktivieren und stilllegen. Die PRE-Regionen, an denen die Polycomb-Proteine gebunden sind, bilden mit Hilfe des Polycomb-Körper solche TADs. Das Chromatin von aktiven TADs wird mit durch H3K4me3, inaktive mit dem Histon H3K27me3 markiert. Dieses hierarchische Netzwerk, das von der Rekrutierung von PcG-Proteinen bis zum Gen-Silencing führt, ist weithin akzeptiert.[12][11] Mit Pflanzen wurden Conformation Capture Studien durchgeführt, so ist im Modellorganismus Arabidopsis das Vorhandensein von TADs jedoch noch nicht eindeutig nachgewiesen, die Ergebnisse hängen noch von der Auflösung der verwendeten Hi-C-Methoden ab.[5]
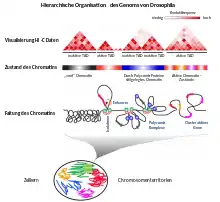
Diese hierarchische Organisation des Genoms wird rechts dargestellt anhand einer vereinfachten Darstellung der Analyse des Drosophila-Genoms:
- Visualisierung der HI-C Daten: Diese Grafik zeigt die Daten, die mit der Chromosome Confirmation Capture Analyse ermittelt wurden. Abschnitte der DNA, die häufiger miteinander reagieren, werden hier in dunkelrot dargestellt. Das Genom konnte so in aktive und nicht-aktive Abschnitte zerlegt werden, sogenannte topologisch assoziierte Domänen.
- Diese Domänen korrespondieren mit den unterschiedlichen Zuständen des Chromatins. "void"-Chromatin sind stillgelegte DNA-Bereiche, die nicht mehr abgelesen werden (Heterochromatin). Die durch Polycomb-Proteine stillgelegten DNA-Bereiche sind ebenfalls sichtbar als inaktive TADs. Diese Stilllegung kann jedoch wieder aufgehoben werden.
- Faltung des Chromatin: Das Heterochromatin wird so stark kondensiert, dass es nicht mehr abgelesen werden kann. Bei der Stilllegung durch Polycomb-Proteine bildet sich ebenso ein inaktive Domäne aus, so dass die darin enthaltenen Gene nicht zugänglich sind. Bei aktiven TADs bilden sich Schleifen aus, die durch Enzyme wie die RNA-Polymerase leicht ablesbar sind. Zudem sind hier die Regulationsabschnitte wie Enhancer leicht zugänglich, die die Transkriptionsaktivität des Gens verstärken können. Die TADs werden durch Bindungsstellen für Isolatoren begrenzt.
- Jedes Chromosom nimmt sein eigenes Volumen im Zellkern ein (Chromosomenterritorium, hier schematisch in verschiedenen Farben dargestellt); diese Volumina überschneiden sich jedoch teilweise und ermöglichen interchromosomale Wechselwirkungen.
Wie wird sichergestellt, dass die Repression auch bei Zellteilung aufrechterhalten wird? Während der DNA-Replikation werden durch posttranslationale Modifikation die elterlichen Histone wieder auf die entstehende DNA aufgebracht. Neu synthetisierte Histone liefern den zusätzlichen Bedarf an Nukleosomen an der entstehenden DNA. Durch diese Koexistenz von elterlichen und neuen Histonen könnten die Markierungen auf elterlichen Histonen als Blaupause für die Modifikation von neuen Histonen in der Nähe dienen. Das Histon H3K27me3 könnte so als epigenetisches Merkmal betrachtet werden, das an einem bestimmten Ort über Zellteilungen hinweg stabil gehalten wird. Jedoch zeigen detaillierte Analysen, dass eine genaue Ausbreitung von H3K27me3 eine kontinuierliche Modifikation neuer Histone sowie bisher unveränderter elterlicher Histone über mehrere Zellgenerationen erfordert. Weitere Forschungen zeigten, dass es für die Markierung der Histone nicht erforderlich ist, dass H3K27me3 an einer bestimmten Position lokalisiert ist. Es reicht aus, dass über ein gewisses Gebiet verteilte Markierungen ein Schwellenniveau erreichen, um den epigenetischen Zustand aufrechtzuerhalten.[11]
Knotenpunkte für die Genunterdrückung
Die Untersuchung der Lokalisation von Polycomb-Proteinen hat ergeben, dass diese in Polycomb-Körper organisiert werden, die oft in der Nähe von pericentromerischem Heterochromatin lokalisiert sind.[16] Mit Hilfe von hochauflösender Fluoreszenz-in-situ-Hybridisierung (FISH) und Immunfärbung von PcG-Proteinen zeigte sich die Kolokalisation von PcG-Zielgenen in den Polycomb-Körpern nur dann, wenn die Gene stillgelegt wurden. In diesem Zusammenhang wurden Polycomb-Körper als Gen-Silencing-Fabriken bezeichnet.[12] In Drosophila melanogaster legen Polycomb-Proteine Hox-Gene durch Bindung an cis-regulierende DNA-Abschnitte still, die sogenannten Polycomb Response Elements (PREs). Wie Polycomb-Körper in Säugersystemen genau funktionieren, muss noch bestimmt werden.[16]
Sumoylierungszentrum
Das SUMO-Molekül ist ein Ubiquitin-ähnliches Protein, das kovalent mit einer Vielzahl von Proteinsubstraten bindet und die Eigenschaften der modifizierten Proteine verändert. Die SUMO-Bindung ist für die Lebensfähigkeit von Zellen und Organismen von der Hefe bis zu Säugetieren unerlässlich. Sie beeinflusst viele biologische Prozesse, einschließlich der Progression des Zellzyklus, der Aufrechterhaltung der Genomintegrität und der Transkription.[17] Es wurde nachgewiesen, dass das menschliche PcG-Protein Pc2 als SUMO E3-Ligase wirkt, indem es SUMO E2 (Ubc9) und die Substrate (CtBP und CTCF) zusammenbringt. So könnten die Polycomb-Körper Sumoylierungszentren bilden. In Caenorhabditis elegans ist das PcG-Protein SOP-2 sumoyliert und enthält RNA-bindende Motive, die in der Lage sind, kleine RNAs zu binden. Diese sind evolutionär in Wirbeltier-PcG-Proteinen konserviert.[16] Es wurde auch gezeigt, dass das Zinkfingerprotein CTCF für Polycomb-Körper rekrutiert und von SUMO modifiziert wird.[18]
Antwort auf Stresssituationen
Die durch Polycomb-Proteine vermittelte Repression könnte auch einen wichtigen Teil in der Antwort auf Stresssituationen spielen. Wird die Zelle einem Hitzeschock ausgesetzt, so führt dies zu einer weitläufigen Repression der Gene. Dies wird durch Polycomb-Proteine vermittelt. Das Genom wird dann substantiell neu organisiert. Zudem ändert sich die Verteilung der Polycomb-Körper: Normalerweise befinden sie sich nicht im Nucleolus. Nach einem Hitzeschock lassen sie sich im gesamten Zellkern inklusive des Nucleolus finden.[19]
Langstrecken-Paarung von Genen
Polycomb-Körper vermitteln Kontakte zwischen weit auseinander liegenden Genen. Wie diese Langstreckenpaarung durchgeführt wird, bleibt weitgehend unbekannt. Es wurde vorgeschlagen, dass PRE-haltige PcG-Zielgene dynamisch an Polycomb-Körpern lokalisieren, so dass Gene, die in einem Polycomb-Körper lokalisiert sind, nur für eine bestimmte Zeit verweilen und dann in einen anderen Polycomb-Körper integriert werden können.[16]
Dieser Prozess des "Hoppings" zwischen Polycomb-Körpern könnte verhindern, dass PcG-Zielgene zufällig im Karyoplasma diffundieren, ermöglicht es diesen Genen aber gleichzeitig, Teile des Kerns zu erforschen und über Polycomb-Körper in der Nähe anderer Gene zu bleiben. Sobald sich die Gene angenähert haben, könnte eine starke Assoziation durch regulatorische Komponenten hergestellt werden.[20]
Vernalisation
Die Vernalisation, die Anregung der Blüte durch eine längere Kälteperiode, beinhaltet eine Polycomb-vermittelte epigenetische Stilllegung des FLOWERING-LOCUS-C-Gens (FLC-Gen) in Arabidopsis. Anhaltende Kälte fördert hier die Umschaltung in einen stillgelegten Zustand.
Allele des FLC-Gens gruppieren sich während der Kälte zusammen. Dies bleibt im Allgemeinen auch nach der Rückkehr der Pflanzen in eine warme Umgebung erhalten. Diese Clusterbildung ist abhängig von den Polycomb-Faktoren, die für die Stilllegung der FLC-Gene notwendig sind. Es wurde eine genau definierte Abfolge von Ereignissen beschrieben, die den Einfluss des PRC2-Proteins und einer weiteren Reihe von Pflanzen-Homöodomänen-Proteinen (PHD) umfasst. Dies führt zu einer fortschreitenden Akkumulation von H3K27me3-Proteinen an der Nukleationsstelle bei zunehmender Einwirkung von Kälte. Eine kälteinduzierte nicht-kodierende RNA namens COLDAIR unterstützt hier die Rekrutierung von PRC2.
Nachdem die Pflanzen wieder in eine warme Umgebung versetzt wurden, breiteten sich PHD-PRC2-Komplexe über das gesamte Gen aus und verursachen eine erhöhte H3K27me3-Ansammlung über den gesamten Locus. Die Menge an H3K27me3 spiegelt quantitativ die Dauer der Kälteexposition wider.[21]
Weblinks
Einzelnachweise
- Bernd Schuettengruber, Henri-Marc Bourbon, Luciano Di Croce, Giacomo Cavalli: Genome Regulation by Polycomb and Trithorax: 70 Years and Counting. In: Cell. Band 171, Nr. 1, September 2017, S. 34–57, doi:10.1016/j.cell.2017.08.002 (englisch, elsevier.com [abgerufen am 17. September 2019]).
- Vincenzo Pirrotta: Polycomb Group Proteins. ISBN 0-12-809737-X, S. 61 (eingeschränkte Vorschau in der Google-Buchsuche).
- Stefania Del Prete, Javier Arpón, Kaori Sakai, Philippe Andrey, Valérie Gaudin: Nuclear Architecture and Chromatin Dynamics in Interphase Nuclei of Arabidopsis thaliana. In: Cytogenetic and Genome Research. Band 143, Nr. 1-3, 2014, ISSN 1424-8581, S. 28–50, doi:10.1159/000363724 (englisch, karger.com [abgerufen am 1. März 2020]).
- Stefanie Rosa, Filomena De Lucia, Joshua S. Mylne, Danling Zhu, Nobuko Ohmido: Physical clustering of FLC alleles during Polycomb-mediated epigenetic silencing in vernalization. In: Genes & Development. Band 27, Nr. 17, 1. September 2013, ISSN 0890-9369, S. 1845–1850, doi:10.1101/gad.221713.113, PMID 24013499, PMC 3778238 (freier Volltext) – (englisch).
- Stefania del Prete, Pawel Mikulski, Daniel Schubert, Valérie Gaudin: One, Two, Three: Polycomb Proteins Hit All Dimensions of Gene Regulation. In: Genes. Band 6, Nr. 3, 10. Juli 2015, ISSN 2073-4425, S. 520–542, doi:10.3390/genes6030520, PMID 26184319, PMC 4584315 (freier Volltext) – (englisch, mdpi.com [abgerufen am 17. September 2019]).
- Thierry Cheutin, Giacomo Cavalli: Progressive Polycomb Assembly on H3K27me3 Compartments Generates Polycomb Bodies with Developmentally Regulated Motion. In: PLoS Genetics. Band 8, Nr. 1, 19. Januar 2012, ISSN 1553-7404, S. e1002465, doi:10.1371/journal.pgen.1002465, PMID 22275876, PMC 3262012 (freier Volltext) – (englisch, plos.org [abgerufen am 17. September 2019]).
- M. Carmo-Fonseca, M. T. Berciano, M. Lafarga: Orphan Nuclear Bodies. In: Cold Spring Harbor Perspectives in Biology. Band 2, Nr. 9, 1. September 2010, ISSN 1943-0264, S. a000703–a000703, doi:10.1101/cshperspect.a000703, PMID 20610547, PMC 2926751 (freier Volltext) – (englisch, cshlp.org [abgerufen am 17. September 2019]).
- Valentina Casa, Davide Gabellini: A repetitive elements perspective in Polycomb epigenetics. In: Frontiers in Genetics. Band 3, 2012, ISSN 1664-8021, doi:10.3389/fgene.2012.00199, PMID 23060903, PMC 3465993 (freier Volltext) – (englisch, frontiersin.org [abgerufen am 17. September 2019]).
- Hua-Bing Li, Katsuhito Ohno, Hongxing Gui, Vincenzo Pirrotta: Insulators Target Active Genes to Transcription Factories and Polycomb-Repressed Genes to Polycomb Bodies. In: PLoS Genetics. Band 9, Nr. 4, 18. April 2013, ISSN 1553-7404, S. e1003436, doi:10.1371/journal.pgen.1003436, PMID 23637616, PMC 3630138 (freier Volltext) – (englisch, plos.org [abgerufen am 17. September 2019]).
- Vincenzo Pirrotta, Hua-Bing Li: A view of nuclear Polycomb bodies. In: Current Opinion in Genetics & Development. Band 22, Nr. 2, April 2012, S. 101–109, doi:10.1016/j.gde.2011.11.004, PMID 22178420, PMC 3329586 (freier Volltext) – (englisch, elsevier.com [abgerufen am 17. September 2019]).
- Sergi Aranda, Gloria Mas, Luciano Di Croce: Regulation of gene transcription by Polycomb proteins. In: Science Advances. Band 1, Nr. 11, Dezember 2015, ISSN 2375-2548, S. e1500737, doi:10.1126/sciadv.1500737, PMID 26665172, PMC 4672759 (freier Volltext) – (englisch, sciencemag.org [abgerufen am 17. September 2019]).
- J. ŠMIGOVÁ, P. JUDA, J. KREJČÍ, I. RAŠKA: Structural Basis of Polycomb Bodies. (PDF) 27. Juni 2014, abgerufen am 17. September 2019 (englisch).
- Moritz Bauer, Johanna Trupke, Leonie Ringrose: The quest for mammalian Polycomb response elements: are we there yet? In: Chromosoma. Band 125, Nr. 3, Juni 2016, ISSN 0009-5915, S. 471–496, doi:10.1007/s00412-015-0539-4, PMID 26453572, PMC 4901126 (freier Volltext) – (englisch, springer.com [abgerufen am 17. September 2019]).
- Sandip De, Apratim Mitra, Yuzhong Cheng, Karl Pfeifer, Judith A. Kassis: Formation of a Polycomb-Domain in the Absence of Strong Polycomb Response Elements. In: PLOS Genetics. Band 12, Nr. 7, 28. Juli 2016, ISSN 1553-7404, S. e1006200, doi:10.1371/journal.pgen.1006200, PMID 27466807, PMC 4965088 (freier Volltext) – (englisch, plos.org [abgerufen am 17. September 2019]).
- Alaguraj Veluchamy, Teddy Jégu, Federico Ariel, David Latrasse, Kiruthiga Gayathri Mariappan: LHP1 Regulates H3K27me3 Spreading and Shapes the Three-Dimensional Conformation of the Arabidopsis Genome. In: PLoS ONE. Band 11, Nr. 7, 13. Juli 2016, ISSN 1932-6203, doi:10.1371/journal.pone.0158936, PMID 27410265, PMC 4943711 (freier Volltext) – (englisch).
- Yuntao S. Mao, Bin Zhang, David L. Spector: Biogenesis and Function of Nuclear Bodies. In: Trends in genetics (TIG). Band 27, Nr. 8, August 2011, ISSN 0168-9525, S. 295–306, doi:10.1016/j.tig.2011.05.006, PMID 21680045, PMC 3144265 (freier Volltext) – (englisch).
- M. Carmo-Fonseca, M. T. Berciano, M. Lafarga: Orphan Nuclear Bodies. In: Cold Spring Harbor Perspectives in Biology. Band 2, Nr. 9, 1. September 2010, ISSN 1943-0264, S. a000703–a000703, doi:10.1101/cshperspect.a000703, PMID 20610547, PMC 2926751 (freier Volltext) – (englisch, cshlp.org [abgerufen am 20. September 2019]).
- M. J. MacPherson, L. G. Beatty, W. Zhou, M. Du, P. D. Sadowski: The CTCF Insulator Protein Is Posttranslationally Modified by SUMO. In: Molecular and Cellular Biology. Band 29, Nr. 3, 1. Februar 2009, ISSN 0270-7306, S. 714–725, doi:10.1128/MCB.00825-08, PMID 19029252, PMC 2630690 (freier Volltext) – (englisch, asm.org [abgerufen am 20. September 2019]).
- Christophe Lavelle, Jean-Marc Victor: Nuclear architecture and dynamics. Elsevier, London 2018, ISBN 978-0-12-803503-0 (englisch).
- F. Bantignies: Inheritance of Polycomb-dependent chromosomal interactions in Drosophila. In: Genes & Development. Band 17, Nr. 19, 1. Oktober 2003, ISSN 0890-9369, S. 2406–2420, doi:10.1101/gad.269503, PMID 14522946, PMC 218078 (freier Volltext) – (englisch, genesdev.org [abgerufen am 20. September 2019]).
- Stefanie Rosa, Filomena De Lucia, Joshua S. Mylne, Danling Zhu, Nobuko Ohmido: Physical clustering of FLC alleles during Polycomb-mediated epigenetic silencing in vernalization. In: Genes & Development. Band 27, Nr. 17, 1. September 2013, ISSN 0890-9369, S. 1845–1850, doi:10.1101/gad.221713.113, PMID 24013499, PMC 3778238 (freier Volltext) – (englisch).