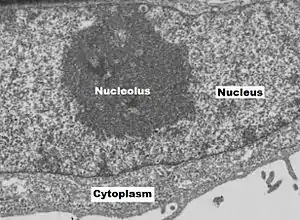
Nucleolus
Ein Nukleolus oder Nucleolus (von lateinisch nucleolus ‚Kernchen‘; Plural: nucleoli), auch Kernkörperchen genannt, ist ein lichtmikroskopisch auffälliger, etwa kugelförmiger Bereich des Nukleoplasmas innerhalb des Zellkerns (Nukleus) einer (eukaryotischen) Zelle. Nukleoli gehen aus bestimmten Chromosomenregionen, den Nukleolusorganisatorregionen (NOR), hervor nach einer Kernteilung (Mitose), sind typischerweise in der Interphase zu finden und dienen dem Aufbau ribosomaler RNA sowie deren Zusammenbau mit ribosomalen Proteinen zu den Vorstufen von Ribosomen. Ribosomen ermöglichen die Proteinbiosynthese im Cytoplasma.
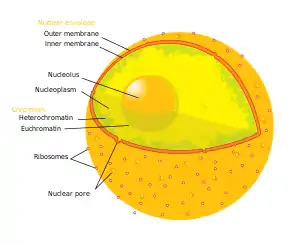
| Übergeordnet |
| Zellkern Karyoplasma |
| Untergeordnet |
| Ribosomale DNA Pars granulosa Pars fibrosa Präribosomen Proteinkomplexe |
| Gene Ontology |
|---|
| QuickGO |
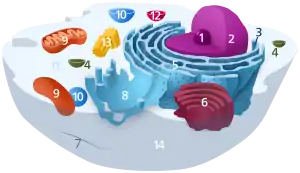
1. Nucleolus (Kernkörperchen)
2. Zellkern (Nukleus)
3. Ribosomen
4. Vesikel
5. Raues (Granuläres) ER (Ergastoplasma)
6. Golgi-Apparat
7. Cytoskelett
8. Glattes (Agranuläres) ER
9. Mitochondrien
10. Lysosom
11. Cytoplasma (mit Cytosol und Cytoskelett)
12. Peroxisomen
13. Zentriolen
14. Zellmembran
In jedem eukaryotischen Genom gibt es mindestens ein Chromosom, das einen Nukleolus entstehen lässt.[1] Liegen innerhalb der Kernhülle mehrere solcher Nukleusorganisatoren vor, so können deren Nukleoli getrennt bleiben oder sich vereinen. Bei menschlichen Zellen verschmelzen die zehn zunächst gebildeten Nukleolen meist zu einem großen Nukleolus im Interphase-Kern. In Zellen mit intensiver Proteinsynthese ist der Nukleolus stets besonders groß.[2]
Jeder Nukleolus enthält entspiralisierte Chromosomenabschnitte mit aneinandergereihten Genen sogenannter ribosomaler DNA (rDNA), an denen mit hoher Genaktivität die Transkription stattfindet. Mit Hilfe der RNA-Polymerase I wird dabei jeweils ein RNA-Einzelstrang (45S-prä-ribosomale RNA) aufgebaut. Dieser wird anschließend so zerlegt, dass drei verschiedene Moleküle ribosomaler RNA (rRNA) entstehen: 18S- sowie 5,8S- und 28S-rDNA. Mit 5S-rRNA sind diese die funktionell grundlegenden Bausteine von Ribosomen. Sie bilden zusammen mit einer Reihe unterschiedlicher ribosomaler Proteine, die zum Nukleolus transportiert werden, die Vorstufen kleiner (40S-) beziehungsweise großer (60S-) Untereinheiten eukaryotischer Ribosomen.
Die Vorgänge der Transkription von rDNA, der Prozessierung zu rRNA-Molekülen und deren Assoziation mit spezifischen Proteinen zu komplexen Ribonukleoproteinen laufen im Nukleolus in aufeinanderfolgenden Zonen ab und prägen so dessen Struktur. Hauptsächliche Aufgabe eines Nukleolus ist diese Biogenese präribosomaler Partikel, die schließlich durch die Kernporen transportiert im Cytosol die Proteinbiosynthese ermöglichen, als freie Ribosomen oder membrangebunden am (rauen) Endoplasmatischen Retikulum.
Lage und Vorkommen
Humangenetik
Im menschlichen Genom liegen die rDNA-Gene für rRNA jeweils auf den kurzen (p-)Armen der akrozentrischen Chromosomen an den Genorten 13p12, 14p12, 15p12 sowie 21p12 und 22p12. Die repetitiven DNA-Sequenzen der entsprechenden Nuklolusorganisatoren dieser Regionen weisen ieweils ungefähr etwa 50 Sequenzwiederholungen pro NOR auf. In einem diploiden menschlichen Zellkern liegen also insgesamt zehn Chromosomen mit Nukleolus-Organisator-Regionen vor. Die in der Telophase zunächst gebildeten bis zu zehn kleinen Nukleolen[3] vereinigen sich im Laufe der Interphase in vielen Zellkernen nicht selten zu einem großen, gemeinsamen Nukleolus. Dabei ist kaum zu entscheiden, ob noch alle NOR gleichermaßen aktiv sind.
Die DNA-Sequenzen für die 5S-rRNA liegen beim Menschen im langen (q-)Arm des Chromosoms 1. Auch bei anderen Spezies befinden sich die Gene für die 5S-rRNA außerhalb von Nukleolusorganisatorregionen.
Allgemeine Genetik

Anders als etwa bei Pflanzen oder Wirbeltieren mit ihren kleinen Zellkernen der Interphase sind Nukleolen verhältnismäßig leicht an Polytänchromosomen zu beobachten, wie sie in Fliegen-Maden und Mücken-Larven vorkommen. Diese Insekten entwickeln in Speicheldrüsen und in Malpighi-Gefäßen riesige Zellkerne. In diesen ist eindeutig zu erkennen, welches der entsprechend großen Chromosomen eine NOR besitzt. Als Beispiel dient hier die Abbildung des polytänen Chromosompaares 4 aus einer larvalen Speicheldrüse der Zuckmücke Chironomus halophilus. Am linken Ende sind die elterlichen Chromatidenbündel getrennt und tragen unterschiedlich große Nukleolen, dunkel gefärbt mit Uranylacetat. Als Diagnose bietet sich an: Die beiden elterlichen NOR sind unterschiedlich aktiv, sie zeigen heterozygote Transkription. Obwohl sie nahe beieinander liegen, sind die beiden Nukleolen voneinander getrennt.
Chromosomen mit Nukleolusorganisator (NOR)
Die folgende Tabelle zeigt, dass die Zahl nuklelolusorganisierender Regionen von Art zu Art verschieden sein kann.
| Spezies | Karyotyp | Chromosom mit NOR | Autoren |
|---|---|---|---|
| Zea mays | 2n =20 | #6 | Silva et al, 2018.[4] McClintock 1929.[5] |
| Drosophila melanogaster | 2n = 8,XY | X bzw. Y | Warsinger-Pepe et al, 2020.[6] |
| Chironomus commutatus | 2n = 6 | FEG-Element | Keyl 1960. Tafel 8.[7] |
| Chironomus halophilus | 2n = 8 | #4 | Keyl & Keyl 1959. Tafel 9.[8] |
| Chironomus obtusidens | 2n = 8 | G | Beermann 1962.[9] |
| Chironomus plumosus | 2n = 8 | #4 | Keyl & Keyl 1959. Tafel 10. |
| Chironomus tentans | 2n = 8 | #1, #2 | Pelling 1964.[10] |
| Pseudodiamesa branickii | 2n = 8 | #4 | Zacharias 1984.[11] |
| Sus scrofa Hausschwein | 2n = 38,XY | #8, #10 | Mellink et al 1994.[12]
Rønne et al 1987.[13] |
| Bos taurus Hausrind | 2n = 62,XY | 2q, 3q, 4q, 11q, 25q | Andraszek & Smalec 2012.[14] |
| Homo sapiens | 2n = 46,XY | 13p, 14p, 15p, 21p, 22p | Mangan & McStay 2021.[15]
Van Sluisa et al 2020.[16] |
Für Chironomus commutatus ist aus seinem polytänen Chromosomensatz ein für diese Gattung ungewöhnlicher Karyotyp 2n = 6 abzuleiten. Der Nukleolus ist an ein langes Chromosom gebunden, das durch Translokation aus den chromosomalen Untereinheiten F+E+G zusammengesetzt ist. Die NOR liegt im Teil G, der ehemals – wie bei C. obtusidens – ein selbständiges Chromosom war.
Um den Karyotyp zu formulieren, darf man sich nicht immer auf den polytänen Satz berufen. Davor warnt die Zuckmücke Pseudodiamesa branickii: Auf den ersten Blick besitzt sie drei polytäne Chromosomenpaare. Doch keinem ist ein Nukleolus zuzuordnen. Tatsächlich zeigen mitotische Metaphasen 2n = 8 Chromosomen. Mit verschiedenen Methoden wurde die multiplizierte NOR in Riesenkernen als unabhängiges Chromatinnetz neben den Polytänchromosomen nachgewiesen. Die Transkription in der NOR ist so stark, dass die multiplizierten Chromatiden der beiden kleinen Chromosomen 4 nicht zusammenbleiben, sich nicht bündeln konnten. Diese Besonderheit bestätigt die NOR als den mächtigsten unter den aktiven Genorten, die in polytänen Elementen als Puffs (pʌfs; engl.) charakterisiert sind.
Anzahl der Transkriptionseinheiten für rRNA
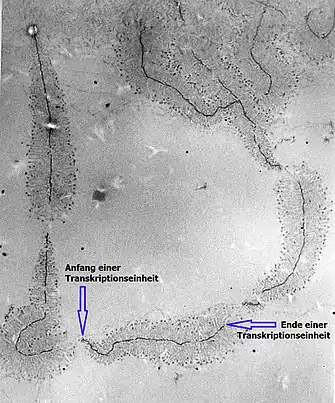
Auf der ribosomalen DNA (rDNA) von nukleolusorganisierenden Regionen wiederholen sich identische DNA-Sequenzen mehrfach bis tausendfach im Genom. Diese repetitiven Sequenzen können als verschiedene Transkriptionseinheiten für den Aufbau von rRNA gleichzeitig abgelesen werden. Bei dem Transkriptionsvorgang mittels RNA-Polymerase I wird jeweils als erstes Genprodukt ein längerer RNA-Einzelstrang aufgebaut, die sogenannte 45S-prä-rRNA. Diese wird anschließend verändert und so in mehrere Teile zerlegt, dass daraus je ein 18S-, 5,8S- und 28S-rRNA-Molekül hervorgehen, die Grundbausteine späterer Ribosomen. Die Sedimentationszahl S ist ein Maß für die Größe der RNA-Moleküle unter Beschleunigung in einer Ultrazentrifuge. Die folgende Tabelle führt exemplarisch – inzwischen überholte – Schätzungen der Gesamtzahl an Transkriptionseinheiten für 45S-prä-rRNA in verschiedenen einfachen (1n, 1c) Genomen auf:[17][18]
| Spezies | deutscher Name | rRNA-Gene |
|---|---|---|
| Saccharomyces cerevisiae | Backhefe | 140 |
| Allium cepa | Zwiebel | 6950 |
| Pisum sativum | Erbse | 3900 |
| Triticum aestivum | Weizen | 6350 |
| Bombyx mori | Seidenspinner | 240 |
| Drosophila melanogaster | Taufliege | 200 |
| Salmo salar | Atlantischer Lachs | 710 |
| Gallus domesticus | Haushuhn | 200 |
| Mus musculus | Hausmaus | 100 |
| Homo sapiens | Mensch | 200 |
Gene für 5S-rRNA
Nicht in der rDNA der Nukleolusorganisatoren sind die repetitiven DNA-Sequenzen von Genen für die 5S-rRNA zu finden. Im menschlichen Genom liegen diese auf dem langen (q)-Arm des großen Chromosoms 1. Die Mehrzahl dieser Transkriptionseinheiten wurde in der Region 1q42.11–13 nachgewiesen,[20][21] etwa ein Viertel der Gene liegen in der Region 1q31.[22]
Bei der Taufliege (Drosophila melanogaster) enthält das Chromosom 2 in Region 56EF die etwa 200 Kopien des Gens für 5S-rRNA. Getrennt davon enthält das X-Chromosom – wie in der obenstehenden Tabelle gelistet – die Region des Nucleolusorganisators mit den Genen für 18S- und 28S-rRNA.[23] Ähnlich ist das Genom der Zuckmücke Chironomus thummi organisiert. Die DNA-Sequenzen für die 5S-rRNA wurden im rechten Arm des polytänen Chromosoms 2 aufgespürt, und zwar in der Region B3c–e.[24] (Auch bei dieser Art gehört der Nukleolus zum kleinen Chromosom 4.)
Proteine für die rRNAs
Die Gene für alle Proteine liegen in den Chromosomen. Von den Genen werden die entsprechenden mRNAs als Informationsboten abgeschrieben, die dann durch die Kernporen ins Zytoplasma gelangen. An den Ribosomen erfolgt, entsprechend der genetischen Information einer mRNA, die Biosynthese eines Proteins. Im Falle der Polymerasen für rRNA werden diese durch die Kernporen zurück in den Zellkern befördert, um an den für sie bestimmten Genorten von der rDNA die rRNA abzuschreiben.
Zwei Polymerasen für die rRNAs
Die rDNA in einer NOR wird von der RNA-Polymerase I abgelesen. Das DNA-abhängige Enzym synthetisiert die 45S-prä-rRNA. Dieses Vorprodukt wird im Nukleolus in mehreren Schritten zu den rRNAs 28S, 18S und 5.8S prozessiert.[25][26]
Die Genorte für die 5S-rRNAs liegen jedoch außerhalb der NORs. Die RNA-Polymerase III besorgt die Transkription der 5S-rRNA, welche vom ursprünglichen Genort abwandert und in den Nukleolus gelangt.[27]
Prozessproteine für die rRNAs
Das nukleoläre prä-rRNA-prozessierende Protein NIP7 sei als Beispiel vorgestellt. Es ist nötig, um für die Biosynthese der kleinen Ribosomen-Untereinheiten reife 18S-rRNA herzustellen. Das Adjektiv im Namen betont, dass der Wirkbereich des NIP7 auf den Nukleolus beschränkt bleibt.[28] Das Gen für das NIP7 liegt im langen Arm des menschlichen Chromosoms 16, und zwar in 16q22.1.[29]
Zwei Ribosomen-Bauteile
Die ribosomalen Proteine werden aus dem Zytosol durch die Kernporen zum Nukleolus geschleust und dort verwertet. Zusammen mit diesen Proteinen bilden die rRNAs 28S, 5,8S und 5S die großen Untereinheiten der künftigen Ribosomen. Die 18S-RNA ist das Molekül für die kleine Untereinheit der künftigen Ribosomen. Die großen (60S) und die kleinen (40S) Ribosomen-Untereinheiten werden anschließend wieder ins Zytosol gebracht und gehen dort, nach ihrem Zusammenschluss zu 80S-Ribosomen, am rauen Endoplasmatischen Reticulum (ER) oder frei im Zytosol, ihrer Tätigkeit als Translationseinheiten nach (siehe Ribosom).
Funktion des Nukleolus
Als Produktionsort der Ribosomen-Untereinheiten ist ein Nukleolus in der Interphase des Zellzyklus aktiv. In dieser Zeit ist das „Kernkörperchen“ mit einem Lichtmikroskop zu beobachten. Das ist naheliegend, weil die Interphase die Hauptzeit des zellulären Stoffwechsels ist, gekennzeichnet von Proteinsynthese, die massenhaft Ribosomen erfordert. In Zellen mit intensiver Proteinsynthese ist der Nukleolus besonders groß.[30] Allerdings sind die Bildungsorte der Nukleolen während der mitotischen Interphase nicht ohne weiteres zu erkennen, weil die Chromosomen derweil völlig aufgelockert sind. Organe mit Polytänchromosomen bieten diesbezüglich einen diagnostischen Vorteil.
Bis zum Begin der Kernteilung werden die Nukleolen meist aufgebraucht: Ihre Struktur verschwindet und ist folglich auch nicht mehr anfärbbar. Die Nukleolen sind in der Mitose inaktiv, weil die Zellen zur Metaphase und Anaphase (fast) keine Proteine herstellen und deswegen vom Zellkern keine neue genetische Information brauchen.[31] Sollte während der Interphase eine NOR besonders aktiv gewesen sein, mag sie in manchen Metaphasen nicht so stark kondensieren wie das übrige Nukleolus-Chromosom. Diese Chromosomen-Stelle geringer Verpackung wird als sekundäre Konstriktion bezeichnet. (Als primäre Konstriktion gilt die Zentromer-Region.) Nach der Telophase nehmen die NORs der Tochterkerne die Transkription auf und bilden neue Nukleolen.
Keine Lehrbuchregel ohne Ausnahme. In manchen Arten bleiben die Nukleolen während der Mitose erhalten und teilen sich in der Anaphase. Für dieses Ausnahmephänomen steht das Stichwort „Nukleolenpersistenz“; ein Beispiel bieten die Wurzelspitzen der Gewöhnlichen Sonnenblume, Helianthus annuus.[32]
Struktur
Im Kernkörperchen selbst lassen sich mit einem Elektronenmikroskop Pars fibrosa und Pars granulosa unterscheiden. Die Pars fibrosa kann weiterhin in fibrilläre und dicht fibrilläre Komponenten untergliedert werden:
- Die fibrillären Komponenten werden als mehrere rundlich, aufgehellte Bereiche im Nucleolus erkennbar. Hier findet durch die RNA-Polymerase I die Transkription von 45S-Prä-rRNA statt.
- Die dicht fibrillären Komponenten liegen meist schalenförmig als dunkle Anteile um diese Transkriptionsbereiche angeordnet. Hier finden Zerschneidungen der 45S-rRNA in 28S-, 18S- und 5,8S-rRNA statt.
Die Pars granulosa stellt den größten Teil des Nucleolus dar. Ihr gekörntes Erscheinungsbild wird hauptsächlich durch die Synthese präribosomaler Partikel geprägt. Diese bestehen aus den verschiedenen rRNA-Formen und assoziierenden Proteinen, wobei der großen ribosomalen Untereinheit 49, der kleinen 33 zusätzliche Proteine zugeordnet werden.
Färbbarkeit
- Für die Nukleolen-Diagnose mit dem Lichtmikroskop mischt man einen Teil 1 % Ameisensäure und 2 % Gelatine mit zwei Teilen 50 % Silbernitrat. In dem dunkelgestellten Gemisch bleiben die Zellpräparate 30 Minuten, bevor sie zusätzlich die May-Grünwald-Färbung erhalten.[33]
- Bevorzugt ist der Nukleolus mit Uranylacetat darzustellen.[34] Siehe polytänes Chromosom 4 in Chironomus halophilus.
- Ein Fluoreszenzmikroskop erlaubt, in Zellkernen mit einem anti-rA.dT-IgG die molekularen Hybride von rRNA.rDNA in der NOR nachzuweisen.[35]
Geschichte
Erstes Interesse für die Nucleoli erregten John Gurdon und Donald Brown 1964, die beim Krallenfrosch Xenopus laevis entdeckten, dass 25 % von dessen Eiern keinen Nucleolus besaßen und allesamt nicht lebensfähig waren. Die Hälfte der Eier besaßen nur einen Nucleolus und 25 % besaßen zwei. Demnach mussten die Kernkörperchen einen lebensnotwendigen Bereich enthalten. 1966 zeigten Max Birnstiel und Hugh Wallace mit Hilfe von Hybridisierungsexperimenten, dass sie ribosomale DNA kodieren.
Krankheit und Nukleolen
Der Nukleolus reguliert nicht nur die Biogenese der Ribosomen, sondern ist auch für andere Zellfunktionen verantwortlich. Dazu zählen Antworten auf Stress, Alterung und Lebensdauer sowie die Genom-Organisation. Was menschliche Krankheiten anbelangt, spielen Nukleolus-Defekte eine Rolle, so beim vorzeitigen Altern im Hutchinson-Gilford-Syndrom oder bei der Blutarmut des Diamond-Blackfan-Syndroms.[36][37] Nicht zuletzt verdächtigt man den Nukleolus, die Krebsentstehung anzuzeigen oder ursächlich dabei mitzuwirken.[38][39]
Bei der Taufliege kommt eine Mutation bobbed (bb) vor. Den „gestutzten“ Körperbau verursacht ein Mangel an rDNA, der das Wachstum dramatisch verlangsamt und durchaus tödlich sein kann.[40] Von dieser genetischen Mangelkrankheit erholen sich Männchen über wenige Generationen, indem zwischen den Chromosomen X und Y mehrmals Crossing-over eintritt. Solche meiotische Rekombinationen ergeben eine „Magnifikation“, eine Vergrößerung der rDNA bis zum 20-Fachen. In der Modellvorstellung bildet die rDNA Ringe, die in den chromosomalen Genort eingefügt werden.[41][42]
Viren und Nukleolen
Viele Viren, deren Replikation in Zellkernen abläuft, lösen auch Wechselwirkungen mit den Nukleolen aus. Einige RNA-Viren, die im Zytoplasma repliziert werden, verursachen die Produktion gewisser Proteine, die in den Zellkern eindringen und dort den Nukleolus ansteuern.[43] Dadurch werden Nukleolus-Proteine in andere Bereiche der Wirtszelle umverteilt. So kommt es zu schwerwiegenden Auswirkungen auf Transkription und Translation. Die Veränderungen des zellulären Stoffwechsels dienen der Viren-Produktion.[44][45] – „Viren sind gelebtes Leben!“[46]
Literatur
- Konstantin I Panov, Katherine Hannan, Ross D Hannan, Nadine Hein: The ribosomal gene loci: The power behind the throne. In: Genes 12, 2021: 763. PDF.
- Renate Lüllmann-Rauch, Esther Asan: Taschenbuch Histologie. Thieme, Stuttgart, 6. Auflage 2019. ISBN 3-13-242533-8. Ganzes Buch für persönliche Verwendung: 68MB-PDF.
- Attila Németh, Ingrid Grummt: Dynamic regulation of nucleolar architecture. In: Curr Opin Cell Biol 52, 2018: 105–111.
- Jane B Reece, Lisa A Urry, Michael L Cain, Steven A Wasserman, Peter V Minorsky, Robert B Jackson: Campbell Biologie. 2015. Buch-Download 280 MB-PDF.
- Thoru Pederson: The nucleolus. In: Coldspring Harb Perspect Biol 3, 3, 2011: a000638. PDF.
- Németh A, Conesa A, Santoyo-Lopez J, et al.: Initial genomics of the human nucleolus. In: PLoS Genet.. 6, Nr. 3, 2010, S. e1000889. doi:10.1371/journal.pgen.1000889. PMID 20361057. PMC 2845662 (freier Volltext).
- Otto Bucher, Hubert Wartenberg: Cytologie, Histologie und mikroskopische Anatomie des Menschen, 12. vollständig überarbeitete Auflage. Huber, Stuttgart 1997; ISBN 3-456-82785-7.
Einzelnachweise
- Wolfgang Beermann: Der Nukleolus als lebenswichtiger Bestandteil des Zellkerns. In: Chromosoma 11, 1960: 263–296.
- Eintrag Nucleolus im Lexikon der Biologie auf spektrum.de.
- Walther Traut: Chromosomen: Klassische und molekulare Cytogenetik. Springer: Berlin, Heidelberg 1991: 189–193. ISBN 3-540-53319-2. → Seite 191: „Die maximale Zahl der Nukleolen entspricht der Zahl der aktiven NORs eines Zellkerns.“
- Jéssica Coutinho Silva, Carlos Roberto Carvalho, Wellington Ronildo Clarindo: Updating the maize karyotype by chromosome DNA sizing. In: PLoS ONE 13, 1, 2018: e0190428. PDF.
- Barbara McClintock: Chromosome morphology in Zea mays. In: Science 69, 1798, 1929: 629. Kurzmitteilung.
- Natalie Warsinger-Pepe, Duojia Li, Yukiko M Yamashita: Regulation of nucleolar dominance in Drosophila melanogaster. In: Genetics 214, 2020: 991–1004. PDF.
- Hans-G Keyl: Die cytologische Diagnostik der Chironomiden: II. Diagnosen der Geschwisterarten Chironomus acidophilus n. sp. und Ch. uliginosus n. sp. In: Arch Hydrobiol 57, 1960: 187–195. → Tafel 8.
- Hans-G Keyl, Ilse Keyl: Die cytologische Diagnostik der Chironomiden: I. Bestimmungstabelle für die Gattung Chironomus auf Grund der Speicheldrüsen-Chromosomen. In: Arch Hydrobiol 56, 1959: 43–57; Tafel 9.
- Wolfgang Beermann: Riesenchromosomen. Springer, Wien 1962: → S. 21.
- Claus Pelling: Ribonukleinsäure-Synthese der Riesenchromosomen: Autoradiographische Untersuchungen an Chironomus tentans. In: Chromosoma 15, 1964: 71–122.
- Helmut Zacharias: Allocyclic behaviour and underreplication of the nucleolus chromosome in Pseudodiamesa (Chironomidae). In: Chromosoma 89, 1984: 263–273.
- Clemens H Mellink, A A Bosma, N A De Haan: Variation in size of Ag-NORs and fluorescent rDNA in situ hybridization signals in six breeds of domestic pig. In: Hereditas 120, 2, 1994: 141–149.
- Mogens Rønne, Vera Stefanova, Dino di Bernardino, Birger Strandby Poulsen: The R-banded karyotype of the domestic pig (Sus scrofa dornestica L.) . In: Hereditas 106, 1987: 219–231.
- Katarzyna Andraszek, Elżbieta Smalec: Structure of the nucleoli in domestic cattle spermatocytes. In: Folia Histochem Cytobiol 50, 3, 2012: 346–351. PDF.
- Hazel Mangan, Brian McStay: Human nucleoli comprise multiple constrained territories, tethered to individual chromosomes. In: Genes Dev 35, 7–8, 2021: 483–488. PDF.
- Marjolein van Sluisa, Chelly van Vuurena, Hazel Mangana, Brian McStay: NORs on human acrocentric chromosome p-arms are active by default and can associate with nucleoli independently of rDNA. In: Proc Natl Acad Sci USA 117, 19, 2020: 10368–10377. PDF.
- Adrian T Sumner: Chromosomes: Organisation and function. Blackwell, Oxford UK 2003; ISBN 0-632-05407-7. → Tab 11.1, S 134.
- Harris Busch, Lawrence Rothblum (eds): The cell nucleus. Vol X: rDNA, part A. Academic Press, New York 1982.
- Hans-H Trepte: Ultrastructural analysis of Balbiani ring genes of Chironomus pallidivittatus in different states of Balbiani ring activity. In: Chromosoma102, 1993: 433–445.
- Pernille Dissing Sørensen, B Lomholt, Sune Frederiksen, N Tommerup: Fine mapping of human 5S rRNA genes to chromosome 1q42.11-q42.13. In: Cytogenet Cell Genet 57, 1991: 26–29. Abstract.
- Pernille Dissing Sørensen, Sune Frederiksen: Characterization of human 5S rRNA genes. In: Nucleic Acids Res 19, 15. 1991: 4147–4151. PDF.
- B Lomholt, Sune Frederiksen, J Nederby Nielsen, C Hallenberg: Additional assignment of the human 5S rRNA genes to chromosome region 1q31. In: Cytogenetics and Cell Genetics. Band 70, 1995, S. 76–79; Abstract.
- James D Procunier, Kenneth D Tartof: Genetic analysis of the 5S RNA genes in Drosophila melanogaster. In: Genetics. Band 81, Nr. 3, 1975, S. 515–523; PDF.
- Helmut Bäumlein, Ulrich Wobus: Chromosomal localization of ribosomal 5S RNA genes in Chironomus thummi by in situ hybridization of iodinated 5S RNA. In: Chromosoma 57, 1976: 199–204.
- Ingrid Grummt: Regulation of mammalian ribosomal gene transcription by RNA polymerase I. In: Progr Nucleic Acid Res Mol Biol 62, 1999: 109–154.
- Walter Traut: Chromosomen: Klassische und molekulare Cytogenetik. Springer, Berlin, Heidelberg1991. ISBN 3-540-53319-2. → Seite 192, Abb. 8.18: „Tandem-Repeats aktiver ribosomaler Transkriptionseinheiten aus dem Nukleolus einer Oocyte des Molches Pleurodeles“.
- Ian M Willis: RNA polymerase III: Genes, factors and transcriptional specificity. In: Eur J Biochem 212, 1993: 1–11.
- Luis G Morello, Cédric Hesling, Patrícia P Coltri, Beatriz A Castilho, Ruth Rimokh, Nilson I T Zanchin: The NIP7 protein is required for accurate pre-rRNA processing in human cells. In: Nucleic Acids Res 39, 2, 2011: 648–665. PDF.
- Matthew B Gross: Persönliche Mitteilung an OMIM: 2021. PDF.
- Eintrag Nucleolus im Lexikon der Biologie auf spektrum.de.
- Walter Traut: Chromosomen. 1991: S. 189.
- Georg Tischler: Handbuch der Pflanzenanatomie. Band II: Allgemeine Pflanzenkaryologie. 2. Hälfte: Kernteilung und Kernverschmelzung. Naturwissenschaftlicher Verlag, Berlin 1951. → Fig. 113, Seite 201.
- Yu Furusawa, Masashi Takahashi, Mariko Shima-Sawa, Osamu Yamamoto, Akira Yabuki: Argyrophilic nucleolar organizer regions staining for cytology smears in dogs and cats. In: J Vet Med Sci 82, 9, 2020: 1267–1270. PDF.
- Juan C Stockert. Uranyl-EDTA-Hematoxylin: A new selective staining technique for nucleolar material. In: Histochem 43, 1975: 313–322.
- Werner Büsen, J M Amabis, Orillio Leoncini, B D Stollar, F J S Lara: Immunofluorescent characterization of DNA.RNA hybrids on polytene chromosomes of Trichosia pubescens (Diptera, Sciaridae). In: Chromosoma 87, 1982: 247–262.
- Ursula Stochaj, Stephanie C Weber: Nucleolar organization and functions in health and disease. In: Cells 9, 2020: 526. PDDF.
- Kai Yang, Jie Yang, Jing Yi: Nucleolar Stress: Hallmarks, sensing mechanism and diseases. In: Cell Stress 2, 6, 2018: 125–140.PDF.
- Pietro Carotenuto, Annalisa Pecoraro, Gaetano Palma, Giulia Russo, Annapina Russo: Therapeutic approaches targeting nucleolus in cancer. In: Cells 8, 9, 2019: 1090. PDF.
- Davide Ruggero: Revisiting the nucleolus: From marker to dynamic integrator of cancer signaling. In: Sci Signal 5, 241, 2012: pe38. PDF.
- Roberto Weinmann: Regulation of ribosomal RNA and 5S RNA synthesis in Drosophila melanogaster: I. Bobbed mutants. In: Genetics 72, 1972: 267–276.PDF.
- Ferruccio Ritossa: Crossing-over between X and Y chromosomes during ribosomal DNA magnification in Drosophila melanogaster. In: Proc Natl Acad Sci USA 70, 7, 1973: 1950–1954. PDF.
- Lino C Polito, Daniela Cavaliere, Anna Zazo, Maria Furia: A study of rDNA magnification phenomenon in a repair-recombination deficient mutant of Drosophila melanogaster. In: Genetics 102, 1982: 39–48. PDF.
- Stephen M Rawlinson, Gregory W Moseley: The nucleolar interphase of RNA viruses. In: Cell Microbiol 17, 8, 2015: 1108-1120.
- Anna Greco: Involvement of the nucleolus in replication of human viruses. In: Rev Med Virol 19, 2009: 201–214. PDF.
- Julian A Hiscox: The nucleolus – a gateway to viral infection? In: Arch Virol 147, 2002: 1077–1089. PDF.
- Unbekannter Autor.
Weblinks
- Alexander Schlaak: Organisationsprinzip des menschlichen Genoms identifiziert. Universität Regensburg, Pressemitteilung vom 13. April 2010 beim Informationsdienst Wissenschaft (idw-online.de), abgerufen am 15. September 2015.