Hox-Gen
Hox-Gene sind eine Familie von regulativen Genen. Ihre Genprodukte sind Transkriptionsfaktoren, welche die Aktivität anderer, funktionell zusammenhängender Gene im Verlauf der Individualentwicklung (Morphogenese) steuern. Sie gehören also zu den homöotischen Genen.
Der charakteristische Bestandteil eines Hox-Gens ist die Homöobox. Dabei handelt es sich um eine charakteristische Sequenz homöotischer Gene. Die Homöoboxen codieren in den Zellen für abgrenzbare besondere Proteinbereiche oder Proteindomänen (Homöodomänen). Diese bestehen in der Regel aus 60 Aminosäuren und besitzen eine DNA-Bindedomäne. Die Basensequenz der Homöobox ist bei allen Hox-Genen aller Tierarten sehr ähnlich. Dies lässt den Schluss zu, dass sie bereits früh in der Evolutionsgeschichte konserviert worden ist; offensichtlich wirken sich Mutationen hier meist letal aus.

1 eines der beiden Chromatiden (blau)
2 am Centromer (rot) hängen die beiden Chromatiden zusammen – hier setzen in der Mitose die Mikrotubuli an.
3 kurzer Arm (p-Arm)
4 langer Arm (q-Arm)
Aufgaben
Primäre Aufgabe der Hox-Gene ist die Gliederung des Embryos entlang der Körperlängsachse (anatomisch: kranio-kaudale Achse). Diese Aufgabe erfüllen sie bei allen Tieren, die eine Körperachse besitzen. Verschiedene Stämme oder Ordnungen verfügen über eine unterschiedliche Anzahl von Hox-Genen, die bei der Embryonalentwicklung jeweils in einem bestimmten Abschnitt exprimiert werden. Dadurch ergibt sich eine Abfolge von Streifen innerhalb des Embryonalgewebes. Die Zellen dieses Streifens erhalten dadurch eine Lageinformation über ihre Position im sich entwickelnden Embryo; ihre weitere Teilung, ihre Differenzierung und ggf. ihr programmierter Zelltod (Apoptose) erfolgen dann dieser Lage entsprechend. Besonders markant ist die Ausbildung und die spezifische Form von Extremitäten. Bei Insekten bestimmen die Hox-Gene, ob an einem Segment Extremitätenanlagen entstehen. Später regeln sie dann, welche Art von Extremitäten gebildet wird (z. B. Antennen, Mundwerkzeuge, Beine, Flügel). Beim Wirbeltierembryo (auch beim Menschen) bestimmen die Hox-Gene unter anderem die Ausbildung und Form der Wirbel (Halswirbel, Brustwirbel, Lendenwirbel) und Rippen. Die Stammgruppen der Insekten (Arthropoda) und Wirbeltiere (Chordata) sind segmentierte Organismen. Hox-Gene sind aber bei Organismen ohne Segmente in gleicher Weise an der Organisation beteiligt.
Die zweite Körperachse der Bilateria, die Rücken-Bauch-Achse (anatomisch: Dorsal-Ventral-Achse) wird nicht von Hox-Genen gesteuert. Für ihre Festlegung sind andere Transkriptionsfaktoren verantwortlich: decapentaplegic oder bone morphogenetic protein (Dpp/Bmp) und short gastrulation oder chordin (Sog/Chrd).
Evolution
Die Hox-Gene sind nur ein Bestandteil einer größeren Familie von Genen mit jeweils ähnlichen Funktionen. Außer den Hox-Genen existieren die Familien der ParaHox-Gene und der sog. NK-Gene (benannt nach ihren Entdeckern Niremberg und Kim). Mit einigen weiteren, kleineren Genfamilien bilden sie den sogenannten ANTP-Megacluster. Möglicherweise besitzen die jeweiligen Familien besondere Beziehungen zu jeweils einem der embryonalen Keimblätter: Hox zum Neuroektoderm, ParaHox zum Endoderm, NK zum Mesoderm. Wenn sich diese Theorie bestätigen würde, hätte dies tiefgreifende Auswirkungen auf die Rekonstruktion des gemeinsamen Stammbaums der Tierstämme.
Alle hierher gehörigen Gene weisen so große Übereinstimmung in ihrer Basensequenz auch abseits der Homöobox auf, dass man ihre Entstehung durch Genverdoppelung aus einem einzigen Ursprungsgen annimmt. Gene aus dem ANTP-Megacluster wurden in allen daraufhin untersuchten mehrzelligen Tieren gefunden. Sie traten in keinem bisher darauf untersuchten Einzeller (Protozoa) auf, insbesondere auch nicht in den Choanoflagellaten, die allgemein als Schwestergruppe der vielzelligen Tiere gelten, d. h. unter den Einzellern am nächsten mit ihnen verwandt sind. Die Genfamilie muss damit bereits beim gemeinsamen Urahnen aller Metazoa (im Präkambrium) existiert haben. Eigentliche Hox-Gene sind allen Tierstämmen mit Ausnahme der Schwämme, der Rippenquallen[1] (und möglicherweise der Placozoa) gemeinsam. Bei den Nesseltieren (Cnidaria) existieren zwei Hox-Gene. Die primitivsten Bilateria, die Acoelomorpha, besitzen vier. Bei allen höher organisierten Tierstämmen werden die Verhältnisse komplizierter, weil in verschiedenen Linien sich einzelne Hox-Gene aufgespalten (verdoppelt) haben und andere verschwunden sind. Dadurch entsprechen sich die verbleibenden Hox-Gene nicht immer direkt, auch wenn ihre Anzahl möglicherweise gleich ist. Arthropoden und Mollusken weisen zum Beispiel beide jeweils neun Hox-Gene auf.
Am kompliziertesten sind die Verhältnisse bei dem Überstamm der Deuterostomia, zu dem auch die Wirbeltiere gehören. Das Lanzettfischchen Amphioxus, letzter überlebender Vertreter der Schädellosen (Acrania), die als nächste Verwandte des Vorfahren der höheren Wirbeltiere gelten, besitzt fünfzehn Hox-Gene. Die vierfüßigen Wirbeltiere (Tetrapoda) besitzen 39 Hox-Gene, die sich in vier Gencluster einteilen lassen. Heute wird allgemein angenommen, dass sich das Wirbeltiergenom (einschließlich der Hox-Gene) im Verlauf der Evolution zweimal komplett verdoppelt hat. Die heutige Zahl kommt durch den nachträglichen Verlust einzelner Gene zu einem späteren Zeitpunkt zustande. Verschiedene Entwicklungslinien der Knochenfische, bei denen es nach der Abspaltung der Tetrapoda zu weiteren Genverdoppelungen gekommen ist, besitzen noch mehr Hox-Gene.
Die Hox-Gene aller höher organisierten Stämme lassen sich vier Genfamilien zuordnen, die auf die vier Hox-Gene des Urbilateriers zurückgeführt werden. Die einzelnen Gene innerhalb der verschiedenen Stämme lassen sich in vielen Fällen anhand ihrer Basensequenzen parallelisieren, das heißt, sie sind vermutlich aus demselben Ursprungsgen beim gemeinsamen Vorfahren beider Linien hervorgegangen (homolog).
Regulation
Hox-Proteine sind Transkriptionsfaktoren, welche verschiedenen Körperregionen unterschiedliche Identitäten zuweisen. Dies geschieht durch Regulation zahlreicher dahinter (im Jargon der Genetiker: „stromab“) gelegener Gene, viele davon werden durch mehrere Hox-Proteine gesteuert. In einem bestimmten Segment spielen kleine Unterschiede im Ort und Zeitpunkt der Hox-Genexpression eine wichtige Rolle für das Schicksal sich entwickelnder Organe und Zelllinien. Manche Hox-Gene haben Rollen zusätzlich zur zellulären Musterbildung (Lageinformation) übernommen.
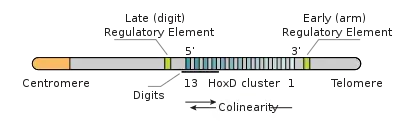
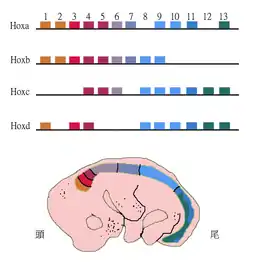
Besonders auffällig ist: Die Reihenfolge der Hox-Gene auf dem Chromosom entspricht der Reihenfolge der von ihnen gesteuerten Körperabschnitte. Zumindest bei den Wirbeltieren entspricht diese auch der Reihenfolge ihrer zeitlichen Exprimierung. Außerdem sind die Hox-Gene meistens in einem einzigen (oder wenigen) Abschnitten direkt benachbart auf dem DNA-Strang angeordnet. Diese regelhafte Anordnung lässt auf zugrunde liegende Regulationsvorgänge schließen, die in großen Teilen des Tierreichs konserviert sind. Sie wird Colinearität genannt, ein wegweisender Begriff, der mit dem Genetiker Denis Duboule verbunden ist.[2][3]
Hox-Gene wirken auf andere Transkriptionsfaktoren, aber auch auf zahlreiche (manchmal hunderte) Effektorgene ein, die sie in der Art eines Schalters ein- oder ausschalten können. Dazu lagern sie sich, wie alle Transkriptionsfaktoren, an einen der proteincodierenden Sequenz des Gens benachbarten Abschnitt (sog. cis-regulatorische Sequenz) an. Gesteuert werden Hox-Gene von anderen, in der Organisation früher ausgeprägten Transkriptionsfaktoren. Diese steuernden Elemente sind im Detail extrem schwierig zu erforschen. Einige Erkenntnisse liegen vom wichtigsten Modellorganismus der Genetiker, der Taufliege Drosophila vor. Demnach sind die regulierenden Sequenzen in Modulen organisiert, die jeweils durch „Puffer“ (trennende Elemente) gegeneinander abgeschirmt sind. Während die Basensequenz der Hox-Gene evolutionär konserviert ist, erwiesen sich die cis-regulatorischen Abschnitte schon bei acht verschiedenen Taufliegen-Arten als sehr verschieden. Innerhalb dieses variablen Gesamtensembles existieren aber offensichtlich kürzere Domänen, die zwischen den verschiedenen Arten so ähnlich sind, dass Schalter einer Art durch Proteine einer anderen Art gesteuert werden konnten. Über die Expression des einzelnen Hox-Gens entscheiden Initiator-Elemente innerhalb des jeweiligen Moduls. Darüber hinaus gibt es weitere regulatorische Proteine, die ganze Module abschalten oder in expressionsfähigem Zustand halten können, indem sie die „Verpackung“ der DNA in Chromatin-Histonkomplexen verändern. Insgesamt sind die Verhältnisse extrem kompliziert und ihre Erforschung steht noch am Anfang. Die Größenordnungen lassen sich etwa dadurch abschätzen, dass an einem bestimmten Hox-Gen von Drosophila (Ubx) die regulatorischen Sequenzen 98 Prozent der Gesamtgröße ausmachen, der proteincodierende Abschnitt 2 Prozent.
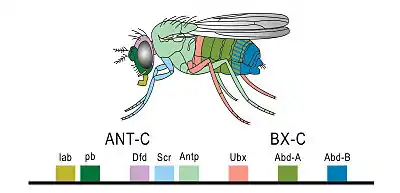
Wird ein Hox-Protein durch eine natürliche oder eine künstlich erzeugte Mutation im falschen Körperabschnitt (ektopisch) exprimiert, hat dies gravierende Konsequenzen für die Entwicklung. Es resultieren schwere Missbildungen, bei denen Organe oder Körperanhänge am falschen Ort entstehen und sich die Identität ganzer Segmente im Körper verändern kann. Man spricht hier von homöotischen Transformationen bzw. Mutationen. Zum Beispiel können bei der Taufliege anstelle von Fühlern Beine wachsen („Antennapedia“), oder anstelle von abdominalen Segmenten werden die thorakalen verdoppelt („Bithorax“). Solche homöotischen Mutationen wurden von Genetikern bereits ab 1915 entdeckt. Sie führten schließlich zur Entdeckung der Hox-Gene. Homöotische Mutationen führen in der Regel zum Tode. Beim Menschen geht z. B. die Ausbildung zusätzlicher Finger (Polydaktylie) vermutlich auf eine homöotische Mutation zurück.
Evolutionäre Entwicklungsbiologie
Die Entdeckung der Hox-Gene war vermutlich der wichtigste Auslöser für die Entstehung einer neuen Forschungsrichtung: der evolutionären Entwicklungsbiologie (englisch: evolutionary developmental biology, häufig abgekürzt als „Evo-Devo“). Dadurch, dass es nun erstmals möglich war, die genetische Basis für grundlegende Entwicklungsmechanismen direkt zu erforschen, ist die besondere Bedeutung der individuellen (ontogenetischen) Entwicklung für die Evolution Basis für ein ganz neues Forschungsprogramm geworden. Wichtig ist über die Bedeutung von entwicklungsmechanischen Zwängen auf Richtung und Geschwindigkeit von Evolutionsvorgängen vor allem, dass nun erstmals eine tatsächliche genetische Basis für grundlegende Körperbaupläne, und damit Möglichkeiten für ihre evolutionäre Veränderung, erkennbar sind. Durch unterschiedliche Expression von Hox-Genen lassen sich Vorgänge wie z. B. die Entwicklung der Körperabschnitte (Tagmata) der Insekten aus gleichartig segmentierten Vorläufern oder die Entstehung des Körperbauplans der Schlangen aus eidechsenartigen Vorläufern verstehen. Weitere wesentliche Erkenntnisse betreffen die Verwandtschaftsverhältnisse ganz an der Basis des Stammbaums der Tiere. Bisher völlig rätselhafte Vorgänge wie die Entstehung der Tierstämme sind dadurch viel besser verstehbar geworden.
Über die tatsächliche Rolle der Hox-Gene selbst innerhalb dieser Veränderungen gibt es innerhalb der Wissenschaft noch kontroverse Vorstellungen. Einige Wissenschaftler sind der Ansicht, neue Körperbaupläne könnten über eine kleine Mutation so quasi in einem Schritt neu entstehen. Sie stellen sich die Entstehung eines neuen Bauplans also wie eine sehr seltene, günstige homöotische Transformation vor. Die meisten Wissenschaftler sind darüber allerdings anderer Ansicht. Sie haben Modelle entwickelt, wie sich durch kleine Verschiebungen im Verhältnis verschiedener Hox-Proteine dieselben Veränderungen ganz graduell ergeben können.
Literatur
- Douglas J. Futuyma, Scott V Edwards, John R True: Evolution. Das Original mit Übersetzungshilfen (= Easy reading.). 1. Auflage, Elsevier, München 2007, ISBN 978-3-8274-1816-6.
- Rüdiger Wehner, Walter Gehring, Alfred Kühn: Zoologie. 24., vollständig überarbeitete Auflage, Thieme, Stuttgart 2007, ISBN 978-3-13-367424-9.
- Edward B. Lewis: A gene complex controlling segmentation in Drosophila. In: Nature. Band 276, Dezember 1978, S. 565–570, doi:10.1038/276565a0.
- Shigehiro Kuraku, Axel Meyer: The evolution and maintenance of Hox gene clusters in vertebrates and the teleost-specific genome duplication. In: International Journal of Developmental Biology. 53, 2009, S. 765–773.
- Gabriel Gellon, William McGinnis: Shaping animal body plans in development and evolution by modulation of Hox expression patterns. In: BioEssays. 20.2, 1998, S. 116–125.
- Joseph C. Pearson, Derek Lemons, William McGinnis: Modulating hox gene functions during animal body patterning. In: Natur Reviews Genetics. 6, 2005, S. 893–904.
- Denis Duboule: The rise and fall of Hox gene clusters. In: Development. 134, 2007, S. 2549–2560.
- Graham E. Budd: Does evolution in body patterning genes drive morphological change – or vice versa? In: BioEssays. 21, 1999, S. 326–332.
- Michalis Averof, Michael Akam: Hox genes and the diversification of insect and crustacean body plans. In: Nature. Band 376, Nr. 3, August 2002, S. 420–423, doi:10.1038/376420a0.
- Jordi Garcia-Fernàndez: The genesis and evolution of homeobox gene clusters. In: Nature Reviews Genetics. 6, 2005, S. 881–892.
- Robert K. Maeda, François Karch: The ABC of the BX-C: the bithorax complex explained. In: Development. 133, 2006, S. 1413–1422.
Weblinks
- Sebastian Teitz: Die Rolle von Homeobox-Genen in der Entwicklung von Drosophila … und insbesondere die Bedeutung von bcd bei der Achsenbildung (Memento vom 13. März 2009 im Internet Archive)
- Mario Hupfeld: Die Geschichte der Hox-Gene; Zentrale für Unterrichtsmedien im Internet e.V.
- Vorlesung Entwicklungsbiologie II (Memento vom 12. November 2004 im Internet Archive) über die Wirkungsweise von Hox Gen-Clustern bei Drosophila
Einzelnachweise
- Kevin Pang, Mark Q. Martindale: Developmental expression of homeobox genes in the ctenophore Mnemiopsis leidyi. In: Development Genes and Evolution. 218, 2008, S. 307–319.
- Stephen J. Gaunt, Paul T. Sharpe, Denis Duboule: Spatially restricted domains of homeo-gene transcripts in mouse embryos: relation to a segmented body plan. In: Development. Band 104, Supplement, Oktober 1988, S. 169–179 (Volltext als PDF-Datei).
- J. C. Izpisua-Belmonte, H. Falkenstein, P. Dolle, A. Renucci, D. Duboule: Murine genes related to the Drosophila AbdB homeotic genes are sequentially expressed during development of the posterior part of the body. In: The EMBO journal. (Embo J) Band 10, Nr. 8, August 1991, S. 2279–2289, PMID 1676674.