Ubiquitin
Ubiquitin ist ein kleines Protein, das in allen eukaryotischen Zellen und Zelltypen zu finden ist – also in Eukaryoten ubiquitär vorkommt – und an der Regulation verschiedener Zellvorgänge beteiligt ist.
| Ubiquitin | ||
|---|---|---|
| ||
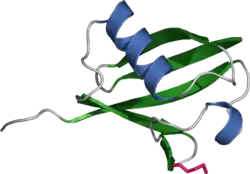
| Bändermodell | ||
| Eigenschaften des menschlichen Proteins | ||
| Masse/Länge Primärstruktur | 8,5 kDa / 76 Aminosäuren | |
| Bezeichner | ||
| Gen-Namen | RPS27A ; UBA52; UBB; UBC | |
| Externe IDs | ||
| Vorkommen | ||
| Übergeordnetes Taxon | Eukaryoten | |
Es wird mittels Ubiquitin-Protein-Ligasen enzymatisch an andere Proteine gekoppelt, die durch diese Ubiquitinierung in ihren Eigenschaften verändert werden. Abhängig von Anzahl und Art der Ubiquitin-Bindungen kann ein ubiquitiniertes Zielprotein dadurch in seiner Interaktion mit anderen Proteinen gefördert oder behindert, seine Aktivität beeinflusst, seine Lokalisation in der Zelle verändert oder sein Abbau beschleunigt werden. Mehrere in Kette angehängte Ubiquitine markieren bei der Proteinqualitätskontrolle das so poly-ubiquitinierte Protein für die Degradation im Proteasom. Ubiquitinierungen sind daneben für die Regelung von Transkription und Translation bedeutend, in die Signaltransduktion und die Endozytose eingebunden, an der DNA-Reparatur beteiligt und treten in geregelten Abläufen von Zellzyklus, Zelldifferenzierung und Entzündungsreaktionen auf.
Die Ubiquitinierung selbst ist ein mehrphasiger Prozess, dessen drei Hauptschritte verschiedene Enzyme katalysieren: ubiquitin-aktivierende (E1), ubiquitin-konjugierende (E2) und schließlich Ubiquitin-Ligasen (E3), die Ubiquitin auf unterschiedliche Art an bestimmte Substratproteine binden.
Demgegenüber steht eine Reihe verschiedener desubiquitinierender Enzyme (DUB), unter deren spezifischer Wirkung unter anderem angehängte Ubiquitinmoleküle wieder entfernt werden können.
Eine Ubiquitinierung, auch Ubiquitinylierung genannt, stellt eine posttranslationale Modifikation von Proteinen dar. Vergleichbare Modifikationen sind Ankopplungen ubiquitin-ähnlicher Proteine wie SUMO, Urm1 oder Nedd8, entsprechend Sumoylierung, Urmylierung bzw. Neddylierung genannt. Daneben ist bei manchen Prokaryoten, beispielsweise Mycobacterium tuberculosis, ein zu Ubiquitin analoges Protein bekannt,[1] das Prokaryotic ubiquitin-like protein (Pup) genannt wird.[2]
Ubiquitin wurde 1975 entdeckt (ubiquitous immunopoietic polypeptide genannt),[3] und in den Folgejahren näher charakterisiert. Für die Erforschung der Grundlagen des Ubiquitin-Systems Anfang der 1980er Jahre wurde Aaron Ciechanover, Avram Hershko und Irwin Rose 2004 der Nobelpreis für Chemie verliehen.[4]
Struktur
Ubiquitin besteht aus 76 Aminosäuren und hat eine Molekülmasse von 8,5 kDa.[5] Sein Aufbau veränderte sich im Laufe der Evolution wenig, es ist somit hoch konserviert. So unterscheiden sich das Protein beim Menschen und bei dem Einzeller Hefe Saccharomyces cerevisiae in nur 3 der 76 Aminosäuren.

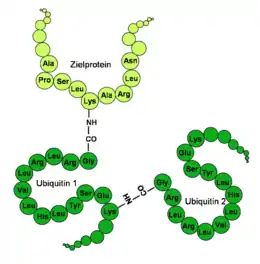
Ubiquitin hat eine globuläre Form, lediglich die letzten vier C-terminalen Aminosäuren ragen hervor. Wichtige funktionelle Aminosäuren sind das C-terminale Glycin (G) an der 76. Stelle (G76) und die Lysine (K) an der 48. (K48) und 63. Stelle (K63) der Aminosäuresequenz. Über die C-terminale Carboxygruppe an G76 wird Ubiquitin an spezifische Lysine, Cysteine, Serine, Threonine oder den N-Terminus des zu markierenden Proteins kovalent gebunden.[6] An ein bereits gebundenes Ubiquitin können über die Lysine weitere Ubiquitinmoleküle angehängt werden, sodass sich eine Ubiquitinkette bildet. Da ein Ubiquitin insgesamt sieben Lysine enthält, sind mindestens sieben verschiedene Verbindungsarten eines Ubiquitins möglich.
Die Aminosäuresequenz für menschliches Ubiquitin im Einbuchstabencode – K48, K63 und G76 gefettet hervorgehoben –:
| N-term MQIFVKTLTGKTITLEVEPSDTIENVKAKIQDKEGIPPDQQRLIFAGKQLEDGRTLSDYNIQKESTLHLVLRLRGG C-term |
Mechanismus der Ubiquitinierung
Der Prozess des Markierens von Zielproteinen durch Ubiquitin wird Ubiquitinierung oder auch Ubiquitinylierung genannt. Dessen Ablauf erfordert – wie eine Sumoylierung, Urmylierung oder Neddylierung – mehrere nacheinander folgende Reaktionsschritte und wird von drei Enzymen katalysiert, Ubiquitin-Protein-Ligasen, die nach der Reaktionsfolge als E1 (auch Modifikation aktivierendes Enzym), E2 (auch Modifikation konjugierendes Enzym) und E3 (auch E3-Ligase) bezeichnet werden.
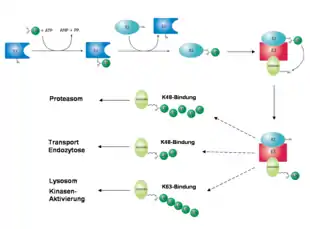
Im ersten Schritt wird Ubiquitin durch eine Thioesterbindung zwischen seiner C-terminalen Carboxygruppe (G76) und einem Cystein des E1-Enzyms gebunden und so „aktiviert“. Diese Aktivierung ist energieabhängig; die Energie wird durch die Spaltung von ATP zu AMP und Pyrophosphat bereitgestellt. Für die Aktivierung des Modifikator-Moleküls gibt es ein spezifisches E1-Enzym, in Pflanzen sogar zwei E1-Enzyme für Ubiquitin.[7]
Nachdem Ubiquitin an E1 gebunden wurde, wird das Ubiquitin an das Enzym E2 überführt. Für Ubiquitin sind allein in der Hefe über elf verschiedene E2-Enzyme bekannt, in anderen Organismen ist ihre Anzahl noch größer (während für Sumo1 und Nedd8 je ein spezifisches E2-Enzym existiert).[8]
Im letzten Schritt wird das Ubiquitin durch spezifische E3-Ligasen auf das Zielprotein übertragen. Hierbei wird eine Isopeptid-Bindung zwischen dem C-terminalen Glycin des Ubiquitins und einem Lysin des Zielproteins gebildet. Im Unterschied zu einer klassischen Peptidbindung dient hier nicht der α-Aminorest, sondern der ε-Aminorest des Lysins als Bindungspartner. Darüber hinaus können Ubiquitine auch auf andere Verknüpfungsarten angeschlossen werden, auch lysin-freie Proteine wurden ubiquitiniert vorgefunden.[9][10] In der Anzahl verschiedener E3-Enzyme spiegelt sich die Vielfalt der von Ubiquitin modifizierten Zielproteine wider. Berücksichtigt man alle Enzyme, die strukturell zu den drei Unterfamilien der E3-Enzyme (HECT, RING und U-Box) gehören, so ist bei höheren Organismen von einer Zahl zwischen mehreren Hundert und Eintausend auszugehen.[7]
Arten der Ubiquitinierung
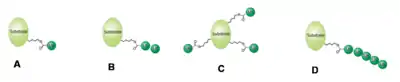
An das jeweilige Zielprotein können Ubiquitine auf verschiedene Weise gebunden sein und weitere an unterschiedlicher Stelle angehängt werden. Nach Anzahl der verbundenen Ubiquitin-Moleküle wird zwischen Mono- und Oligo-, Multi- bzw. Poly-Ubiquitinierung unterschieden, je nachdem ob nur ein Molekül vorliegt oder wenige, mehrere bzw. viele Ubiquitine.[11]
Wenn mindestens fünf Ubiquitinmoleküle als Kette mit einem Zielprotein verbunden sind, spricht man von einer Poly-Ubiquitinierung. Sind diese Moleküle am Lysin 48 (K48) miteinander verknüpft, wird das Zielprotein hauptsächlich dem Abbau durch das Proteasom zugeführt.[12] Verbindung am Lysin 63 (K63) kann zum lysosomalen Abbau des Proteins führen.[13] Des Weiteren wurde beobachtet, dass diese Modifikation Einfluss auf die zelluläre Toleranz von DNA-Schäden, entzündliche Immunantworten, endozytotische Vorgänge und die ribosomale Protein-Synthese hat.[14]
Mono- und Multi-Ubiquitinierungen hingegen beeinflussen weniger die Stabilität einzelner Proteine als deren intrazelluläre Verteilung und können die Interaktion mit anderen Proteinen ermöglichen.[15] Oligo-Ubiquitinierung hat beispielsweise Einfluss auf die Aktivität eines Transkriptionsfaktors, ohne dessen Abbau zu initiieren.[16]
Beispiele für Ubiquitinierungen
Abbau fehlerhaft gefalteter Proteine
Das Ubiquitin-Proteasom-System spielt eine bedeutende Rolle in der „Qualitätssicherung“ intrazellulär hergestellter Proteine.[17] Proteine sollten während und nach ihrer Produktion richtig gefaltet werden, damit sie funktionieren. Bei einigen Proteinen ist die Faltung so komplex und fehleranfällig wie beim Chlorid-Ionenkanal CFTR in Epithelzellen, bei dem bis zu 60–80 % der hergestellten Proteine fehlerhaft gefaltet sind.[18] Diese fehlerhaft gefalteten Proteine werden von sogenannten Chaperonen gebunden, Enzymen, die unter Umständen die richtige Faltung des Proteins fördern können. Bei einer „irreparabelen“ Missfaltung wurde die Bildung eines Protein-Chaperon-Ubiquitin-E3-Ligase-Komplexes beobachtet, der das fehlgefaltete Protein poly-ubiquitiniert und damit die Degradierung durch das Proteasom ermöglicht.[17] Auf diese Weise wird dafür gesorgt, dass strukturell entartete Proteine weder cytosolisch noch membranassoziiert die Zellabläufe beeinflussen.
Ereignet sich aber im Falle des Ionenkanals CFTR in der codierenden DNA eine Mutation, die sich in einer Mutation des Phenylalanins an Position 508 (F508) niederschlägt, führt dies zur Poly-Ubiquitinierung und zu vorzeitigem Abbau aller produzierten CFTR-Proteine.[19] Die Folge ist das Krankheitsbild der Mukoviszidose. Obgleich eine ordnungsgemäße Funktion des mutierten Ionenkanal-Proteins prinzipiell nicht ausgeschlossen ist, wird es vorzeitig abgebaut. Dieses Beispiel zeigt, dass sich das eigentlich positiv wirkende strikte Kontrollsystem des ubiquitinvermittelten Abbaus strukturell falscher Proteine auch negativ auf den Organismus auswirken kann.
Regulation der Transkription
Der erste Schritt der Proteinbiosynthese ist die Transkription. Hierbei wird DNA über ein Enzym, die RNA-Polymerase, in RNA umgeschrieben. Für den Transkriptionsstart der Polymerase werden an der DNA verschiedene Transkriptionsfaktoren benötigt. Die Zugänglichkeit der DNA für die Transkriptionsfaktoren und die Polymerase kann von permanent DNA-gebundenen Proteinkomplexen, den Histonen, reguliert werden. Histone, die von DNA „umwickelt“ sind, werden Nukleosomen genannt.
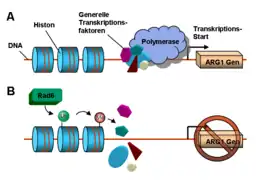
(B) Rad6 mono-ubiquitiniert ein Histon, infolgedessen wird ARG1 nicht mehr exprimiert.
In der Backhefe wurde das ubiquitinverknüpfende Protein Rad6 entdeckt, das die Transkription von ARG1 (Argininosuccinat-Synthase-Gen1) reguliert.[20] In der Abwesenheit von Rad6 können die Transkriptionsfaktoren und die Polymerase an den Promotor (eine regulatorische DNA-Sequenz) vor dem ARG1-Gen binden und die Transkription starten. In der Gegenwart von Rad6 verknüpft dieses ein Ubiquitin-Molekül mit dem Lysin K123 einer Histon-Untereinheit H2B. Dies führt zu Modifikationen eines H3-Histons im Nachbar-Nukleosom: Das Histon H3 wird an den Lysinen K4 und K49 methyliert. Infolgedessen wird der Promotor ruhiggestellt, sodass keine Transkriptionsfaktoren binden können. Durch dieses Gen-Silencing wird nun das Gen ARG1 nicht mehr exprimiert und das Enzym Argininosuccinat-Synthase in der Zelle nicht mehr hergestellt.[21]
Darüber hinaus war das Histon H2A aus der Taufliege das erste ubiquitinierte Protein, das beschrieben wurde.[22][23] In Säugetieren wurde der Ubiquitinierungszustand der Histone H2A und H2B zum ersten Marker für transkriptionell aktives Chromatin, der Gesamtheit aus der DNA und deren assoziierten Proteinen.[24]
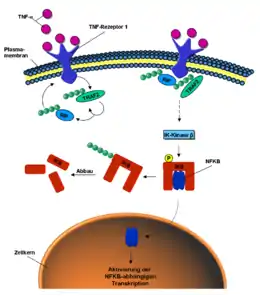
Ubiquitin als Teil der Signaltransduktion
Ubiquitin ist auch an der intrazellulären Signal-Weiterleitung von äußeren Stimuli beteiligt, so zum Beispiel beim NF-κB-Signalweg (engl. nuclear factor kappa B).[25] Dieser kann durch das Signalmolekül Tumornekrosefaktor (TNF) aktiviert werden. Bindet TNF an den TNF-Rezeptor der Zellmembran, wird durch dessen Konformationsänderung die E3-Ligase TRAF2 an den intrazellulären Teil des Rezeptors rekrutiert. Diese poly-ubiquitiniert sich selbst und das Protein RIP über K63-Verbindungen.[26] Durch die ubiquitinierten Proteine RIP und TRAF2 werden verschiedene Kinasen, phosphorylierende Enzyme, aktiviert. Die Iκ-Kinase β letztendlich phosphoryliert das Protein IκB. Dieses setzt nun den vorher gebundenen und inaktiven NF-κB frei. NF-κB wandert in den Zellkern und aktiviert dort die Transkription bestimmter Gene.[27] IkB hingegen wird über K48 poly-ubiquitiniert und über das Proteasom abgebaut.[28]
Weitere Beispiele für Ubiquitinierungen
- Nach Ende der Mitose wird das am Zellzyklus beteiligte Cyclin durch Ubiquitinierung markiert und abgebaut.[29]
- Bei der HIV-Infektion werden anti-virale Enzyme der Zelle (ABOBEC3G) durch ein virales HIV-Protein (Vif) gebunden. Vif vermag gleichzeitig Teile der Ubiquitinierungs-Maschinerie zu binden. Vif wird dadurch ubiquitiniert und zusammen mit APOBEC3G degradiert, wodurch die Effizienz der HIV-Infektion gesteigert wird.[30]
- Vermehrte Mono-Ubiquitinierung tritt bei der Differenzierung von multipotenten Stammzellen auf.[31]
Erkrankungen
Das Angelman-Syndrom ist eine neurologische Erkrankung, die sich u. a. durch eine verlangsamte kognitive und motorische Entwicklung äußert. Der häufigste genetische Defekt ist hierbei eine 4 Mio. (MBp) Basenpaar-Deletion auf dem mütterlichen Chromosom 15 Genlocus q11-13. Diese Region ist jedoch nur im Hippocampus und im Kleinhirn aktiv und codiert u. a. für die E3-Ubiquitinligase E6-AP.[32] Mäuse, denen diese Ligase fehlt, entwickeln Lerndefizite, beispielsweise bei der Konditionierung von Angst. Zudem ist die längerfristige neuronale Plastizität der Mäuse nicht mehr gegeben. Diese Defizite korrelieren teils mit den Beeinträchtigungen von Patienten mit Angelman-Syndrom.
- Im dominant vererbten Morbus Hippel-Lindau führt eine Mutation im Gen der VHL-Ubiquitinligase zu einer Akkumulation des Transkriptionsfaktors Hypoxie-induzierter Faktor (HIF) und Tumorentstehung.
- Mutationen der Ubiquitinligase Parkin wurde bei bestimmten Formen der Parkinson-Krankheit nachgewiesen.[33]
- Mutationen der Cullin7-E3-Ubiquitinligase wurden als Ursache der autosomal-rezessiven Wachstumsstörung 3M-Syndrom identifiziert.[34]
- Mutationen im Ubiquitin Activating Enzyme UBA1 finden sich beim VEXAS-Syndrom und der X-chromosomalen infantilen Spinalen Muskelatrophie
Literatur
- Roland John Mayer, Aaron J. Ciechanover, Martin Rechsteiner: Protein Degradation (= The Ubiquitin-Proteasome System and Disease, Band 4), Wiley-VCH, Weinheim 2008, ISBN 978-3-527-31436-2.
- P. Ebner, G. A. Versteeg, F. Ikeda: Ubiquitin enzymes in the regulation of immune responses. In: Critical Reviews in Biochemistry and Molecular Biology. Band 52, Nummer 4, August 2017, S. 425–460, doi:10.1080/10409238.2017.1325829, PMID 28524749, PMC 5490640 (freier Volltext) (Review).
- A. Varshavsky: The Ubiquitin System, Autophagy, and Regulated Protein Degradation. In: Annual review of biochemistry. Band 86, Juni 2017, S. 123–128, doi:10.1146/annurev-biochem-061516-044859, PMID 28654326 (Review).
- P. M. Lombardi, M. J. Matunis, C. Wolberger: RAP80, ubiquitin and SUMO in the DNA damage response. In: Journal of molecular medicine. Band 95, Nummer 8, August 2017, S. 799–807, doi:10.1007/s00109-017-1561-1, PMID 28681078, PMC 5570449 (freier Volltext) (Review).
Einzelnachweise
- M. J. Pearce, J. Mintseris u. a.: Ubiquitin-like protein involved in the proteasome pathway of Mycobacterium tuberculosis. In: Science. Band 322, Nummer 5904, November 2008, S. 1104–1107, doi:10.1126/science.1163885
- J. A. Maupin-Furlow: Prokaryotic ubiquitin-like protein modification. In: Annual review of microbiology. Band 68, 2014, S. 155–175, doi:10.1146/annurev-micro-091313-103447
- G. Goldstein, M. Scheid, U. Hammerling, D. Schlesinger, H. Niall, E. Boyse: Isolation of a polypeptide that has lymphocyte-differentiating properties and is probably represented universally in living cells. In: Proc Natl Acad Sci U S A. Band 72, Nr. 1, Januar 1975, S. 11–5; doi:10.1073/pnas.72.1.11, PMID 1078892, PMC 432229 (freier Volltext).
- Informationen der Nobelstiftung zur Preisverleihung 2004 an Avram Hershko und Irwin Rose (englisch)
- UniProt P62988
- Cecile M. Pickart, Shahri Raasi: Controlled Synthesis of Polyubiquitin Chains. In: Methods in Enzymology. Elsevier, 2005, ISBN 978-0-12-182804-2, S. 21–36, doi:10.1016/s0076-6879(05)99002-2 (elsevier.com [abgerufen am 27. Mai 2018]).
- C. M. Pickart, M. J. Eddins: Ubiquitin: structures, functions, mechanisms. In: Biochim Biophys Acta. 1695 (1–3), 29. November 2004, S. 55–72. PMID 15571809.
- C. M. Pickart: Mechanisms underlying ubiquitination. In: Annu Rev Biochem. 70, 2001, S. 503–533. PMID 11395416.
- K. Cadwell, L. Coscoy: Ubiquitination on nonlysine residues by a viral E3 ubiquitin ligase. In: Science. 309 (5731), 1. Juli 2005, S. 127–130. PMID 15994556.
- A. Ciechanover, R. Ben-Saadon: N-terminal ubiquitination: more protein substrates join in. In: Trends Cell Biol. 14 (3), März 2004, S. 103–106. PMID 15055197.
- D. Mukhopadhyay, H. Riezman: Proteasome-independent functions of ubiquitin in endocytosis and signaling. In: Science. 315 (5809), 12. Januar 2007, S. 201–205. PMID 17218518.
- A. Hershko, A. Ciechanover: The ubiquitin system for protein degradation. In: Annu Rev Biochem. 61, 1992, S. 761–807. PMID 1323239.
- H. Barriere, C. Nemes, K. Du, G. L. Lukacs: Plasticity of polyubiquitin recognition as lysosomal targeting signals by the endosomal sorting machinery. In: Mol Biol Cell. 18 (10), Oktober 2007, S. 3952–3965. PMID 17686993.
- C. M. Pickart, D. Fushman: Polyubiquitin chains: polymeric protein signals. In: Curr Opin Chem Biol. 8 (6), Dezember 2004, S. 610–616. PMID 15556404.
- S. Polo, S. Sigismund, M. Faretta, M. Guidi, M. R. Capua, G. Bossi, H. Chen, P. De Camilli, P. P. Di Fiore: A single motif responsible for ubiquitin recognition and monoubiquitination in endocytic proteins. In: Nature. 416 (6879), 28. März 2002, S. 451–455. PMID 11919637.
- K. Flick, I. Ouni, J. A. Wohlschlegel, C. Capati, W. H. McDonald, J. R. Yates, P. Kaiser: Proteolysis-independent regulation of the transcription factor Met4 by a single Lys 48-linked ubiquitin chain. In: Nat Cell Biol. 6 (7), Juli 2004, S. 634–641. 20. Juni. PMID 15208638.
- C. Esser, S. Alberti, J. Höhfeld: Cooperation of molecular chaperones with the ubiquitin/proteasome system. In: Biochim Biophys Acta. 1695 (1–3), 29. November 2004, S. 171–188. PMID 15571814.
- R. R. Kopito: Biosynthesis and degradation of CFTR. In: Physiol Rev. 79 (1 Suppl), Januar 1999, S. 167–173. PMID 9922380.
- C. L. Ward, S. Omura, R. R. Kopito: Degradation of CFTR by the ubiquitin-proteasome pathway. In: Cell. 83 (1), 6. Oktober 1995, S. 121–127. PMID 7553863.
- K. Robzyk, J. Recht, M. A. Osley: Rad6-dependent ubiquitination of histone H2B in yeast. In: Science. 287 (5452), 21. Januar 2000, S. 501–504. PMID 10642555.
- Z. W. Sun, C. D. Allis: Ubiquitination of histone H2B regulates H3 methylation and gene silencing in yeast. In: Nature. 418 (6893), 4. Juli 2002, S. 104–108. PMID 12077605.
- I. L. Goldknopf, H. Busch: Isopeptide linkage between nonhistone and histone 2 A polypeptides of chromosomal conjugate-protein A24. In: Proc Natl Acad Sci U S A. 74 (3), März 1977, S. 864–868. PMID 265581.
- L. T. Hunt, M. O. Dayhoff: Amino-terminal sequence identity of ubiquitin and the nonhistone component of nuclear protein A24. In: Biochem Biophys Res Commun. 74 (2), 24. Januar 1977, S. 650–655. PMID 836318.
- S. Y. Huang, M. B. Barnard, M. Xu, S. Matsui, S. M. Rose, W. T. Garrard: The active immunoglobulin kappa chain gene is packaged by non-ubiquitin-conjugated nucleosomes. In: Proc Natl Acad Sci U S A. 83 (11), Juni 1986, S. 3738–3742. PMID 3012532.
- M. Karin, Y. Ben-Neriah: Phosphorylation meets ubiquitination: the control of NF-[kappa]B activity. In: Annu Rev Immunol. 18, 2000, S. 621–663. PMID 10837071.
- E. Meylan, J. Tschopp: The RIP kinases: crucial integrators of cellular stress. In: Trends Biochem Sci. 30 (3), März 2005, S. 151–159. PMID 15752987.
- T. D. Gilmore: Introduction to NF-kappaB: players, pathways, perspectives. In: Oncogene. 25 (51), 30. Oktober 2006, S. 6680–6884. PMID 17072321.
- M. Magnani, R. Crinelli, M. Bianchi, A. Antonelli: The ubiquitin-dependent proteolytic system and other potential targets for the modulation of nuclear factor-kB (NF-kB). In: Curr Drug Targets. 1 (4), Dezember 2000, S. 387–399. PMID 11467077.
- F. Bassermann, C. von Klitzing, S. Münch, R. Y. Bai, H. Kawaguchi, S. W. Morris, C. Peschel, J. Duyster: NIPA defines an SCF-type mammalian E3 ligase that regulates mitotic entry. In: Cell. 122 (1), 15. Juli 2005, S. 45–57. PMID 16009132.
- Y. Dang, L. M. Siew, Y. H. Zheng: APOBEC3G is degraded by the proteasomal pathway in a Vif-dependent manner without being polyubiquitylated. In: J Biol Chem. 6. März 2008. PMID 18326044.
- O. Karpiuk, Z. Najafova, F. Kramer, M. Hennion, C. Galonsk, A. König, N. Snaidero, T. Vogel, T. A. Shchebet, Y. Begus-Nahrmann, M. Kassem, M. Simons, H. Shcherbata, T. Beissbarth, S. A. Johnsen: The histone H2B monoubiquitination regulatory pathway is required for differentiation of multipotent stem cells. In: Molecular Cell. 46 (5), 2002, S. 705–713.
- E. Weeber, J. Levenson, J. Sweatt: Molecular genetics of human cognition. In: Mol Interv. Band 2, Nr. 6, 2002, S. 376–391, 339, doi:10.1124/mi.2.6.376, PMID 14993414.
- H. Shimura, N. Hattori, S. Kubo, Y. Mizuno, S. Asakawa, S. Minoshima, N. Shimizu, K. Iwai, T. Chiba, K. Tanaka, T. Suzuki: Familial Parkinson disease gene product, parkin, is a ubiquitin-protein ligase. In: Nat Genet. 25 (3), Juli 2000, S. 302–305. PMID 10888878.
- C. Huber, D. Dias-Santagata, A. Glaser, J. O’Sullivan, R. Brauner, K. Wu, X. Xu, K. Pearce, R. Wang, M. L. Uzielli, N. Dagoneau, W. Chemaitilly, A. Superti-Furga, H. Dos Santos, A. Mégarbané, G. Morin, G. Gillessen-Kaesbach, R. Hennekam, I. Van der Burgt, G. C. Black, P. E. Clayton, A. Read, M. Le Merrer, P. J. Scambler, A. Munnich, Z. Q. Pan, R. Winter, V. Cormier-Daire: Identification of mutations in CUL7 in 3-M syndrome. In: Nat Genet. 37 (10), Oktober 2005, S. 1119–1124. PMID 16142236.
Weblinks
- The Ubiquitin System for Protein Modification and Degradation. Informationen zu Ubiquitin an der Universität Nottingham.
- Jennifer McDowall: Ubiquitin. (englisch).