Transkriptionsfabrik
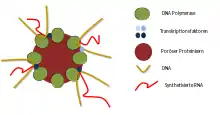
In der Genetik beschreiben Transkriptionsfabriken (auch Transkriptionsfoci, engl. Transcription Factory) die Orte, an denen die Transkription im Zellkern stattfindet. Sie wurden erstmals 1993 entdeckt und weisen ähnliche Strukturen auf wie Replikationsfabriken, also Orte, an denen die Replikation stattfindet. Die Transkriptionsfabriken enthalten eine RNA-Polymerase (aktiv oder inaktiv) und die notwendigen Transkriptionsfaktoren (Aktivatoren und Repressoren) für die Transkription.[1] Transkriptionsfabriken, die RNA-Polymerase II enthalten, wurden bisher am intensivsten untersucht. Es können auch Fabriken für RNA-Polymerase I und III existieren; der Nucleolus kann als Prototyp für Transkriptionsfabriken angesehen werden. Es ist möglich, diese sowohl unter der Licht- als auch unter der Elektronenmikroskopie zu betrachten. Die Entdeckung der Transkriptionsfabriken hat die ursprüngliche Sichtweise, wie RNA-Polymerase mit der DNA interagiert, in Frage gestellt. Es wird angenommen, dass die Anwesenheit dieser Fabriken wichtige Auswirkungen auf die Genregulation und die Struktur des Zellkerns hat.
Entdeckung
Die erste Verwendung des Begriffs "Transcription Factories" wurde 1993 von D. A. Jackson und seinen Kollegen verwendet, die feststellten, dass die Transkription an bestimmten Stellen im Kern erfolgte.[2] Dies widersprach der ursprünglichen Ansicht, dass die Transkription gleichmäßig über den Kern verteilt erfolgte.
Struktur
Die Struktur einer Transkriptionsfabrik scheint durch den Zelltyp, die Transkriptionsaktivität der Zelle, aber auch durch die Methode der Technik zur Visualisierung der Struktur bestimmt zu sein. Eine verallgemeinerte Sicht auf eine Transkriptionsfabrik würde zwischen 4 und 30 RNA-Polymerase-Moleküle[3] enthalten. Es wird angenommen, dass je transkriptionell aktiver eine Zelle ist, desto mehr Polymerasen sind in einer Fabrik vorhanden, um die Anforderungen der Transkription zu erfüllen. Der Kern einer Transkriptionsfabrik ist porös und proteinreich, mit den hyperphosphorylierten, länglich geformten Polymerasen am Rand. Zu den vorhandenen Proteinen gehören: Ribonukleoproteine, Coaktivatoren, Transkriptionsfaktoren, RNA-Helikase sowie Spleiß- und Verarbeitungsenzyme.[4] Eine Fabrik enthält nur eine Art der RNA-Polymerase. Der Durchmesser der Fabrik variiert je nach der verwendeten RNA-Polymerase; die RNA-Polymerase-I-Fabriken sind etwa 500 nm breit, während die RNA-Polymerase-II- und III-Fabriken mit 50 nm eine Größenordnung kleiner sind.[5] Es wurde experimentell gezeigt, dass eine Transkriptionsfabrik stationär ist. Es wird postuliert, dass diese Immobilisierung auf eine Bindung an die Kernmatrix zurückzuführen ist. Hier wurde gezeigt, dass sie an eine Struktur gebunden ist, die von Restriktionsenzymen nicht beeinflusst wird. Proteine, von denen angenommen wurde, dass sie an der Bindung beteiligt sind, umfassen Spektrin, Aktin und Lamin.[6]
Funktion
Die Struktur einer Transkriptionsfabrik steht in direktem Zusammenhang mit ihrer Funktion. Die Transkription wird durch die bündelnde Natur der Transkriptionsfabrik effizienter gestaltet. Alle notwendigen Proteine wie RNA-Polymerase, Transkriptionsfaktoren und andere Co-Regulatoren sind in der Transkriptionsfabrik vorhanden, was eine schnellere RNA-Polymerisation ermöglicht, wenn die DNA die Fabrik erreicht. Es ermöglicht zudem, dass eine Reihe von Genen gleichzeitig transkribiert werden können.[7]
Anzahl
Die Anzahl der pro Kern vorhandenen Transkriptionsfabriken scheint durch Zelltyp, Spezies und die Art der Messung bestimmt zu sein. Es wurde festgestellt, dass embryonale Fibroblasten von Zuchtmäusen etwa 1500 Fabriken enthalten, nachgewiesen durch Immunfluoreszenznachweis von RNA-Polymerase II. Zellen aus verschiedenen Geweben derselben Gruppen von Mäusen hatten jedoch zwischen 100 und 300 Fabriken.[8] Messungen der Anzahl der Transkriptionsfabriken in HeLa-Zellen lieferten unterschiedliche Ergebnisse. So wurden beispielsweise mit dem traditionellen Fluoreszenzmikroskopieansatz 300–500 Fabriken gefunden, aber mit Hilfe der Konfokal- und Elektronenmikroskopie wurden etwa 2100 nachgewiesen.[9]
Spezialisierung von Fabriken
Neben den Spezialisierung von Fabriken durch die Art der enthaltenden RNA-Polymerasen können sie es eine weitere Spezialisierungsstufe aufweisen. Es gibt einige Fabriken, die nur einen bestimmten Satz verwandter Gene transkribieren. Dies stärkt das Konzept weiter, dass die Hauptfunktion einer Transkriptionsfabrik in der Transkriptionseffizienz besteht.[10]
Zusammenbau und Wartung
Es wird darüber diskutiert, ob sich Transkriptionsfabriken aufgrund der transkriptionellen Anforderungen des Genoms zusammenschließen oder ob es sich um stabile Strukturen handelt, die über die Zeit bestehen bleiben. Experimentell scheint es, dass sie über einen kurzen Zeitraum fixiert bleiben; neu hergestellte mRNA wurden über 15 Minuten markiert und es zeigten sich keine neuen Transkriptionsfabriken.[11] Dies wird auch durch Experimente zur Hemmung der Transkription unterstützt. In diesen Studien wurde ein Hitzeschock durchgeführt, um die Transkription abzuschalten. Dies führte zu keiner Änderung der Anzahl der erfassten Polymerasen.[12] Nach weiterer Analyse der Western-Blot-Daten wurde angenommen, dass es tatsächlich einen leichten Rückgang der Anzahl der Transkriptionsfabriken im Laufe der Zeit gab. Daher könnte man behaupten, dass die Polymerase-Moleküle im Laufe der Zeit aus der Fabrik freigesetzt werden, wenn es an Transkription mangelt. Dies würde schließlich zum vollständigen Verlust der Transkriptionsfabrik führen.[13]
Es gibt auch mehrere Hinweise darauf, dass Transkriptionsfabriken aufgrund von Notwendigkeiten für die Transkription neu aufgebaut werden. Fluoreszenzexperimente mit GFP-Polymerase haben gezeigt, dass die Induktion der Transkription in den Polytänchromosomen von Drosophila zur Entstehung einer Fabrik führt. Dies widerspricht dem Begriff einer stabilen und sicheren Struktur widerspricht.[14]
Mechanismen
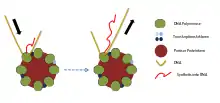
Bisher wurde angenommen, dass es die relativ kleine RNA-Polymerase war, die sich während der Transkription entlang der vergleichsweise größeren DNA-Vorlage bewegt. Immer mehr Beweise sprechen jedoch dafür, dass es aufgrund der Anbindung einer Transkriptionsfabrik an die Kernmatrix tatsächlich das große DNA-Molekül ist, das bewegt wird, um die RNA-Polymerisation aufzunehmen. In vitro-Studien haben zum Beispiel gezeigt, dass RNA-Polymerasen, die an eine Oberfläche gebunden sind, in der Lage sind, sowohl die DNA-Vorlage zu drehen als auch sie durch die Polymerase zu führen, um die Transkription zu starten; dies zeigt, dass die RNA-Polymerase ein molekularer Motor ist. Untersuchungen mit der Chromosom Conformation Capture (3C) Technik unterstützen auch die Idee, dass die DNA-Vorlage durch eine stationäre RNA-Polymerase diffundiert.
An diesem vorgeschlagenen Mechanismus der Transkription bestehen jedoch auch Zweifel. Erstens ist nicht bekannt, wie eine stationäre Polymerase in der Lage ist, Gene auf dem (+)-Strang und (−)-Strang am gleichen genomischen Ort gleichzeitig zu transkribieren. Hinzu kommt ein Mangel an schlüssigen Beweisen dafür, wie die Polymerase immobilisiert bleibt und an welche Struktur sie gebunden ist.[15]
Einfluss auf die genomische und nukleäre Struktur
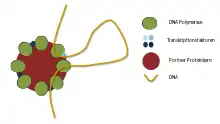
Die Bildung einer Transkriptionsfabrik hat mehrere Konsequenzen für nukleäre und genomische Strukturen. Es wurde vorgeschlagen, dass die Fabriken für die nukleäre Organisation verantwortlich sind. Zudem wurde vorgeschlagen, die Bildung von Chromatinschleifen durch zwei mögliche Mechanismen zu fördern:
Der erste Mechanismus schlägt vor, dass sich Schleifen bilden, weil zwei Gene auf dem gleichen Chromosom die gleiche Transkriptionsmaschine benötigen, die in einer bestimmten Transkriptionsfabrik zu finden wäre. Diese Anforderung zieht die Genorte in die Fabrik und schafft so eine Schleife.[16]
Der zweite Mechanismus deutet darauf hin, dass die Bildung einer Chromatinschleife auf "entropische Anziehungskräfte" (engl. depletion attraction) zurückzuführen ist. Dies ist ein physikalisches Phänomen, das auftritt, wenn sich ein relativ großes Objekt (z. B. eine Transkriptionsfabrik) in einem gefüllten Gebiet mit löslichen Objekten (z. B. Proteine) befindet. Die Transkriptionsfabriken neigen dazu, sich zu aggregieren. Diese Zusammenlagerung verhindert, dass kleinere Objekte Teil der Überlappungsregion werden. Somit wird die Entropie des Systems reduziert. Deshalb würde sich eine Chromatinschleife zwischen den beiden Fabriken bilden.[17]
Für Transkriptionsfabriken wird auch vorgeschlagen, für das Gen-Clustering verantwortlich zu sein, da verwandte Gene die gleiche Transkriptionsmaschine benötigen würden. Wenn eine Fabrik diese Anforderungen erfüllt, würden die Gene von der Fabrik angezogen werden.[18]
Während die Gruppierung von Genen für die Effizienz der Transkription von Vorteil sein kann, könnte dies auch negative Folgen haben. Gen-Verlagerungen treten auf, wenn die Gene in unmittelbarer Nähe zueinander liegen; sie treten häufiger auf, wenn eine Transkriptionsfabrik vorhanden ist. Gen-Translokationsereignisse, wie Punktmutationen, sind in der Regel schädlich für den Organismus und können daher zu Krankheiten führen. Andererseits hat die jüngste Forschung jedoch gezeigt, dass es keinen Zusammenhang zwischen Interaktionen zwischen Genen und Häufigkeit von Translokationen gibt.[19]
Siehe auch
Einzelnachweise
- Iborra F: Active RNA polymerases are localised within discrete transcription "factories" in human nuclei. In: Journal of Cell Science. Band 109, 1996, S. 1427–1436.
- D. A. Jackson, A. B. Hassan, R. J. Errington, P. R. Cook: Visualization of focal sites of transcription within human nuclei. In: The EMBO journal. Band 12, Nr. 3, März 1993, ISSN 0261-4189, S. 1059–1065, PMID 8458323, PMC 413307 (freier Volltext).
- Iborra F: Active RNA polymerases are localised within discrete transcription "factories" in human nuclei. In: Journal of Cell Science. Band 109, 1996, S. 1427–1436.
- Svitlana Melnik, Binwei Deng, Argyris Papantonis, Sabyasachi Baboo, Ian M Carr: The proteomes of transcription factories containing RNA polymerases I, II or III. In: Nature Methods. Band 8, Nr. 11, November 2011, ISSN 1548-7091, S. 963–968, doi:10.1038/nmeth.1705, PMID 21946667, PMC 3324775 (freier Volltext) – (nature.com [abgerufen am 8. August 2019]).
- C. H. Eskiw, P. Fraser: Ultrastructural study of transcription factories in mouse erythroblasts. In: Journal of Cell Science. Band 124, Nr. 21, 1. November 2011, ISSN 0021-9533, S. 3676–3683, doi:10.1242/jcs.087981, PMID 22045738, PMC 3215576 (freier Volltext) – (biologists.org [abgerufen am 8. August 2019]).
- Svitlana Melnik, Binwei Deng, Argyris Papantonis, Sabyasachi Baboo, Ian M Carr: The proteomes of transcription factories containing RNA polymerases I, II or III. In: Nature Methods. Band 8, Nr. 11, November 2011, ISSN 1548-7091, S. 963–968, doi:10.1038/nmeth.1705, PMID 21946667, PMC 3324775 (freier Volltext) – (nature.com [abgerufen am 11. August 2019]).
- Argyris Papantonis, Peter R. Cook: Fixing the model for transcription: The DNA moves, not the polymerase. In: Transcription. Band 2, Nr. 1, Januar 2011, ISSN 2154-1264, S. 41–44, doi:10.4161/trns.2.1.14275, PMID 21326910, PMC 3023647 (freier Volltext) – (tandfonline.com [abgerufen am 8. August 2019]).
- Cameron S Osborne, Lyubomira Chakalova, Karen E Brown, David Carter, Alice Horton: Active genes dynamically colocalize to shared sites of ongoing transcription. In: Nature Genetics. Band 36, Nr. 10, Oktober 2004, ISSN 1061-4036, S. 1065–1071, doi:10.1038/ng1423 (nature.com [abgerufen am 8. August 2019]).
- Iborra F: Active RNA polymerases are localised within discrete transcription "factories" in human nuclei. In: Journal of Cell Science. Band 109, 1996, S. 1427–1436.
- Cameron S Osborne, Lyubomira Chakalova, Karen E Brown, David Carter, Alice Horton: Active genes dynamically colocalize to shared sites of ongoing transcription. In: Nature Genetics. Band 36, Nr. 10, Oktober 2004, ISSN 1061-4036, S. 1065–1071, doi:10.1038/ng1423 (nature.com [abgerufen am 8. August 2019]).
- Iborra F: Active RNA polymerases are localised within discrete transcription "factories" in human nuclei. In: Journal of Cell Science. Band 109, 1996, S. 1427–1436.
- S Lindquist: The Heat-Shock Response. In: Annual Review of Biochemistry. Band 55, Nr. 1, Juni 1986, ISSN 0066-4154, S. 1151–1191, doi:10.1146/annurev.bi.55.070186.005443 (annualreviews.org [abgerufen am 8. August 2019]).
- J. A. Mitchell, P. Fraser: Transcription factories are nuclear subcompartments that remain in the absence of transcription. In: Genes & Development. Band 22, Nr. 1, 1. Januar 2008, ISSN 0890-9369, S. 20–25, doi:10.1101/gad.454008, PMID 18172162, PMC 2151011 (freier Volltext) – (genesdev.org [abgerufen am 11. August 2019]).
- M. Becker: Dynamic behavior of transcription factors on a natural promoter in living cells. In: EMBO Reports. Band 3, Nr. 12, 16. Dezember 2002, S. 1188–1194, doi:10.1093/embo-reports/kvf244, PMID 12446572, PMC 1308318 (freier Volltext) – (embopress.org [abgerufen am 8. August 2019]).
- M. Becker: Dynamic behavior of transcription factors on a natural promoter in living cells. In: EMBO Reports. Band 3, Nr. 12, 16. Dezember 2002, S. 1188–1194, doi:10.1093/embo-reports/kvf244, PMID 12446572, PMC 1308318 (freier Volltext) – (embopress.org [abgerufen am 11. August 2019]).
- Stefan Schoenfelder, Tom Sexton, Lyubomira Chakalova, Nathan F Cope, Alice Horton: Preferential associations between co-regulated genes reveal a transcriptional interactome in erythroid cells. In: Nature Genetics. Band 42, Nr. 1, Januar 2010, ISSN 1061-4036, S. 53–61, doi:10.1038/ng.496, PMID 20010836, PMC 3237402 (freier Volltext) – (nature.com [abgerufen am 9. August 2019]).
- Marenduzzo D: Entropy-driven genome organisation. In: Biophys J. J. 42, 2006, S. 3712–3721.
- Peter R. Cook: A Model for all Genomes: The Role of Transcription Factories. In: Journal of Molecular Biology. Band 395, Nr. 1, Januar 2010, S. 1–10, doi:10.1016/j.jmb.2009.10.031 (elsevier.com [abgerufen am 9. August 2019]).
- I. G. Cowell, Z. Sondka, K. Smith, K. C. Lee, C. M. Manville: Model for MLL translocations in therapy-related leukemia involving topoisomerase II -mediated DNA strand breaks and gene proximity. In: Proceedings of the National Academy of Sciences. Band 109, Nr. 23, 5. Juni 2012, ISSN 0027-8424, S. 8989–8994, doi:10.1073/pnas.1204406109, PMID 22615413, PMC 3384169 (freier Volltext) – (pnas.org [abgerufen am 9. August 2019]).