Topologically associating domain
Eine Topologically associating domain (TAD, engl. „topologisch assoziierte Domäne“) ist eine selbst-interagierende genomische Region, d. h. DNA-Sequenzen innerhalb einer TAD wechselwirken physikalisch häufiger miteinander als mit Sequenzen außerhalb der TAD.[1] Diese dreidimensionalen Chromosomenstrukturen sind bei Tieren sowie bei einigen Pflanzen, Pilzen und Bakterien vorhanden. TADs können in der Größe von Tausenden bis Millionen von DNA-Basen variieren.
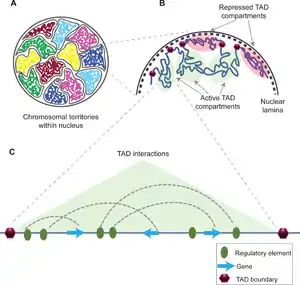
Die Funktionen von TADs sind nicht vollständig verstanden, aber in einigen Fällen führt die Störung von TADs zu Krankheiten, da die Veränderung der 3D-Organisation des Chromosoms die Genregulation stört. Die Mechanismen der TAD-Bildung sind ebenfalls komplex und noch nicht vollständig geklärt, obwohl eine Reihe von Proteinkomplexen und DNA-Elementen mit TAD-Grenzen assoziiert sind.
Entdeckung und Definition
TADs sind definiert als Regionen, deren DNA-Sequenzen vorzugsweise miteinander in Kontakt stehen. Sie wurden 2012 mit Hilfe der Chromosome Conformation Capture Technik einschließlich Hi-C[2][3][4] entdeckt. Die Weiterentwicklungen dieser Technologien, 4C und Hi-C, ermöglichten die genomweite Ermittlung aller interagierenden Genloki miteinander. TADs sind nachweislich in Fruchtfliegen (Drosophila), Mäusen und menschlichen Genomen vorhanden, aber nicht in der Hefe Saccharomyces cerevisiae.
TADs haben die Eigenschaft, dass innerhalb der Domänen chromosomale Wechselwirkungen mit hoher Frequenz stattfinden. Untereinander sind die Interaktionen zwischen den TADs nur noch schwach vorhanden.
Die Positionen von TADs werden durch die Anwendung eines Algorithmus auf Hi-C-Daten definiert. Beispielsweise werden TADs oft durch den sogenannten "Directionality Index" bestimmt.[5] Dieser Richtungsindex wird für einzelne 40kb Abschnitte berechnet, indem die Sequenzierungen, die in diese Abschnitte fallen, gesammelt werden. Dann wird beobachtet, ob ihre gepaarten Sequenzfolgen vor oder hinter dem Abschnitt liegen (diese Paarungen sind erforderlich, um nicht mehr als 2Mb zu überspannen). Ein positiver Wert zeigt an, dass mehr gelesene Paare stromabwärts als stromaufwärts liegen, und ein negativer Wert zeigt das Gegenteil an. Mathematisch gesehen ist der Richtungsindex eine vorzeichenbehaftete Chi-Quadrat-Statistik.
Entstehungsmechanismen
Es ist bekannt, dass eine Reihe von Proteinen mit der TAD-Bildung assoziiert sind, darunter das Protein CTCF und der Proteinkomplex Cohesin.[6][7] Es ist nicht bekannt, welche Komponenten an den TAD-Grenzen benötigt werden; in Säugetierzellen konnte jedoch gezeigt werden, dass diese Grenzregionen ein vergleichsweise hohes Maß an CTCF-Bindungen aufweisen. Darüber hinaus treten einige Arten von Genen (wie Transfer-RNA-Gene und Haushaltgene) häufiger in der Nähe von TAD-Grenzen auf, als es der Zufall erwarten würde.[2][4]
Alle TADs sind auf Hi-C-Kontaktkarten als "Dreiecke" zu erkennen, welche die Regionen mit erhöhter Häufigkeit interner Kontakte beschreiben. Hierbei weisen die gebildeten Dreiecke in etwa 50 % der Fälle sehr starke Spitzen auf. TADs mit solchen Kontaktmustern bilden Chromatinschlaufen, wobei ihre Randelemente einander berühren. ChIP-Seq-Studien zeigten, dass sich berührende Chromatin-Regionen an der Basis der gebildeten Schlaufen durch CTCF-Proteine und Cohesin gebunden sind.[8]
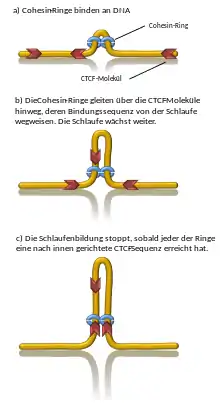
Die Schlaufenbildung beginnt wenn sich ein Cohesin-Komplex an die DNA heftet. Cohesin besteht aus zwei verbundenen, ringförmigen Untereinheiten, ähnlich wie Handschellen. Die DNA tritt durch einen Ring ein und durch den anderen wieder aus. Die Ringe gleiten in entgegengesetzter Richtung der DNA entlang. Dadurch bildet sich eine immer größer werdende DNA-Schlaufe. Erst wenn ein Ring an ein CTCF Molekül stößt, welches an die DNA gebunden ist, kann dieser Prozess gestoppt werden. Hierzu muss die Bindungssequenz für CTCF in Richtung auf das Innere der DNA-Schlaufe zeigen. Ansonsten gleitet der Cohesin-Ring darüber hinweg und die Schlaufe wächst weiter. Die Schlaufenbildung ist abgeschlossen, wenn beide Ringe des Cohesin-Komplexes eine nach innen gerichtete CTCF Sequenz erreicht haben.[7]
Neuere Modelle schlagen vor, dass TADs in einer Supercoiled-(Superspiralisierung)-DNA Struktur vorliegen und dies die Kontakte innerhalb eines TADs erhöht. Mit Hilfe von molekulardynamischen Simulationen konnte von Racko et al. gezeigt werden, dass der Cohesin-Komplex durch Superspiralisierung der DNA, die während der Transkription entsteht, in Richtung der TAD-Grenzen weitergeschoben wird. Diese Modelle erklären auch, was die treibende Kraft der Schleifenbildung sein kann und wie sichergestellt werden kann, dass Schleifen schnell und die richtige Richtung wachsen.
Darüber hinaus steht der durch Supercoiling gesteuerte Schleifenbildungsmechanismus im Einklang mit früheren Erklärungen, die vorschlagen, warum TADs, die von konvergenten CTCF-Bindungsstellen flankiert werden, stabilere Chromatinschleifen bilden als TADs, die von divergenten CTCF-Bindungsstellen flankiert werden.[8][9]
Eigenschaften
Konservierung
Es wurde berichtet, dass TADs zwischen verschiedenen Zelltypen (z. B. in Stammzellen und Blutzellen) und in Einzelfällen sogar zwischen Spezies relativ konstant sind.[10]
Beziehung zu Promoter-Enhancer-Kontakten
Die Mehrheit der beobachteten Interaktionen zwischen Promotoren und Enhancern überschreitet nicht die TAD-Grenzen. Das Entfernen einer TAD-Grenze (z. B. die Verwendung von CRISPR zum Löschen der relevanten Region des Genoms) kann es ermöglichen, neue Kontakte zwischen Promotor und Enhancer zu bilden. Dies kann die Genexpression in der Nähe beeinträchtigen – eine solche Fehlsteuerung verursacht nachweislich Gliedmaßenmissbildungen (z. B. Polydaktylie) bei Mensch und Maus.[10]
Computersimulationen haben gezeigt, dass transkriptionsbedingtes Supercoiling von Chromatinfasern erklären kann, wie TADs gebildet werden und wie sie sehr effiziente Wechselwirkungen zwischen Enhancer und ihren verwandten Promotoren im gleichen TAD gewährleisten können.[8]
Zusammenhang mit anderen strukturellen Merkmalen des Genoms
Es wurde berichtet, dass topologisch assoziierten Domänen die gleichen sind wie Replikationsdomänen. Dies sind Regionen des Genoms, die während der S-Phase der Zellteilung gleichzeitig kopiert (repliziert) werden.[11] Isolierte Nachbarschaften, DNA-Schlaufen, die aus CTCF/cohesingebundenen Regionen gebildet werden, sollen funktionell den TADs zugrunde liegen.[12]
Krankheiten
Eine Unterbrechung der TAD-Grenzen kann die Expression benachbarter Gene beeinträchtigen, was zu Krankheiten führen kann.[13]
So wurden beispielsweise genomische Strukturvarianten, die die TAD-Grenzen durchbrechen, als Ursache für Entwicklungsstörungen wie Fehlbildungen menschlicher Gliedmaßen gemeldet.[14][15] Darüber hinaus haben mehrere Studien gezeigt, dass die Unterbrechung oder Neuordnung der TAD-Grenzen Wachstumsvorteile für bestimmte Krebsarten wie die T-Zell-akute lymphatische Leukämie (T-ALL)[16], Gliome[17] und Darmkrebs[18] bieten kann.
Lamina-associated domains
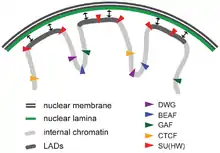
Lamina-assoziierte Domänen (LADs) sind Teile des Chromatins, die stark mit der Lamina interagieren, einer netzwerkartigen Struktur an der inneren Membran des Kerns.[19] LADs bestehen hauptsächlich aus transkriptionell stillem Chromatin, das mit trimethyliertem Lys27 auf Histon H3 angereichert ist, das eine häufige posttranslationale Histonmodifikation von Heterochromatin ist.[20] LADs haben CTCF-Bindungsstellen an ihrer Peripherie.[19]
Einzelnachweise
- Ana Pombo, Niall Dillon: Three-dimensional genome architecture: players and mechanisms. In: Nature Reviews Molecular Cell Biology. Band 16, Nr. 4, April 2015, ISSN 1471-0072, S. 245–257, doi:10.1038/nrm3965.
- Elphège P. Nora, Bryan R. Lajoie, Edda G. Schulz, Luca Giorgetti, Ikuhiro Okamoto: Spatial partitioning of the regulatory landscape of the X-inactivation centre. In: Nature. Band 485, Nr. 7398, Mai 2012, ISSN 0028-0836, S. 381–385, doi:10.1038/nature11049, PMID 22495304, PMC 3555144 (freier Volltext).
- Wouter de Laat, Denis Duboule: Topology of mammalian developmental enhancers and their regulatory landscapes. In: Nature. Band 502, Nr. 7472, Oktober 2013, ISSN 0028-0836, S. 499–506, doi:10.1038/nature12753 (nature.com [abgerufen am 16. Juli 2019]).
- Jesse R. Dixon, Siddarth Selvaraj, Feng Yue, Audrey Kim, Yan Li: Topological domains in mammalian genomes identified by analysis of chromatin interactions. In: Nature. Band 485, Nr. 7398, Mai 2012, ISSN 0028-0836, S. 376–380, doi:10.1038/nature11082, PMID 22495300, PMC 3356448 (freier Volltext).
- Jesse R. Dixon, Siddarth Selvaraj, Feng Yue, Audrey Kim, Yan Li: Topological domains in mammalian genomes identified by analysis of chromatin interactions. In: Nature. Band 485, Nr. 7398, Mai 2012, ISSN 0028-0836, S. 376–380, doi:10.1038/nature11082, PMID 22495300, PMC 3356448 (freier Volltext).
- Ana Pombo, Niall Dillon: Three-dimensional genome architecture: players and mechanisms. In: Nature Reviews Molecular Cell Biology. Band 16, Nr. 4, April 2015, ISSN 1471-0072, S. 245–257, doi:10.1038/nrm3965 (nature.com [abgerufen am 26. Juli 2019]).
- Erez Lieberman Aiden: Die Entwirrung des Genoms. In: Spektrum der Wissenschaft. Spektrum der Wissenschaft Verlagsgesellschaft, 1. Juni 2019.
- Dusan Racko, Fabrizio Benedetti, Julien Dorier, Andrzej Stasiak: Are TADs supercoiled? In: Nucleic Acids Research. Band 47, Nr. 2, 25. Januar 2019, ISSN 0305-1048, S. 521–532, doi:10.1093/nar/gky1091, PMID 30395328, PMC 6344874 (freier Volltext).
- Dusan Racko, Fabrizio Benedetti, Julien Dorier, Andrzej Stasiak: Transcription-induced supercoiling as the driving force of chromatin loop extrusion during formation of TADs in interphase chromosomes. In: Nucleic Acids Research. Band 46, Nr. 4, 28. Februar 2018, ISSN 0305-1048, S. 1648–1660, doi:10.1093/nar/gkx1123, PMID 29140466, PMC 5829651 (freier Volltext).
- Daniel Jost, Cédric Vaillant, Peter Meister: Coupling 1D modifications and 3D nuclear organization: data, models and function. In: Current Opinion in Cell Biology. Band 44, Februar 2017, ISSN 1879-0410, S. 20–27, doi:10.1016/j.ceb.2016.12.001, PMID 28040646.
- Benjamin D. Pope, Tyrone Ryba, Vishnu Dileep, Feng Yue, Weisheng Wu: Topologically associating domains are stable units of replication-timing regulation. In: Nature. Band 515, Nr. 7527, 20. November 2014, ISSN 1476-4687, S. 402–405, doi:10.1038/nature13986, PMID 25409831, PMC 4251741 (freier Volltext).
- Xiong Ji, Daniel B. Dadon, Benjamin E. Powell, Zi Peng Fan, Diego Borges-Rivera: 3D Chromosome Regulatory Landscape of Human Pluripotent Cells. In: Cell Stem Cell. Band 18, Nr. 2, 4. Februar 2016, ISSN 1875-9777, S. 262–275, doi:10.1016/j.stem.2015.11.007, PMID 26686465, PMC 4848748 (freier Volltext).
- Darío G. Lupiáñez, Malte Spielmann, Stefan Mundlos: Breaking TADs: How Alterations of Chromatin Domains Result in Disease. In: Trends in genetics: TIG. Band 32, Nr. 4, April 2016, ISSN 0168-9525, S. 225–237, doi:10.1016/j.tig.2016.01.003, PMID 26862051.
- Darío G. Lupiáñez, Katerina Kraft, Verena Heinrich, Peter Krawitz, Francesco Brancati: Disruptions of topological chromatin domains cause pathogenic rewiring of gene-enhancer interactions. In: Cell. Band 161, Nr. 5, 21. Mai 2015, ISSN 1097-4172, S. 1012–1025, doi:10.1016/j.cell.2015.04.004, PMID 25959774, PMC 4791538 (freier Volltext).
- Martin Franke, Daniel M. Ibrahim, Guillaume Andrey, Wibke Schwarzer, Verena Heinrich: Formation of new chromatin domains determines pathogenicity of genomic duplications. In: Nature. Band 538, Nr. 7624, 13. Oktober 2016, ISSN 1476-4687, S. 265–269, doi:10.1038/nature19800, PMID 27706140.
- Denes Hnisz, Abraham S. Weintraub, Daniel S. Day, Anne-Laure Valton, Rasmus O. Bak: Activation of proto-oncogenes by disruption of chromosome neighborhoods. In: Science (New York, N.Y.). Band 351, Nr. 6280, 25. März 2016, ISSN 1095-9203, S. 1454–1458, doi:10.1126/science.aad9024, PMID 26940867, PMC 4884612 (freier Volltext).
- William A. Flavahan, Yotam Drier, Brian B. Liau, Shawn M. Gillespie, Andrew S. Venteicher: Insulator dysfunction and oncogene activation in IDH mutant gliomas. In: Nature. Band 529, Nr. 7584, 7. Januar 2016, ISSN 1476-4687, S. 110–114, doi:10.1038/nature16490, PMID 26700815, PMC 4831574 (freier Volltext).
- Joachim Weischenfeldt, Taronish Dubash, Alexandros P. Drainas, Balca R. Mardin, Yuanyuan Chen: Pan-cancer analysis of somatic copy-number alterations implicates IRS4 and IGF2 in enhancer hijacking. In: Nature Genetics. Band 49, Nr. 1, Januar 2017, ISSN 1546-1718, S. 65–74, doi:10.1038/ng.3722, PMID 27869826, PMC 5791882 (freier Volltext).
- Adriana Gonzalez-Sandoval, Susan M. Gasser: On TADs and LADs: Spatial Control Over Gene Expression. In: Trends in genetics: TIG. Band 32, Nr. 8, August 2016, ISSN 0168-9525, S. 485–495, doi:10.1016/j.tig.2016.05.004, PMID 27312344.
- Mo Li, Guang-Hui Liu, Juan Carlos Izpisua Belmonte: Navigating the epigenetic landscape of pluripotent stem cells. In: Nature Reviews. Molecular Cell Biology. Band 13, Nr. 8, 23. Juli 2012, ISSN 1471-0080, S. 524–535, doi:10.1038/nrm3393, PMID 22820889.