SUMO-Proteine
Die Small Ubiquitin-Related Modifier (kurz SUMO) bilden eine Proteinfamilie, die zur Superfamilie der Ubiquitin-ähnlichen Proteinen (englisch Ubiquitin-like proteins, UBL oder Ubl) gehört und als posttranslationale Modifikation auf andere Zielproteine agiert. Bei einer posttranslationalen Modifikation, die von SUMO-Proteinen ausgeht, spricht man von einer SUMOylierung. Dabei binden sich SUMO-Proteine kovalent an Lysinreste anderer Proteine (Konjugation). Da die SUMOylierung reversibel ist, bezeichnet man die Rückreaktion als DeSUMOylierung.[1]
| Homo sapiens SMT3 suppressor of mif two 3 homolog 1 (SUMO-Protein) | ||
|---|---|---|
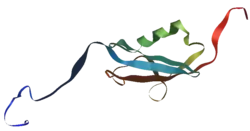 | ||
| Bändermodell | ||
|
Vorhandene Strukturdaten: 1A5R | ||
| Bezeichner | ||
| Externe IDs | ||
| Vorkommen | ||
| Übergeordnetes Taxon | Eukaryoten | |
SUMO bilden eine hochkonservierte Proteinfamilie in allen Eukaryoten und sind für das Überleben der meisten eukaryotischen Zellen notwendig. Die SUMOylierung spielt daher eine wichtige Rolle in vielen biologischen Prozessen, z. B. Transkription, DNA-Reparatur, Proteolyse, Protein-DNA-Interaktion, Protein-Protein-Interaktion, Proteinstabilität, Replikation, Kerntransport von Proteinen, nukleocytoplasmatischer Transport, Chromatinorganisation, Apoptose, Ribosomen-Biogenese, Signaltransduktion, Spleißen von prä-mRNA, Stressreaktion und der Zellzyklus-Regulation.
Eine Fehlregulation von SUMO-Proteinen kann zu Herz-Kreislauf-Erkrankungen, Diabetes mellitus, neurodegenerativen Erkrankungen und Krebs führen.
Struktur
SUMO-Proteine sind mit einer Größe von ungefähr 100 Aminosäuren (ca. 10–12 kDa) eher kleine Proteine.[2][3][4] Obwohl die Sequenzidentität zwischen SUMO und Ubiquitin nur ca. 18 % beträgt, sind sie strukturell relativ ähnlich.[5][6] Ähnlich wie Ubiquitin besitzen die SUMO-Proteine in ihrer Tertiärstruktur eine sogenannte β-grasp fold (β-GF, auf Deutsch auch Ubiquitin-ähnliche Faltung genannt), die ein gemeinsames Charakteristikum innerhalb der Familie der Ubiquitin-ähnlichen Proteinen darstellt.[6]
SUMO-Proteine unterscheiden sich von Ubiquitin darin, dass sie einen ungefähr 20 Aminosäuren langen Fortsatz am N-Terminus besitzen, der bei Ubiquitin nicht vorhanden ist. SUMO-Proteine als auch Ubiquitin werden prozessiert, wodurch ein Glycin-Glycin-Motiv am C-Terminus gebildet wird, das für die Konjugation mit Zielproteinen zuständig ist.[7][8][9][10][11][12] Unter anderem besitzen die Proteine Ubiquitin, SMT3, SUMO1 und SUMO2 die Sekundärstruktur ββααββαβ (α/β-Struktur), die sich zur Ubiquitin-ähnlichen Faltung zusammenschließt.[13] SUMO1, SUMO2 und SUMO3 bestehen zu 15 % aus α-Helices, 35–45 % aus β-Faltblättern, 25–35 % aus β-Schleifen und zu 10–15 % aus ungeordneten Resten.[14][5][15]
Klassischerweise werden SUMO-Proteine an einen Lysinrest konjugiert, der sich innerhalb der Konsensussequenz ψ-K-x-E/D befindet (wobei ψ für einen hydrophoben Rest und x für eine beliebige Aminosäure steht).[16][2] Jedoch finden die Hälfte der Konjugationen an Nicht-Konsensus- oder unvollständigen Konsensussequenzen statt.[17][18][19]
SUMO-Proteine in verschiedenen Organismen
In Säugetieren wurden bisher fünf SUMO-Paraloge entdeckt: SUMO1 (Smt3C, PIC1, UBL1, Sentrin), SUMO2 (Smt3a, Sentrin-3), SUMO3 (Smt3b, Sentrin-2), SUMO4 und SUMO5.[4][20] Das menschliche SUMO1 ist ein 101 Aminosäuren langes Polypeptid, das eine Sequenzidentität von 18 % mit dem menschlichen Ubiquitin besitzt. SUMO2 und SUMO3 von Menschen und Tieren weisen untereinander eine Sequenzidentität von 87–95 % auf, aber gegenüber SUMO1 eine Sequenzidentität von nur 50 %.[4] Da SUMO2 und SUMO3 in ihrer Aminosäuresequenz fast identisch sind, bezeichnet man sie üblicherweise zusammen als SUMO2/3.[21] Der Großteil der SUMO1-Proteine befindet sich in konjugierter Form, wohingegen SUMO2 und SUMO3 in freier, unkonjugierter Form vorliegen. Unter Stressbedingungen wird vor allem eine Konjugation der Proteine SUMO2 und SUMO3 induziert, wohingegen bei Tieren keine Induzierung einer SUMO1-Konjugation hervorgerufen wird, da bei SUMO1 das ψ-K-x-E/D-bindende Motiv fehlt.[22][2][23] Die Konsensussequenz trägt zur Kettenbildung von SUMO2 und SUMO3, aber nicht von SUMO1 bei.[23] Jedoch könnte SUMO1 zum Kettenabbruch beitragen, indem SUMO1 mehrmals an elongierten SUMO2/3-Ketten konjugiert.[24]
Das erste SUMO-Homolog Smt3 (suppressor of mif two 3) wurde ursprünglich als Suppressor des centromeren Proteins MIF2 in Saccharomyces cerevisiae entdeckt[25] und ist zur Vervollständigung der Mitose notwendig. Pmt3, das Homolog von Schizosaccharomyces pombe, ist für das Zellüberleben wichtig, jedoch führt eine Störung von Pmt3 zu Wachstumsdefekten und zu einer fehlerhaften Regulation des Genoms, unter anderem zu einer gestörten Mitose, längeren Telomeren und einer abnormalen Segregation der Chromosomen. Das Genom der Hefen, der Fruchtfliege Drosophila melanogaster und des Fadenwurms Caenorhabditis elegans codieren jeweils nur für ein einzelnes SUMO-Protein.[4] Das erste SUMO-Protein in Pflanzen, T-SUMO, wurde in der Tomate als interagierendes Protein des Enzyms ethylene-inducing xylanase (EIX) des pflanzenpathogenen Pilzes Trichoderma viride entdeckt.[26] In Arabidopsis existieren acht SUMO-Proteine (AtSUM1–AtSUM8), wobei AtSUM9 ein Pseudogen darstellt.[4]
Mechanismus
SUMO-Proteine werden durch Sentrin-spezifische Proteasen (SENPs) in ihre aktive Form überführt. SUMO-1 wird dabei am C-Terminus um vier Aminosäuren gekürzt, SUMO-2 um elf Aminosäuren und SUMO-3 um zwei Aminosäuren. Die SUMOylierung wird, wie die Ubiquitinylierung, durch drei Enzyme katalysiert, E1-Enzyme (SUMO-activating enzyme), E2-Enzyme (SUMO-conjugating enzyme) und E3-Enzyme (SUMO-Ligase).
Durch eine Thioester-Bindung an ein Cystein des heterodimeren E1-Enzyms SAE1-SAE2 wird das SUMO-Protein über ATP aktiviert und anschließend, ebenfalls über eine Thioesterbindung, auf ein Cystein des E2-Enzyms Ubc9 transferiert. Die endständige C-terminale Carboxygruppe des Glycins der SUMO-Proteine wird im dritten Teilschritt durch das E3-Enzym SUMO-Ligase an das Lysin der Erkennungssequenz der abzubauenden Proteine über eine Isopeptidbindung an das ε-Amin geknüpft. Die SUMO-Ligase (ein E3-Enzym, z. B. RanBP2) bindet an das Ubc9 (ein E2-Enzym) und an das abzubauende Protein. Die Freisetzung der SUMO-Proteine zur Wiederverwendung erfolgt durch die SENPs (DeSUMOylierung).[27]
Die E3-Enzyme lassen sich aufgrund ihrer Homologien in drei Gruppen unterteilen, den RanBP2-Typ, den HECT-Typ und den RING-Typ (z. B. bei Hefen Siz1, Siz2, Mms21, Cst9 und Zip3 und bei Menschen PIAS1, PIAS3, PIASxα, PIASxβ und PIASy). Das E1-Enzym in Saccharomyces cerevisiae heißt Aos1-Uba2. Es gibt bei Hefen als SUMO-Protease Ulp1 an Kernporen und Ulp2 im Zellkern.
Experimentell kann man eine SUMOylierung von DNA-bindenden Proteinen durch eine anti-SUMO-ChIP feststellen.
Regulierung biologischer Prozesse
Die SUMOylierung spielt in verschiedenen zellulären Prozessen eine Rolle, z. B. beim Kerntransport,[28] bei der Apoptose, bei der Regulation der Transkription durch Induktion eines Abbaus von Histonen, bei der Proteindegradation (Autophagie), als antiviraler Mechanismus, bei Stressreaktionen, bei der Chromosomentrennung in der Mitose und bei der Einleitung der nächsten Phase im Zellzyklus.[27][29][30] SUMO-Proteine dienen hierbei als Regulatorprotein im Organismus.[31]
DNA-Reparatur
Die DNA ist permanent Angriffen durch mutagene Substanzen ausgesetzt. Ein komplexer Apparat steuert die Erkennung von DNA-Schäden, deren Reparatur oder die Auslösung der Apoptose. SUMO hat gewissermaßen die Funktion eines molekularen Klebers, um die an der Reparatur beteiligten großen Proteinkomplexe zusammenzuhalten. SUMO ist an folgenden Reparaturmechanismen beteiligt:
Transkription
In zahlreichen Fällen wird durch SUMO die Transkriptionen bestimmter Gene gehemmt.[33] Die E3-Ligase PIAS4 ist erforderlich für die SUMOylierung des Transkriptionsfaktors YY1 (englisch Ying Yang 1) der Hefe. Sie ist unabhängig von einer RING-Fingerdomäne (eine Zinkfingerdomäne). RING-Fingerdomänen stellen ansonsten die funktionelle Domäne der E3-Ligasen dar.
Mitose
Während der Mitose wandert SUMO-2/3 zu den Zentromeren und den kondensierten Chromosomen. SUMO-1 dagegen wandert zur mitotischen Spindel und zur mittleren Spindelzone. Dies ist ein Hinweis dafür, dass SUMO an der Regulierung des mitotischen Prozesses von Säugerzellen beteiligt ist.[34] Bemerkenswert ist, dass die Topoisomerase II während der Mitose von SUMO-2/3 SUMOyliert wird.[35]
Kerntransport
Das erste entdeckte Substrat der SUMOylierung war RanGAP1. Die Modifikation führte zum Transport von RanGAP1 zum Kernporenkomplex.[36][28] Die SUMOylierung von hNinein (englisch human Ninein) führt zum Transport vom Zentrosom zum Zellkern.[37]
Sonstige Funktionen
SUMO-2/3 ist an der Reaktion auf Stress beteiligt. SUMO-4 unterscheidet sich von SUMO-2 und SUMO-3 durch ein Prolin anstelle von Glutamin in Position 90. Als Folge dieser Modifikation ist SUMO-4 nicht unter normalen Bedingungen aktiv. Erst unter Stresssituationen wie Hunger wird es aktiviert.[38]
Krankheitsprozesse mit Beteiligung von SUMO
Bei Proteinfehlfaltungserkrankungen wie Chorea Huntington, der Alzheimer-Krankheit, der Parkinson-Krankheit und der spinozerebellären Ataxie 1 werden die Einschlusskörperchen zwar SUMOyliert, aber nicht abgebaut.
Bei der Mukoviszidose, einer häufigen Erbkrankheit, wird ein mutiertes CFTR-Protein (Cystic Fibrosis Transmembrane Conductance Regulator) produziert, welches nicht zur Zellmembran transportiert wird und daher seine wichtige Transporterfunktion nicht erfüllen kann. Die SUMOylierung des CRTF-Proteins ist erforderlich, um den Transport vom ER zur Zellmembran zu ermöglichen. Bei einer Mutation von CFTR im SUMO-Motiv kann CFTR nicht an SUMO binden und wird im Golgi-Apparat zurückgehalten.[31]
In verschiedenen Krankheiten ist eine fehlerhafte SUMOylierung beobachtet worden, z. B. bei Krebs, bei der amyotrophen Lateralsklerose sowie bei Infektionen mit Adenoviren und Herpesviren.[39] Diese Viren verwenden die SUMOylierung zum Transport viraler Proteine.[40]
Verwendung von SUMO-Proteinen
Spezielle Proteine können dadurch gewonnen werden, dass man die Synthese zum Beispiel in Kolibakterien durchführt. Die erhaltenen Proteine sind jedoch häufig schlecht gefaltet und bilden Aggregate, sogenannte Inclusion Bodies.[41] Für die weitere Prozessierung müssen die Proteine in eine lösliche Form überführt werden. Dies kann durch eine Verbindung mit SUMO erreicht werden. Später kann SUMO von dem Ziel-Protein durch eine SUMO-spezifische Protease wie Ulp1-Peptidase gelöst werden.[42]
Siehe auch
Einzelnachweise
- J. R. Gareau, C. D. Lima: The SUMO pathway: emerging mechanisms that shape specificity, conjugation and recognition. In: Nat Rev Mol Cell Biol. (2010), Band 11, Ausgabe 12, S. 861–871. doi:10.1038/nrm3011. PMID 21102611; PMC 3079294 (freier Volltext).
- E. S. Johnson: Protein modification by SUMO. In: Annual review of biochemistry. Band 73, 2004, S. 355–382, doi:10.1146/annurev.biochem.73.011303.074118, PMID 15189146 (Review).
- R. Sabate, A. Espargaro, R. Graña-Montes, D. Reverter, S. Ventura: Native structure protects SUMO proteins from aggregation into amyloid fibrils. In: Biomacromolecules. Band 13, Nummer 6, Juni 2012, S. 1916, doi:10.1021/bm3004385, PMID 22559198.
- H. J. Park, W. Y. Kim, H. C. Park, S. Y. Lee, H. J. Bohnert, D. J. Yun: SUMO and SUMOylation in plants. In: Molecules and cells. Band 32, Nummer 4, Oktober 2011, S. 305–316, doi:10.1007/s10059-011-0122-7, PMID 21912873, PMC 3887640 (freier Volltext) (Review).
- S. Vijay-Kumar, C. E. Bugg, W. J. Cook: Structure of ubiquitin refined at 1.8 A resolution. In: Journal of molecular biology. Band 194, Nummer 3, 5. April 1987, S. 531–544, doi:10.1016/0022-2836(87)90679-6, PMID 3041007.
- P. Bayer, A. Arndt, S. Metzger, R. Mahajan, F. Melchior, R. Jaenicke, J. Becker: Structure determination of the small ubiquitin-related modifier SUMO-1. In: Journal of molecular biology. Band 280, Nummer 2, 10. Juli 1998, S. 275–286, doi:10.1006/jmbi.1998.1839, PMID 9654451.
- E. Ozkaynak, D. Finley, M. J. Solomon, A. Varshavsky: The yeast ubiquitin genes: a family of natural gene fusions. In: The EMBO Journal. Band 6, Nummer 5, Mai 1987, S. 1429–1439, PMID 3038523, PMC 553949 (freier Volltext).
- K. D. Wilkinson: Regulation of ubiquitin-dependent processes by deubiquitinating enzymes. In: FASEB Journal. Band 11, Nummer 14, Dezember 1997, S. 1245–1256, doi:10.1096/fasebj.11.14.9409543, PMID 9409543 (Review).
- C. N. Larsen, B. A. Krantz, K. D. Wilkinson: Substrate specificity of deubiquitinating enzymes: ubiquitin C-terminal hydrolases. In: Biochemistry. Band 37, Nummer 10, 10. März 1998, S. 3358–3368, doi:10.1021/bi972274d, PMID 9521656.
- S. J. Li, M. Hochstrasser: A new protease required for cell-cycle progression in yeast. In: Nature. Band 398, Nummer 6724, 18. März 1999, S. 246–251, doi:10.1038/18457, PMID 10094048.
- S. Fang, A. M. Weissman: A field guide to ubiquitylation. In: Cellular and molecular life sciences. Band 61, Nummer 13, Juli 2004, S. 1546–1561, doi:10.1007/s00018-004-4129-5, PMID 15224180 (Review).
- W. Li, Y. Ye: Polyubiquitin chains: functions, structures, and mechanisms. In: Cellular and molecular life sciences. Band 65, Nummer 15, August 2008, S. 2397–2406, doi:10.1007/s00018-008-8090-6, PMID 18438605, PMC 2700825 (freier Volltext) (Review).
- A. Alonso, M. Greenlee, J. Matts, J. Kline, K. J. Davis, R. K. Miller: Emerging roles of sumoylation in the regulation of actin, microtubules, intermediate filaments, and septins. In: Cytoskeleton. Band 72, Nummer 7, Juli 2015, S. 305–339, doi:10.1002/cm.21226, PMID 26033929, PMC 5049490 (freier Volltext) (Review).
- J. Song, Z. Zhang, W. Hu, Y. Chen: Small ubiquitin-like modifier (SUMO) recognition of a SUMO binding motif: a reversal of the bound orientation. In: Journal of Biological Chemistry. Band 280, Nummer 48, Dezember 2005, S. 40122–40129, doi:10.1074/jbc.M507059200, PMID 16204249.
- W. C. Huang, T. P. Ko, S. S. Li, A. H. Wang: Crystal structures of the human SUMO-2 protein at 1.6 A and 1.2 A resolution: implication on the functional differences of SUMO proteins. In: European Journal of Biochemistry. Band 271, Nummer 20, Oktober 2004, S. 4114–4122, doi:10.1111/j.1432-1033.2004.04349.x, PMID 15479240.
- F. Melchior: SUMO–nonclassical ubiquitin. In: Annual review of cell and developmental biology. Band 16, 2000, S. 591–626, doi:10.1146/annurev.cellbio.16.1.591, PMID 11031248 (Review).
- H. A. Blomster, V. Hietakangas, J. Wu, P. Kouvonen, S. Hautaniemi, L. Sistonen: Novel proteomics strategy brings insight into the prevalence of SUMO-2 target sites. In: Molecular & cellular proteomics : MCP. Band 8, Nummer 6, Juni 2009, S. 1382–1390, doi:10.1074/mcp.M800551-MCP200, PMID 19240082, PMC 2690485 (freier Volltext).
- I. Matic, J. Schimmel, I. A. Hendriks, M. A. van Santen, F. van de Rijke, H. van Dam, F. Gnad, M. Mann, A. C. Vertegaal: Site-specific identification of SUMO-2 targets in cells reveals an inverted SUMOylation motif and a hydrophobic cluster SUMOylation motif. In: Molecular cell. Band 39, Nummer 4, 27. August 2010, S. 641–652, doi:10.1016/j.molcel.2010.07.026, PMID 20797634.
- S. Teng, H. Luo, L. Wang: Predicting protein sumoylation sites from sequence features. In: Amino Acids. Band 43, Nummer 1, Juli 2012, S. 447–455, doi:10.1007/s00726-011-1100-2, PMID 21986959.
- A. Pichler, C. Fatouros, H. Lee, N. Eisenhardt: SUMO conjugation - a mechanistic view. In: Biomolecular concepts. Band 8, Nummer 1, März 2017, S. 13–36, doi:10.1515/bmc-2016-0030, PMID 28284030 (Review).
- L. Wang, C. Wansleeben, S. Zhao, P. Miao, W. Paschen, W. Yang: SUMO2 is essential while SUMO3 is dispensable for mouse embryonic development. In: EMBO reports. Band 15, Nummer 8, August 2014, S. 878–885, doi:10.15252/embr.201438534, PMID 24891386, PMC 4197045 (freier Volltext).
- K. M. Bohren, V. Nadkarni, J. H. Song, K. H. Gabbay, D. Owerbach: A M55V polymorphism in a novel SUMO gene (SUMO-4) differentially activates heat shock transcription factors and is associated with susceptibility to type I diabetes mellitus. In: Journal of Biological Chemistry. Band 279, Nummer 26, 25. Juni 2004, S. 27233–27238, doi:10.1074/jbc.M402273200, PMID 15123604.
- H. Saitoh, J. Hinchey: Functional heterogeneity of small ubiquitin-related protein modifiers SUMO-1 versus SUMO-2/3. In: The Journal of Biological Chemistry. Band 275, Nr. 9, 3. März 2000, ISSN 0021-9258, S. 6252–6258, doi:10.1074/jbc.275.9.6252, PMID 10692421.
- I. Matic, M. van Hagen, J. Schimmel, B. Macek, S. C. Ogg, M. H. Tatham, R. T. Hay, A. I. Lamond, M. Mann, A. C. Vertegaal: In vivo identification of human small ubiquitin-like modifier polymerization sites by high accuracy mass spectrometry and an in vitro to in vivo strategy. In: Molecular & cellular proteomics : MCP. Band 7, Nummer 1, Januar 2008, S. 132–144, doi:10.1074/mcp.M700173-MCP200, PMID 17938407, PMC 3840926 (freier Volltext).
- P. B. Meluh, D. Koshland: Evidence that the MIF2 gene of Saccharomyces cerevisiae encodes a centromere protein with homology to the mammalian centromere protein CENP-C. In: Molecular Biology of the Cell. Band 6, Nummer 7, Juli 1995, S. 793–807, doi:10.1091/mbc.6.7.793, PMID 7579695, PMC 301241 (freier Volltext).
- U. Hanania, N. Furman-Matarasso, M. Ron, A. Avni: Isolation of a novel SUMO protein from tomato that suppresses EIX-induced cell death. In: The Plant journal : for cell and molecular biology. Band 19, Nummer 5, September 1999, S. 533–541, doi:10.1046/j.1365-313x.1999.00547.x, PMID 10504575.
- K. D. Sarge, O. K. Park-Sarge: Sumoylation and human disease pathogenesis. In: Trends Biochem Sci. (2009), Band 34, Ausgabe 4, S. 200–205. doi:10.1016/j.tibs.2009.01.004. PMID 19282183; PMC 2974900 (freier Volltext).
- R. Mahajan, C. Delphin, T. Guan, L. Gerace, F. Melchior: A small ubiquitin-related polypeptide involved in targeting RanGAP1 to nuclear pore complex protein RanBP2. In: Cell (1997), Band 88, Ausgabe 1, S. 97–107. PMID 9019411.
- S. P. Jackson, D. Durocher: Regulation of DNA damage responses by ubiquitin and SUMO. In: Mol Cell. (2013), Band 49, Ausgabe 5, S. 795–807. doi:10.1016/j.molcel.2013.01.017. PMID 23416108.
- J. Wan, D. Subramonian, X. D. Zhang: SUMOylation in control of accurate chromosome segregation during mitosis. In: Curr Protein Pept Sci. (2012), Band 13, Ausgabe 5, S. 467–481. PMID 22812528; PMC 3474960 (freier Volltext).
- Schulze, Christine: SUMOylierung: ein neuer Regulations-mechanismus des Transportes von CFTR zur Membran. Dissertation, LMU München: Fakultät für Biologie, 2009, urn:nbn:de:bvb:19-107929.
- Deena Jalal, Jisha Chalissery, Ahmed H. Hassan: Genome maintenance in Saccharomyces cerevisiae: the role of SUMO and SUMO-targeted ubiquitin ligases. In: Nucleic Acids Research. Band 45, Nr. 5, 17. März 2017, ISSN 1362-4962, S. 2242–2261, doi:10.1093/nar/gkw1369, PMID 28115630, PMC 5389695 (freier Volltext).
- Grace Gill: Something about SUMO inhibits transcription. In: Current Opinion in Genetics & Development. Band 15, Nr. 5, Oktober 2005, ISSN 0959-437X, S. 536–541, doi:10.1016/j.gde.2005.07.004, PMID 16095902.
- Xiang-Dong Zhang, Jacqueline Goeres, Hong Zhang, Tim J. Yen, Andrew C. G. Porter: SUMO-2/3 modification and binding regulate the association of CENP-E with kinetochores and progression through mitosis. In: Molecular Cell. Band 29, Nr. 6, 28. März 2008, ISSN 1097-4164, S. 729–741, doi:10.1016/j.molcel.2008.01.013, PMID 18374647, PMC 2366111 (freier Volltext).
- Yoshiaki Azuma, Alexei Arnaoutov, Mary Dasso: SUMO-2/3 regulates topoisomerase II in mitosis. In: The Journal of Cell Biology. Band 163, Nr. 3, 10. November 2003, ISSN 0021-9525, S. 477–487, doi:10.1083/jcb.200304088, PMID 14597774, PMC 2173648 (freier Volltext).
- A novel ubiquitin-like modification modulates the partitioning of the Ran-GTPase-activating protein RanGAP1 between the cytosol and the nuclear pore complex. In: The Journal of Cell Biology. Band 135, Nr. 6, 2. Dezember 1996, ISSN 0021-9525, S. 1457–1470, doi:10.1083/jcb.135.6.1457, PMID 8978815, PMC 2133973 (freier Volltext).
- Tai-Shan Cheng, Li-Kwan Chang, Shen-Long Howng, Pei-Jung Lu, Chu-I. Lee: SUMO-1 modification of centrosomal protein hNinein promotes hNinein nuclear localization. In: Life Sciences. Band 78, Nr. 10, 2. Februar 2006, ISSN 0024-3205, S. 1114–1120, doi:10.1016/j.lfs.2005.06.021, PMID 16154161.
- Wenzhong Wei, Ping Yang, Junfeng Pang, Shu Zhang, Ying Wang: A stress-dependent SUMO4 sumoylation of its substrate proteins. In: Biochemical and Biophysical Research Communications. Band 375, Nr. 3, 24. Oktober 2008, ISSN 1090-2104, S. 454–459, doi:10.1016/j.bbrc.2008.08.028, PMID 18708028.
- P. Wimmer, S. Schreiner, T. Dobner: Human pathogens and the host cell SUMOylation system. In: J Virol. (2012), Band 86, Ausgabe 2, S. 642–54. doi:10.1128/JVI.06227-11. PMID 22072786; PMC 3255802 (freier Volltext).
- Y. E. Wang, O. Pernet, B. Lee: Regulation of the nucleocytoplasmic trafficking of viral and cellular proteins by ubiquitin and small ubiquitin-related modifiers. In: Biol Cell (2012), Band 104, Ausgabe 3, S. 121–138. doi:10.1111/boc.201100105. PMID 22188262; PMC 3625690 (freier Volltext).
- Richard R. Burgess: Refolding solubilized inclusion body proteins. In: Methods in Enzymology. Band 463, 2009, ISSN 1557-7988, S. 259–282, doi:10.1016/S0076-6879(09)63017-2, PMID 19892177.
- Richard J. Giannone, Andrew B. Dykstra: Protein Affinity Tags: Methods and Protocols. Springer, New York, NY 2014, ISBN 978-1-4939-1034-2, S. 71–80.