Nukleosom
Nukleosomen bilden einen Komplex aus DNA und Histonen. Dies ist die erste Verpackungsstufe der DNA im Zellkern eukaryotischer Zellen; Vorstufen finden sich auch bei Archaeen. Die Abfolge der Nukleosomenpakete, welche die DNA im Chromatin als 30 nm dicke Faser zusammenhalten, wird Solenoidstruktur genannt.
| Übergeordnet |
| Chromosom |
| Untergeordnet |
| DNA Histone |
| Gene Ontology |
|---|
| QuickGO |
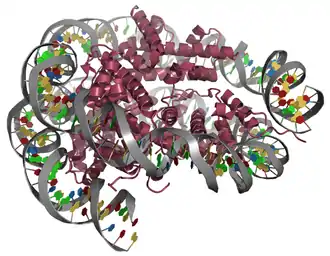
Aufbau und Struktur
Als Nukleosom bezeichnet man die Einheit von DNA und einem Histonoktamer. Das Oktamer besteht aus je zwei Exemplaren der Proteine H2A, H2B, H3 und H4. Um so einen Proteinkomplex sind 146 oder 147 Basenpaare der DNA als linksgängige Superhelix gewunden. Durch die Windung der DNA um den Histon-Komplex verkürzt sich die Länge der DNA auf ein siebtel von 68 nm auf rund 10 nm.
Durch Verdau des Chromatins mit MNase (Endonuklease, die freie DNA verdaut) erhält man Histonoktamere, um die ein ca. 147 Basenpaare langes DNA Stück gewunden ist sowie andere DNA bindende Proteine und deren gebundene DNA-Fragmente[1]. Diese Einheit wird als Nukleosomen Core Partikel (NCP) oder Nukleosomen-Grundpartikel bezeichnet.
Im Chromatin sind die einzelnen Nukleosomen Core Partikel durch unterschiedlich lange DNA Linker miteinander verbunden. Zusammen mit dem Linker Histon H1 bezeichnet man das Nukleosomen Core Partikel inklusive Linker DNA als Chromatosom. Das Linker Histon H1 erwirkt eine Kondensation der einzelnen Nukleosome und resultiert in einer kompakteren Organisation des Chromatins, welche zumindest in vivo als 30-nm Faser identifiziert werden konnte.
Die Struktur der Nukleosome wurde in den 1980ern in Pionierarbeit von Aaron Klugs Arbeitsgruppe aufgeklärt. Mittels Röntgenstrukturanalyse konnte gezeigt werden, dass im Core Partikel ein Tetramer von (H3)2(H4)2, sowie zwei Dimere der Histone H2A-H2B vorliegen.[2]
Entdeckung
Von Ada und Donald Olins in elektronenmikroskopischen Darstellungen gequollener Zellkerne entdeckt und 1973 erstmals auf dem "Third Annual Meeting of the American Society for Cell Biology" als "ν-body" (‚neues Partikel‘) vorgestellt, wurde die helikale Form (Solenoidstruktur) nahezu umgehend als elementare Verpackungseinheit der DNA im Chromatin akzeptiert[3].
1974 gelangen mehreren Teams, darunter jenem von Roger Kornberg, Analysen, die den Aufbau dieser Partikel aus einem aus acht Histonen aufgebauten Komplex, einem verbindenden Linker-Histon und etwa 160–200 Basenpaaren an DNA zeigten. 1975 wurde diese Einheit als Nukleosom eingeführt. 1974 gilt heute als Geburtsjahr der molekularen Epigenetik.
Neben Wechselwirkungen, die zur Verdichtung der DNA führen, gehen die Histone Interaktionen untereinander ein. So wird der Nukleosomenkern (das "core particle") aus jeweils zwei Exemplaren der Histone H2a, H2b, H3 und H4 gebildet. Um diesen Proteinkomplex sind in 1,65 Windungen 146 Basenpaare von DNA gewickelt.[4] Der Bereich zwischen zwei Nukleosomen (der variable "linker"-Bereich, der zwischen 160 Basenpaaren in Hefe und 200 Basenpaaren in höheren Organismen umfassen kann; beim Menschen sind es 50–60 Basenpaare) wird durch ein weiteres Histon, H1, besetzt, welches am Aufbau nächsthöherer Strukturen (der sog. 30-nm-Faser, erklärt z. B. im Solenoid-Modell) beteiligt ist. Die Komponenten des Nukleosomenkerns wurden in der Evolution hoch konserviert (nur zwei Aminosäurereste unterscheiden das Histon H3 des Menschen von jenem der Erbse), was die fundamentale Bedeutung dieser Einheit (und ihrer Modifikationen – siehe unten) unterstreicht.
Arbeiten zur Struktur der Nukleosomen wurden durch Aaron Klug (Nobelpreis 1982[5] für Kristall-Strukturanalysen an Protein/Nucleinsäure-Komplexen) in London am Medical Research Council aufgenommen. Diese Arbeiten führten 1984 bei noch relativ geringer Auflösung zum ersten Strukturvorschlag.[6][7] Die Arbeiten werden seitdem systematisch durch Timothy Richmond[8], der bereits in der Gruppe von Klug 1984 an dem ersten Strukturvorschlag beteiligt war, am Institute for Molecular Biology & Biophysics der ETH Zürich vorangetrieben. 1997 publizierte die Arbeitsgruppe von Richmond eine Struktur des Nukleosoms mit einer Auflösung von 2,8 Å[4] und 2002 folgte die Publikation der Struktur mit einer Auflösung von 1,9 Å.[9][10]
Im Jahr 2005 publizierte die Arbeitsgruppe von Richmond eine Röntgen-Kristallstruktur des Tetranukleosoms.[11]
Einzelnachweise
- Kristin Brogaard, Liqun Xi, Ji-Ping Wang & Jonathan Widom: A map of nucleosome positions in yeast at base-pair resolution. Nature, 2012, abgerufen am 24. Juni 2018 (englisch).
- Jeremy M. Berg, John L. Tymoczko, Lubert StryerStryer Biochemie In: Springer Spektrun. 7. Auflage, S. 951–952
- A. L. Olins, M. B. Senior, D. E. Olins: Ultrastructural features of chromatin nu bodies. In: The Journal of Cell Biology. Band 68, Nr. 3, März 1976, ISSN 0021-9525, S. 787–793, PMID 1035912, PMC 2109642 (freier Volltext) – (rupress.org [abgerufen am 17. November 2017]).
- Karolin Luger, Armin W. Mäder, Robin K. Richmond, David F. Sargent, Timothy J. Richmond: Crystal structure of the nucleosome core particle at 2.8 Å resolution. In: Nature. 389, Nr. 6648, 1997, S. 251–260, doi:10.1038/38444.
- nobel.se: Aaron Klug - Autobiography
- T. J. Richmond, J. T. Finch,B. Rushton, D. Rhodes, A. Klug: Structure of the nucleosome core particle at 7 Å resolution. In: Nature. 311, 1984, S. 532–537, PMID 6482966.
- M. M. Struck, A. Klug, T. J. Richmond: Comparison of X-ray structures of the nucleosome core particle in two different hydration states. In: J. Mol. Biol. 224, 1992, S. 253–264, PMID 1548703.
- Gruppe T. J. Richmond (Memento des Originals vom 3. Juni 2004 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis.
- C. A. Davey CA, Sargent, K. Luger, A. W. Maeder, T. J. Richmond: Solvent mediated interactions in the structure of the nucleosome core particle at 1.9 Å resolution. In: J. Mol. Biol. 319, Nr. 5, 2002, S. 1097–1113, PMID 12079350.
- T. J. Richmond, C. A. Davey: The structure of DNA in the nucleosome core. In: Nature. 423, 2003, S. 145–150, PMID 12736678.
- T. Schalch, S. Duda, D. F. Sargent, T. J. Richmond: X-ray structure of a tetranucleosome and its implications for the chromatin fibre. In: Nature. 436, Nr. 7047, 2005, S. 138–141, PMID 16001076.
Literatur
- C. P. Prior, C. R. Cantor, E. M. Johnson, V. C. Littau, V. G. Allfrey: Reversible changes in nucleosome structure and histone H3 accessibility in transcriptionally active and inactive states of rDNA chromatin. In: Cell. 34, 1983, S. 1033–1042.
- J. Gòmez-Lira M. M. Bode, H. Schröter: Nucleosomal particles open as the histone core becomes hyperacetylated. In: Eur. J. Biochem. 130, 1983, S. 437–445.
- B. D. Strahl, C. D. Allis: The language of covalent histone modifications. In: Nature. 403, Nr. 6765, 2000, S. 41–45.
- V. B. Teif, K. Rippe: Predicting nucleosome positions on the DNA: combining intrinsic affinities and remodeler activities. In Nucleic Acids Res. 37, 2009, S. 5641–5655, doi:10.1093/nar/gkp610.