L-Ribonukleinsäureaptamer
L-Ribonukleinsäureaptamere (kurz L-RNA-Aptamere) sind der Ribonukleinsäure (RNA) ähnliche Moleküle, die aus unnatürlichen L-Ribonukleotiden aufgebaut sind. Sie sind künstliche Oligonukleotide und stereochemische Spiegelbilder natürlicher Oligonukleotide. L-Ribonukleinsäureaptamere stellen eine spezielle Form der Aptamere dar und können wie diese spezifische Moleküle, beispielsweise Proteine, über ihre dreidimensionale Struktur binden. Dank der Verwendung von L-Nukleotiden zeichnen sie sich durch eine hohe enzymatische Stabilität aus. L-Ribonukleinsäureaptamere werden derzeit unter dem Markennamen Spiegelmer (von griechisch meros ‚Gebiet‘) als potenzielle Arzneistoffe entwickelt und in klinischen Studien geprüft.
Eigenschaften
Chemische Eigenschaften
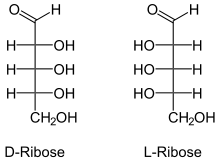
L-Ribonukleinsäureaptamere sind als die stereochemischen Spiegelbilder (Enantiomere) natürlicher Oligonukleotide aus L-Nukleotiden aufgebaut. Nukleotide, aus denen Amptamere einschließlich L-Ribonukleinsäureaptamere aufgebaut sind, also Adenosinmonophosphat (AMP), Guanosinmonophosphat (GMP), Cytidinmonophosphat (CMT) und Uridinmonophosphat (UMP), bestehen aus einem Phosphatrest, einer Nukleobase und einer Ribosegruppe als Träger der Chiralitätszentren. Durch Austausch der natürlichen D-Ribose gegen sein Enantiomer, die künstliche L-Ribose, entstehen L-Nukleotide, die Bausteine der L-Ribonukleinsäureaptamere.[1]
Biologische Eigenschaften
Wie andere Aptamere sind L-Ribonukleinsäureaptamere in der Lage, Moleküle, wie Peptide, Proteine und niedermolekulare Stoffe, zu binden. Die Affinität von L-Ribonukleinsäureaptameren zu ihren Zielmolekülen liegt oft im pico- bis nanomolaren Bereich und ist somit der von Antikörpern vergleichbar. L-Ribonukleinsäureaptamere selbst besitzen eine geringe Antigenität.[2] Im Gegensatz zu anderen Aptameren, die hydrolytisch durch Enzyme gespalten werden können, besitzen L-Ribonukleinsäureaptamere eine hohe Stabilität im Blutserum.[3] Ungeachtet dessen werden sie auf Grund ihrer geringen molaren Masse, die unterhalb der Nierenschwelle liegt, innerhalb kurzer Zeit über die Nieren ausgeschieden. Modifizierte L-Ribonukleinsäureaptamere mit einer höheren molaren Masse, beispielsweise pegylierte Spiegelmere, zeigen eine verlängerte Plasmahalbwertzeit.[2]
Herstellung
Im Gegensatz zu anderen Aptameren können L-Ribonukleinsäureaptamere nicht mit Hilfe der konventionellen, als systematische Evolution von Liganden durch exponentielle Anreicherung (SELEX) bezeichneten Technik gewonnen werden, da L-Nukleinsäuren nicht mit Hilfe enzymatischer Verfahren, wie der der Polymerasekettenreaktion, amplifiziert werden können. Daher erfolgt die Selektion über den Umweg konventioneller D-Nukleinsäuren unter Verwendung von gespiegelten Zielmolekülen.
1. Spiegelung des Zielmoleküls
Im ersten Schritt wird künstlich ein Spiegelbild des Zielmoleküls erzeugt. Im Fall von Peptiden und kleineren Proteinen werden diese synthetisch unter Verwendung künstlicher D-Aminosäuren, den Enantiomeren der natürlichen Aminosäuren, mit Hilfe der Peptidsynthese hergestellt. Ist das Zielmolekül ein größeres Protein, so kann gegebenenfalls das Spiegelbild eines Epitops mit Hilfe der Peptidsynthese unter Verwendung von D-Aminosäuren hergestellt werden.
2. SELEX
Eine konventionelle aus bis zu 1016 verschiedenen Oligonukleotiden bestehende Molekülbibliothek dient als Ausgangspunkt für das anschließende SELEX-Verfahren. In Zyklen aus Selektion unter Verwendung des Spiegelbilds des Zielmoleküls, Separation, Amplifikation und gegebenenfalls Mutation werden die Oligonukleotide isoliert, welche das Spiegelbild des Zielmoleküls am besten binden.
3. Sequenzierung und Synthese
Mit Hilfe der DNA-Sequenzierung wird die Nukleinsäuresequenz der aus dem SELEX-Verfahren gewonnenen Oligonukleotide ermittelt. Diese Information wird zur künstlichen Synthese des Spiegelbild des Oligonukleotids, des Spiegelmers, unter Verwendung von L-Nukleotiden verwendet.
Verwendung
L-Ribonukleinsäureaptamere, die beispielsweise gegen die Chemokine CCL2 und CXCL12, die Komplementkomponente C5a und Ghrelin gerichtet sind, stellen potenzielle Arzneistoffe dar. Sie befinden sich derzeit in der präklinischen oder klinischen Entwicklung. Bis heute haben drei L-Ribonukleinsäureaptamere klinische Phase IIa Studien durchlaufen. Dazu zählen das Anti-CCL2-L-Ribonukleinsäureaptamer Emapticap, das gegen CXCL12 gerichtete Olaptesed und das gegen das Eisen-Stoffwechselregulatorprotein Hepcidin gerichtete Lexaptepid.[1] Auch eine Anwendung als Diagnostika ist möglich.[3]
Einzelnachweise
- Vater A, Klussmann S: Turning mirror-image oligonucleotides into drugs: the evolution of Spiegelmer therapeutics. In: Drug Discovery Today. 20, Nr. 1, Januar 2015, S. 147–155. doi:10.1016/j.drudis.2014.09.004. PMID 25236655.
- Wlotzka B, Leva S, Eschgfäller B, et al.: In vivo properties of an anti-GnRH Spiegelmer: an example of an oligonucleotide-based therapeutic substance class. In: Proc. Natl. Acad. Sci. U.S.A.. 99, Nr. 13, Juni 2002, S. 8898–902. doi:10.1073/pnas.132067399. PMID 12070349. PMC 124395 (freier Volltext).
- Klussmann S, Nolte A, Bald R, Erdmann VA, Fürste JP: Mirror-image RNA that binds D-adenosine. In: Nat. Biotechnol.. 14, Nr. 9, September 1996, S. 1112–5. doi:10.1038/nbt0996-1112. PMID 9631061.
Literatur
- Vater A, Klussmann S: Toward third-generation aptamers: Spiegelmers and their therapeutic prospects. In: Curr Opin Drug Discov Devel. 6, Nr. 2, März 2003, S. 253–61. PMID 12669461.